文章信息
- 王丽娜, 余正, 田野, 张文静, 杨军
- WANG Lina, YU Zheng, TIAN Ye, ZHANG Wenjing, YANG Jun
- 基于克隆文库技术分析瘤棘砂壳虫的食物组成
- Diet composition of Difflugia tuberspinifera (testate amoeba) based on a clone library technique
- 生态学报, 2015, 35(18): 6183-6188
- Acta Ecologica Sinica, 2015, 35(18): 6183-6188
- http://dx.doi.org/10.5846/stxb201401260195
-
文章历史
- 收稿日期: 2014-01-26
- 网络出版日期: 2014-11-19
2. 厦门大学海洋与地球学院, 厦门 361102;
3. 中国地质大学(武汉)环境学院, 武汉 430074;
4. 中国科学院大学, 北京 100049
2. College of Ocean and Earth Sciences, Xiamen University, Xiamen 361102, China;
3. School of Environmental Studies, China University of Geosciences (Wuhan), Wuhan 430074, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
砂壳虫(Difflugia)广泛分布于世界各地,是淡水生境中丰度和多样性最高的有壳虫原生动物,特别是在湖泊水库物质循环和能量流动等关键生态过程中发挥重要的作用。然而,长期以来对有壳虫食性了解极为有限,并且以往的研究主要限于显微镜观察,认为其主要以藻类和真菌为食物[1]。瘤棘砂壳虫(Difflugia tuberspinifera)是东亚特有的淡水自由生原生动物,首次在中国贵州乌江渡被发现[2],虫体大小100—120 μm,广泛分布于我国长江与珠江中下游以及福建地区的湖泊和水库中[3, 4, 5, 6]。近年来,韩博平等[6, 7]首次报道瘤棘砂壳虫以摄食水中的微小颗粒及轮虫为主,说明单细胞生物可以捕食多细胞的后生浮游生物。事实上,原生动物是经典食物链与微食物网的关键连接点,因此,在维持水生态系统结构、功能等方面具有重要的地位和意义[8]。
目前国际学者对砂壳虫的研究重点聚焦于形态学、分类学、生态学、系统发育等方面。杨军等[3, 9]综合运用现代分类学技术方法对瘤棘砂壳虫进行了系统分类描述,利用X射线能谱仪分析淡水瘤棘砂壳虫壳体元素,发现其壳体化学元素组成介于海洋和土壤有壳虫之间;Wanner等[10]通过形态计量学方法,揭示了有壳虫适应不同环境的各种机制;Gomaa等[11]基于SSU rRNA基因序列在分子水平上进一步确定砂壳虫系统发育地位。韩博平等[6]基于显微观察首次发现瘤棘砂壳虫能够捕食轮虫,这些研究结果都为砂壳虫捕食特性的研究奠定了一定的基础。近几年,在福建多个水库中发现大量瘤棘砂壳虫,为了更清楚地了解瘤棘砂壳虫的捕食习性和食物组成,首次尝试将分子生物学技术引入到砂壳虫食物组成的研究中。本研究采用构建克隆文库的分子生物学方法分析砂壳虫食物组成。通过采集样品,分离活体瘤棘砂壳虫,对总DNA提取,设计18S rRNA基因特异引物,构建克隆文库并进行有效序列的分析,进而分析食物的物种组成。此方法操作简单,试验周期短,重复性好,为研究砂壳虫摄食行为特点提供了新的研究手段。
1 材料与方法 1.1 样品采集2010年9月在厦门市湖边水库(24°30′N,118°09′E)水体表层水平及垂直方向上利用网孔直径为64 μm的浮游生物网拖拽捞取样品,将水样转入50 mL PVC瓶,并快速送至实验室。在倒置显微镜下挑取瘤棘砂壳虫虫体,超纯水清洗体表3—5次,以避免外源DNA的污染。选取50个个体装入1.5 mL的无菌离心管中。置于-20 ℃保存,以备进行总DNA提取。
1.2 DNA提取配制异硫氰酸胍法DNA提取液[12],将瘤棘砂壳虫转移到含有100 μL DNA提取液的离心管中,轻微震荡。混匀后,置于65 ℃水浴锅加热10—30 min,利用无菌枪头破碎虫体。再加入100 μL异丙醇,震荡混匀,置于4℃冰箱中沉淀过夜。次日,取出冰箱中的样品,在4 ℃,14000 r/min条件下离心20 min,弃上清液。将预冷的250 μL 70%乙醇加入沉淀物DNA中,混匀漂洗,在4 ℃,14000 r/min条件下离心5 min,弃上清液。再加入250 μL无水乙醇洗涤DNA,在4 ℃,14000 r/min条件下离心5 min,弃上清液。将沉淀物置于无菌处风干1 min,加入适量蒸馏水溶解所提取的DNA,置于-20 ℃冰箱中保存备用。
1.3 基因的PCR扩增及纯化选择18S rRNA基因通用引物Euk-A(5′-AACCTGGTTGATCCTGCCAGT-3′)和 Euk-B(5′-TGATCCTTCTGCAGGTTCACCTAC-3′)为PCR扩增引物[13]。PCR反应体系为50 μL,包括5 μL 10 × Ex PCR缓冲液(10 mmol/L,Mg2+终浓度为1.5 mmol/L),4 μL dNTP(10 mmol/L),0.25 μL Ex Taq酶(5 U/μL),PCR前后引物各1 μL(10 μmol/L),36.75 μL ddH2O和2 μL DNA模板。PCR反应条件如下:95 ℃预变性4 min;40个循环为94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸1.5 min;最后72 ℃延伸10 min。
PCR产物用经EB染色的1.0%琼脂糖凝胶电泳检测。从凝胶上切下特异性扩增的目的条带,置于无菌的1.5 mL Eppendorf管中,利用凝胶回收试剂盒(Wizard SV Gel and PCR Clean-up System)纯化回收18S rRNA基因片段。
1.4 测序及序列分析将纯化后的PCR产物(18S rRNA基因)连接到pGEM-T载体,然后导入大肠杆菌DH5α感受态细胞。涂布到含有50%氨苄青霉素(Ampicillin)的LB固体培养基上孵育、培养并进行蓝白斑筛选,挑取出白色克隆进行PCR检测。将正确插入目的片段的50个白色克隆样品进行基因测序分析,应用DNASTAR软件对测序结果进行编辑,去除载体序列。将所获的序列在NCBI数据库(http:www.ncbi.nlm.nih.gov/blast)中进行比对,挑选与其亲缘关系最相近的已知序列以确定其分类归属。利用Mothur v.1.22.2软件分析操作分类单元(OTUs,Operational taxonomic units),按97%的相似度进行划分。基于OTU结果,计算Chao1值、ACE值[14, 15],并绘制稀释曲线。
1.5 序列登录号本研究获得的18S rRNA基因序列在GenBank中的登录号为:KF910075—KF910085。
2 结果 2.1 18S rRNA基因多样性分析本研究中,PCR扩增获得的18S rRNA基因片段大小约为1500 bp。对挑选的50个阳性克隆进行测序分析,除4个低质量克隆未成功测序外,共得到46条序列。
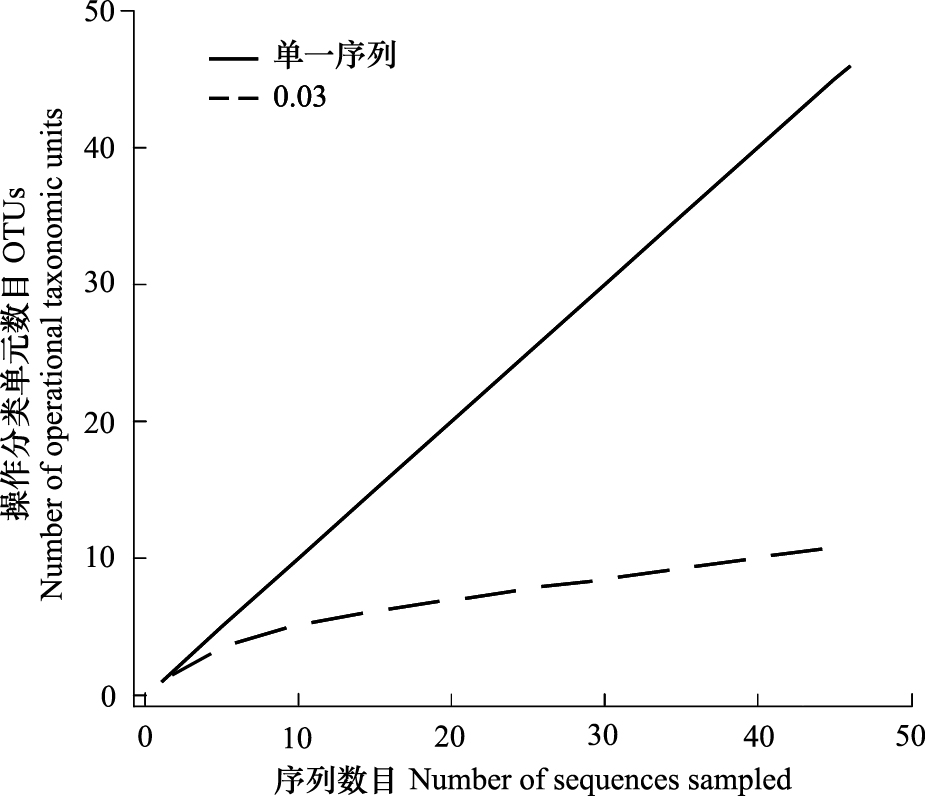
46条基因序列在97%相似度水平归为11类OTU(图 1)。多样性指数Chao1值为32,ACE指数为74。其中,OTU1及OTU2丰度最高,各自包含了12条序列,表明OTU1及OTU2在砂壳虫体内丰度较高(图 2)。OTU3是8条序列,OTU4有7条序列。其他7个OTU均包括1条序列,相对丰度较低。
|
| 图 1 18S rRNA基因序列稀释曲线 Fig.1 Rarefaction curves for 18S rRNA gene sequences |

|
| 图 2 18S rRNA基因克隆文库中OTU0.03等级丰度 Fig.2 Rank-abundance distribution of OTU0.03 based on 18S rRNA gene clone library |
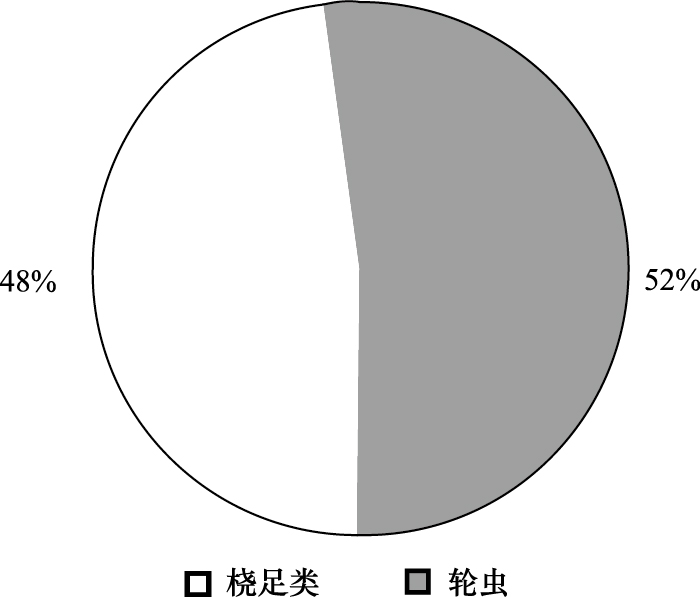
基于系统发育地位分析表明,所有OTUs均可划归为两大类浮游动物:52%的OTUs序列属于轮虫(Rotifera),48%的OTUs序列是桡足类(Copepoda)的哲水蚤(表 1,图 3)。其中6个OTUs属于轮虫,包括24条序列;5个OTUs属于桡足类,包括22条序列。由表 1可知,轮虫主要种类包括矩形龟甲轮虫(Keratella quadrata)、对壶状臂尾轮虫(Brachionus urceolaris)、大肚须足轮虫(Euchlanis dilatata)、方块鬼轮虫(Trichotria tetractis)、萼花臂尾轮虫(Brachionus calyciflorus)、剪形巨头轮虫(Cephalodella forficula);桡足类包括Mastigodiaptomus nesus(镖水蚤科一种),拟细真镖水蚤(Eudiaptomus graciloides),Eudiaptomus gracilis(镖水蚤科一种),Skistodiaptomus oregonensis(镖水蚤科一种),Leptodiaptomus moorei(镖水蚤科一种)。以上结果提示,瘤棘砂壳虫体内出现轮虫及桡足类的18S rRNA基因,推测瘤棘砂壳虫不仅以轮虫为食,而且可以捕食桡足类。
| 操作分类单元Operational taxonomic units,OTUs | 序列登录号Accession number | 分类类群Taxon | 序列最相似物种(序列登录号)Closest relative (accession number) | 相似度/%Similarity |
| 1 | KF910075 | Rotifera | Keratella quadrata (DQ297697) | 99 |
| 2 | KF910076 | Copepoda | Mastigodiaptomus nesus (AY339156) | 99 |
| 3 | KF910077 | Rotifera | Brachionus urceolaris (DQ089734) | 99 |
| 4 | KF910078 | Copepoda | Eudiaptomus graciloides (AY339149) | 99 |
| 5 | KF910079 | Rotifera | Euchlanis dilatata (AY218116) | 93 |
| 6 | KF910080 | Copepoda | Eudiaptomus gracilis (AY339148) | 94 |
| 7 | KF910081 | Copepoda | Skistodiaptomus oregonensis (AY339159) | 97 |
| 8 | KF910082 | Rotifera | Trichotria tetractis (DQ297724) | 97 |
| 9 | KF910083 | Rotifera | Brachionus calyciflorus (DQ297692) | 95 |
| 10 | KF910084 | Copepoda | Leptodiaptomus moorei (AY339154) | 93 |
| 11 | KF910085 | Rotifera | Cephalodella forficula (DQ297693) | 95 |
|
| 图 3 18S rRNA基因克隆文库的物种分类组成 (n=46) Fig.3 Relative abundance of different taxonomic groups based on 18S rRNA gene clone library (n=46) |
基于显微镜观测的研究表明,瘤棘砂壳虫属肉食性动物,会攻击其周围任何微小的生物,尤其是大小相近的微型生物,主要摄食以轮虫为主的浮游生物,包括胶鞘轮虫、独角聚花轮虫、螺形龟甲轮虫、红多肢轮虫、奇异六腕轮虫、对棘异尾轮虫,以及无节幼体[6, 7, 16]。瘤棘砂壳虫的捕食行为主要依赖伪足,伪足可以自由伸缩[17],并且可以准确判断所要捕食对象的位置。在广东流溪河水库轮虫中,胶鞘轮虫和独角聚花轮虫容易被瘤棘砂壳虫摄食,因为它们身体柔软、游泳速度慢;而龟甲轮虫、红多肢轮虫的外壳硬,游泳速度快,不易被瘤棘砂壳虫捕食[7, 16]。针对不同轮虫物种,瘤棘砂壳虫的捕食方式是不一样的。如遇到身体柔软的胶鞘轮虫,瘤棘砂壳虫一般会攻击它们的背部或尾部,容易捕食成功;而对于外壳比较硬的龟甲轮虫,选择先攻击它们的口部,捕食成功率较低[6, 16]。毫无疑问,我们的结果也进一步表明,作为单细胞的瘤棘砂壳虫能够捕食多细胞的后生动物(表 1)。
有趣的是,除了在瘤棘砂壳虫体内检测到轮虫18S rRNA基因序列外,还检测到相当比例的桡足类,进而证明湖边水库瘤棘砂壳虫主要以轮虫及桡足类为食。本研究检测到瘤棘砂壳虫捕食的轮虫及桡足类种属与韩博平等[6, 7, 16]学者观察到的物种有所差异,推测这与不同生境及被捕食物种的丰富度相关。同时,本研究进一步补充扩展了瘤棘砂壳虫捕食对象的范围。由于桡足类的个体相对于瘤棘砂壳虫要大很多,所以捕食相对较难。最近,韩博平等[16]观察到瘤棘砂壳虫可以捕食其周围死亡的桡足类,但关于活体桡足类的捕食尚未见报道。不过,本文推测瘤棘砂壳虫能够捕食桡足类幼体,因为桡足类幼体个体相对较小,并且经历的幼体阶段相对较长。
事实上,瘤棘砂壳虫还会捕食藻类,包括甲藻、丝状藻类等[6]。但在本研究中没有检测到藻类,这并不代表瘤棘砂壳虫没有捕食藻类。究其原因,一方面可能是我们所用的18S rRNA基因引物具有一定特异性,扩增序列产生一定的偏好性,导致没有检测到藻类。另一方面,瘤棘砂壳虫捕食的其他类食物数量不够多,或者已经消化掉了,低于检测线,因而也没能检测到。未来的深入研究有必要筛选更合适的PCR扩增引物和优化PCR条件。
3.2 基因克隆文库技术是一个有效的食物组成分析方法传统的单细胞生物食物组成和捕食行为研究主要是利用显微活体观察技术,在观察时需要具有分类学背景的专业人才,处理大量样本耗时较长,因此分析周期长[18]。而近年来兴起的分子生物学技术方法的方便、高效、快速,弥补了活体观察的这一缺陷。本研究中,利用18S rRNA基因序列克隆文库的分子生物学技术手段,能够发现瘤棘砂壳虫体内含有轮虫及桡足类的基因序列,证明此方法可行可信。因此,单细胞原生动物体内18S rRNA基因的检测可以作为一个有用的工具来快速地分析瘤棘砂壳虫的食物组成。虽然分子生物学技术手段能够快速检测食物组成,但不能直观地观测其捕食行为过程,因此要配合显微镜活体观察来共同研究瘤棘砂壳虫的捕食特征(表 2)。
| 方法Method | 显微观察[6, 7, 16]Microscopic method[6, 7, 16] | 分子方法(本研究)Molecular method (this study) |
| 物种组成Species composition | 轮虫(胶鞘轮虫、聚花轮虫、龟甲轮虫、多肢轮虫、六腕轮虫、异尾轮虫); | 轮虫(龟甲轮虫、臂尾轮虫、鬼轮虫、须足轮虫、巨头轮虫); |
| 桡足类(桡足类幼体) | 桡足类(哲水蚤镖水蚤科5种) | |
| 优点Advantages | 可直观的观察捕食过程;能分辨所捕食生物的死活 | 高效、可获得较高的物种多样性;快速、实验周期短;方便、易设置重复试验;对分类学知识要求不高 |
| 缺点Disadvantages | 观察捕食行为耗时长,实验周期长;需要分类学背景的专业人才;受半透明壳体的影响,对于体内食物观察困难;一些不完整的虫体难于进行物种鉴定 | 一些引物具有偏好性;不能直观的观察捕食过程;不能区分所捕食生物的死活 |
为进一步深入全面认知瘤棘砂壳虫的捕食行为特征,揭示其在水生态系统结构、功能等方面的重要作用和意义,建议后续研究综合显微活体观察与分子生物学方法,达到技术方法上相互补充的效果。
| [1] | Meisterfeld R. Order Arcellinida Kent, 1880//Lee J J, Leedale G F, Bradbury P. An Illustrated Guide to the Protozoa. 2nd ed. Lawrence, USA: Allen Press, 2002: 827-860. |
| [2] | 胡东利, 沈韫芬, 顾曼如, 龚循矩. 武陵山地区原生动物新种和新纪录//宋大祥. 西南武陵山地区无脊椎动物. 北京: 科学出版社, 1997: 40-72. |
| [3] | Yang J, Beyens L, Shen Y F, Feng W S. Redescription of Difflugia tuberspinifera Hu, Shen, Gu et Gong, 1997 (Protozoa: Rhizopoda: Arcellinida: Difflugiidae) from China. Acta Protozoologica, 2004, 43(3): 281-289. |
| [4] | 刘乐冕, 杨军, 张文静, 余正. 原生动物瘤棘砂壳虫六个自然种群的形态计量学分析. 动物学研究, 2010, 31(4): 435-443. |
| [5] | Yu Z, Zhang W J, Liu L M, Yang J. Evidence for two different morphotypes of Difflugia tuberspinifera from China. European Journal of Protistology, 2014, 50(2): 205-211. |
| [6] | Han B P, Wang T, Lin Q Q, Henri J D. Carnivory and active hunting by the planktonic testate amoeba Difflugia tuberspinifera. Hydrobiologia, 2008, 596(1): 197-201. |
| [7] | Wang T, Han B P. Food selectity of Difflugia tuberspinifera in Liuxihe Reservoir, South China. Ecological Science, 2008, 27(5): 398-401. |
| [8] | 沈韫芬. 西藏高原的原生动物//蒋燮治, 沈韫芬, 龚循矩. 西藏水生无脊椎动物. 北京: 科学出版社, 1983: 48-100. |
| [9] | 杨军, Beyens L, 沈韫芬, 冯伟松. 瘤棘砂壳虫 (肉足亚门: 根足总纲) 壳体元素组成. 动物学研究, 2004, 25(5): 452-455. |
| [10] | Wanner M. A review on the variability of testate amoebae: methodological approaches, environmental influences and taxonomical implications. Acta Protozoologica, 1999, 38(1): 15-29. |
| [11] | Gomaa F, Todorov M, Heger T J, Mitchell E A D, Lara E. SSU rRNA phylogeny of Arcellinida (Amoebozoa) reveals that the largest Arcellinid Genus, Diffugia Leclerc 1815, is not monophyletic. Protist, 2012, 163(3): 389-399. |
| [12] | Chomczynski P, Sacchi N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry, 1987, 162(1): 156-159. |
| [13] | Medlin L K, Elwood H J, Stickel S, Sogin M L. The characterization of enzymatically amplified eukaryotic 16S-like rRNA-coding regions. Gene, 1998, 71(2): 491-499. |
| [14] | Hill T C J, Walsh K A, Harris J A, Moffett B F. Using ecological diversity measures with bacterial communities. FEMS Microbiology Ecology, 2003, 43(1): 1-11. |
| [15] | Kemp P F, Aller J Y. Bacterial diversity in aquatic and other environments: what 16S rDNA fibraries can tell us. FEMS Microbiology Ecology, 2004, 47(2): 161-177. |
| [16] | Han B P, Wang T, Xu L, Lin Q Q, Zhang J Y, Dumont H J. Carnivorous planktonic Difflugia (Protista, Amoebina Testacea) and their predators. European Journal of Protistology, 2011, 47(3): 214-223. |
| [17] | 张文静, 杨军, 田野. 有壳肉足虫的结构、运动以及生殖. 生物学通报, 2010, 45(1): 12-14. |
| [18] | Gilbert D, Mitchell E A, Amblard C, Bourdier G, Francez A J. Population dynamics and food preferences of the testate amoeba Nebela tincta major-bohemica-collaris complex (Protozoa) in a Sphagnum Peatland. Acta Protozoologica, 2003, 42(2): 99-104. |
 2015, Vol. 35
2015, Vol. 35