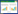文章信息
- 杨飞龙, 李旭东, 闫振天, 付文博, 陈斌
- YANG Feilong, LI Xudong, YAN Zhentian, FU Wenbo, CHEN Bin
- 云南中华按蚊的遗传变异和种群结构
- Genetic variation and population structure of Anopheles sinensis (Diptera:Culicidae) in Yunnan
- 生态学报, 2015, 35(16): 5449-5457
- Acta Ecologica Sinica, 2015, 35(16): 5449-5457
- http://dx.doi.org/10.5846/stxb201311172746
-
文章历史
- 收稿日期:2013-11-17
- 修订日期:2014-08-20
中华按蚊(Anopheles sinensis)是按蚊亚属赫坎按蚊种团中研究比较多的蚊种,它广泛分布于东亚[1, 2, 3],在我国分布于东经100—120°,北纬19—54°[4],属于按蚊亚属中的优势种。近年来,中华按蚊被认为是疟疾特别是间日疟的重要传播媒介[5],亚洲很多地区都有其疟疾传播的报道。在韩国,该种被认为是重要的传疟媒介[6, 7],主要传播间日疟[8, 9]。在日本,中华按蚊分布比较广泛,一直被认为是日本温带地区(包括冲绳及北海道)最重要的疟疾传播媒介[2, 3]。在我国,中华按蚊更多的被认为是间日疟原虫的传播媒介而非恶性疟原虫的传播媒介[4, 10],特别是我国的云南省,由于地处热带和亚热带地区,中华按蚊种群受季节影响较小;同时云南省水稻田分布广泛,致使中华按蚊成为其分布最广泛、最主要的传疟媒介之一;加之云南和缅甸、老挝、越南接壤,常年境外疟疾的输入,使疟疾防治工作变得十分困难,进一步加大了云南疟疾的流行趋势。
尽管中华按蚊具有重要医学意义,但关于其种群遗传结构相关的报道却很少。种群遗传结构的研究有助于阐明自然条件下物种内的遗传变异,及其与地理环境的关系以及物种形成的机理,加深对物种进化过程的认识[11],从而对物种的防治或保护提供重要科学依据。目前,蚊虫的控制主要依赖杀虫剂的使用,理解种群的遗传分化有助于推断杀虫剂抗性的现状和可能扩散途径,从而指导杀虫剂的使用。马雅军对我国部分中华按蚊群体分子遗传多态性进行了研究[12],云南省相对于中国其他地区,拥有其独特的气候条件和复杂的地理环境,中华按蚊种群结构具有一定差别。因此,本研究通过收集云龙、腾冲、盈江、中缅边境、双江、景洪、勐腊、元江和元阳9个采集点的中华按蚊标本,分析其线粒体COII序列,进一步对云南的中华按蚊遗传变异和种群结构进行了研究。
1 材料和方法 1.1 材料本研究的样本为中华按蚊野外成蚊,共89头,采于2010年10月到2012年9月,采自云南省中缅边境、盈江、景洪、勐腊、双江、腾冲、元江、元阳和云龙9个采样点,并根据9个采样点的地理位置间的距离划分为5个种群组(表1,图1)。采集后的样本用85%酒精保存在-20℃冰箱中。
| 种群组 Population group | 地点 Location | 编号 Code | 样本数 Sample size | 经纬度 Latitude and longitude | 采集时间 Time |
| 中缅边境盈江腾冲 | 中缅边境 | ZM | 9 | 24.6°N,97.6°E | 2012-08 |
| 种群组 | 盈江 | YJ | 5 | 24.6°N,97.6°E | 2012-08 |
| 腾冲 | TC | 7 | 25.0°N,98.5°E | 2011-07 | |
| 云龙种群组 | 云龙 | YL | 4 | 25.9°N,99.4°E | 2011-07 |
| 双江种群组 | 双江 | SJ | 12 | 23.4°N,99.8°E | 2011-08 |
| 景洪勐腊种群组 | 景洪 | JH | 12 | 22.0°N,100.7°E | 2011-08 |
| 勐腊 | ML | 8 | 21.5°N,101.6°E | 2011-08 | |
| 元江元阳种群组 | 元江 | YUJ | 15 | 23.6°N,102.0°E | 2011-08 |
| 元阳 | YY | 17 | 23.2°N,102.8°E | 2010-07,2012-08 |
|
| 图 1 云南省中华按蚊9个采样点及5个种群组的地理位置 Fig.1 Nine collecting localities of An. sinensis populations in Yunnan,and five population groups grouped based on their geographical distributions |
形态学上中华按蚊的主要鉴定部位包括触须、翅脉、腹部和后足跗节。本研究根据董学书编著的《云南蚊虫志》检索表[13],在显微镜下对中华按蚊标本主要部位进行鉴定,筛选出中华按蚊。
1.2.2 DNA提取根据QIAGEN试剂盒使用说明提取中华按蚊基因组DNA。提取步奏:(1)组织样品与 Buffer ATL混合,磨碎,再加入蛋白酶K,56 ℃水浴过夜。(2)再依次加入Buffer AL、无水乙醇,震荡混匀。(3)将混匀液加入到DNeasy Mini spin column中,6000r/min离心1 min。(4)将spin column转移到新的收集管中,加入Buffer AW1,6000r/min离心1 min。(5)将spin column转移到新的收集管中,加入Buffer AW2,20000r/min离心3 min。(6)将柱子转移到1.5 mL的离心管中。(7)加入Buffer AE到柱子膜上,在室温下放置1 min,在6000r/min下离心分离1 min。(8)重复第7步,然后将提取的DNA放于-20 ℃冰箱保存备用。
1.2.3 分子鉴定由于形态学鉴定存在一定误差,因此将提取的DNA样品,进行分子鉴定。扩增28S rDNA ITS2片段,扩增引物,D1:5′-TGTGAACTGCAGGACACATGAA-3′,D2:5′-AGGGTCAAGGCATACAGAAGGC-3′[14]。PCR反应总体积为25 μL,PCR扩增的反应体系:模板DNA 1 μL,正反引物(10 μmol/L)各1 μL,再加入Taq Mastermix到25 μL。反应程序:95 ℃预变性5 min,95 ℃变性40 s,55 ℃退火40 s,72 ℃延伸1 min,35个循环后,72 ℃延伸6 min。PCR产物进行电泳检测,结果为目标条带1077 bp的PCR产物,送上海生物工程有限公司测序,将得到的序列放入NCBI进行比对。
1.2.4 COII基因PCR扩增和测序对分子鉴定为中华按蚊的样品,进行COII基因PCR扩增。扩增引物,LEU (forward): 5′-TCTAATATGGCAGATTAGTGCA-3′ 和LYS (reverse): 5′-ACTTGCTTTCAGTCATCTAATG-3′[15]。PCR反应总体积为25 μL,PCR扩增的反应体系:模板DNA 1 μL,正反引物(10 μmol/L)各1 μL,再加入Taq Mastermix到25 μL。反应程序:95 ℃预变性5 min,95 ℃变性1 min,51 ℃退火1 min,72 ℃延伸2 min,35个循环后,72 ℃延伸10 min后4 ℃保存。PCR产物用1.5%琼脂糖凝胶电泳检测样品扩增情况,对成功扩增的样品送上海生工生物工程有限公司进行双向测序。
1.3 数据分析使用MEGA5.1[16]软件将测序得到的序列进行同源比对和构建单倍型邻接(NJ)树,Bootstrap重复抽样1000次检验。用DnaSP 5.0[17]软件计算各地理中华按蚊种群的单倍型多样型指数(Haplotype diversity,h)、核苷酸多样性指数(Nucleotide diversity,π)、多态性位点个数(Polymorphic sites,s)[18],绘制单倍型歧点分布图[19],计算种群间的遗传分化Fst值和基因交流Nm值。利用Arlequin v3.1[20]中的分子变异分析(AMOVA),根据pairwise difference模型,进行分子方差分析,评估种群内与种群间的遗传分化,并进行中性检测(Tajima′s D检测和Fu & Li检测),以检测云南省中华按蚊种群是否符合中性变异。应用Network 4.0[21]构建单倍型网络结构图,根据不同地理关系分析各种群之间的进化关系,以追溯单倍型间的亲缘关系。
2 结果与分析 2.1 中华按蚊形态和分子鉴定从形态学上初步筛选出中华按蚊标本95头,对95头中华按蚊标本开展分子鉴定,有89头标本电泳结果产生了明显的中华按蚊条带(图2)。将扩增的89份PCR产物测序后使用Blast在NCBI数据库中比对,序列与NCBI数据库中的已知中华按蚊28S rDNA ITS2序列相似度为99%—100%。

|
| 图 2 中华按蚊分子鉴定PCR产品电泳图 Fig.2 The agarose gel electrophoresis of PCR product for molecular identification of An. sinensis |
对获得的89条中华按蚊COII序列进行拼接比对,在这些序列中共检测到39个单倍型,占总样本数89的43.8%。A、T、C、G的平均含量分别为:35.99%、39.51%、12.58%、11.91%,A+T含量(75.37%)明显高于G+C含量(24.63%),COII碱基组成具有很大的偏向性,G的含量很低。在所分析的序列中,共检测到变异位点51个(图3),占分析位点总数739 bp的6.9%,其中简约信息位点17个,单一突变位点34个。在不同的群体中变异位点最多的是元江元阳种群组,有24个,变异位点最低的是云龙种群组,有7个。

|
| 图 3 中华按蚊COII基因变异位点 Fig.3 Variation sites of the COII gene in An. sinensis |
总体的单倍型多样性指数较高,但核苷酸多样性指数较低(表2)。其中,云龙种群组单倍型多样性指数最高为1.000,采集到4头标本,分属4个单倍型,但核苷酸多样性不高,说明个体间遗传差异较小;其次是景洪勐腊种群组单倍型指数为0.937,中缅边境盈江腾冲种群组最低,为0.890。云龙种群组核苷酸多样性也是最高,为0.00496,说明相对于其他种群个体间遗传差异较大;其次是景洪勐腊种群组,为0.00429;元江元阳种群组最低,为0.00406。所有单采集样点种群作为一个种群进行分析,单倍型多样性指数为0.931,核苷酸多样性指数为0.00466。
| 种群 Population group | 样品数 Sample size | 单倍型( N *) Haplotypes( N *) | 单倍型多样性 Haplotype diversity( h) | 核苷酸多样性 Nucleotide diversity( π) | 变异位点数 Polymorphic sites( s) |
| 中缅边境盈江腾冲种群组 | 21 | 1(6)、2(3)、3(2)、4(3)、5、9(2)、19、20、21、22 | 0.890 | 0.00419 | 15 |
| 云龙种群组 | 4 | 4、37、38、39 | 1.000 | 0.00496 | 7 |
| 双江种群组 | 12 | 1(6)、2、13、16、17、18 | 0.773 | 0.00416 | 15 |
| 景洪勐腊种群组 | 20 | 1(5)、2、3(2)、4、6、7、8、9(2)、10、11、12、13、14、15 | 0.937 | 0.00429 | 18 |
| 元江元阳种群组 | 32 | 1、2、4、9(6)、12(6)、23(2)、24、25(2)、26、27、28、29、30、31、32、33(2)、34、35、36 | 0.933 | 0.00406 | 24 |
| 总计 Total | 89 | 39 | 0.931 | 0.00466 | 51 |
89头中华按蚊标本COII基因共检测到39个单倍型。其中H1、H2、H9分布最广,除了云龙种群组外,这些单倍型在其余种群组均有分布,可能是在云龙种群组采样数目较少,单倍型类型相应少。H1单倍型频率最高,为20.2%(18/89),而H2、H9单倍型频率分别为6.7%和12.4%;H12主要分布在元江元阳种群组和少部分的景洪勐腊组种群,频率为7.9%;H3分布于中缅边境盈江腾冲种群组和景洪勐腊种群组,频率为4.5%;其他单倍型频率都很低,其中有29个单倍型为种群独有单倍型,分布于所研究的5个种群组,各个地区独有单倍型的多少可能跟采集样本的多少或者跟本身地理环境引起的遗传变异有关。
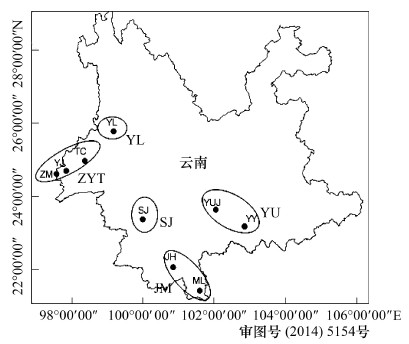
2.3 系统发育分析和单倍型网络关系用MEGA构建的NJ树(图4)显示单倍型系统发育关系的支持率大部分很低,且主要分为两个大枝,分别由12个单倍型和27个单倍型组成,但每个大枝都涉及5个种群组的单倍型分布,单倍型拓扑结构并没有显示出与地理位置相对应关系的聚集,这说明云南的中华按蚊没有形成明显的地理遗传变化。
|
| 图 4 中华按蚊COII序列NJ树 Fig.4 NJ tree of An. sinensis COII sequences |
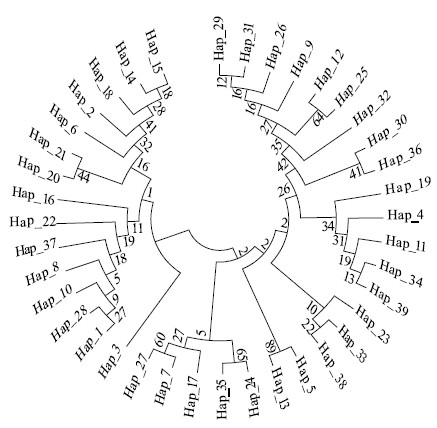
用Network的Median-joining方法构建的单倍型网络图(图5),图中饼图大小与单倍型出现频率成正比。由图可知大部分种群地理位置与单倍型间没有显示出明显的对应关系,但元江元阳种群组单倍型存在与地理位置明显的对应关系。所有单倍型以星状网络结构进行分布,H1、H9、H4、H33和H2是主要的单倍型,其他单倍型以这5个单倍型为中心,辐射分布开来。其中H1单倍型占据主导优势,可能是这些单倍型中最为原始的单倍型,其他单倍型由其进化而来。
|
| 图 5 单倍型的进化网络图 Fig.5 Network relationship of haplotypes |
将云南的9个采集地点划分为5个中华按蚊种群组进行分子变异分析,结果显示种群组间遗传变异为12.58%,达到显著水平(P=0.04888)(表3),说明地理种群组间具有明显种群遗传结构。Fst值显示(表4)大部分种群间没有出现明显的遗传分化现象,但元江元阳种群组除了和云龙种群组没有遗传分化外和其余3个种群都存在明显的遗传分化,同时Nm值显示元江元阳种群组除了和云龙种群组存在明显的基因交流外和其他种群的基因交流并不明显,两组数据反映结果一致,这一结果解释了在AMOVA中种群间的显著性接近于0.05,可能是受到元江元阳种群组的影响。尽管其他种群间的遗传分化不明显,但元江元阳种群组和其他种群存在明显遗传分化,具有明显的种群遗传结构。
| 变异来源 Source of variation |
自由度 df |
平方和 Sum of squares |
变异组成 Variance components |
变异率/% Percentage of variation |
F | P |
| 种群组间 Among groups | 4 | 21.554 | 0.229Va | 12.58 | FCT=0.128 | 0.04888 |
| 种群间 Among populations within groups | 4 | 6.407 | 0.006Vb | 0.32 | FSC=0.004 | 0.37537 |
| 群体内 Within populations | 80 | 123.690 | 1.546Vb | 86.93 | FST=0.132 | |
| 合计 Total | 88 | 151.652 | 1.781 |
| 种群组 Population group | 1 | 2 | 3 | 4 | 5 |
| 1 中缅边境盈江腾冲种群组 | 18.70 | 21.97 | -9.03 | 1.09 | |
| 2 云龙种群组 | 0.013 | 2.50 | 10.71 | 2.47 | |
| 3 双江种群组 | 0.011 * | 0.090 | -38.01 | 0.69 | |
| 4 景洪勐腊种群组 | -0.028 * | 0.023 * | -0.007 * | 1.19 | |
| 5 元江元阳种群组 | 0.186 | 0.092 | 0.266 | 0.173 * |
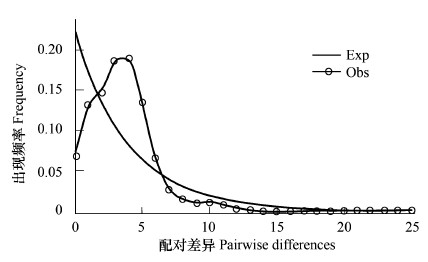
COII序列的中性测试结果显示Tajima′s D = -2.252,P < 0.01; Fu and Li′s F* test statistic = -4.892,P < 0.02。表明目标序列在进化上遵循中性模型,都为负值,同时歧点分布结果显示单倍型和碱基差异呈单峰分布(图6),说明云南地区中华按蚊种群近期经历过种群扩张事件。
|
| 图 6 中华按蚊种群歧点分布检测 Fig.6 Mismatch-distribution of Anopheles sinensis COII haplotypes |
本研究从云南省9个采样点广泛采集、测序和分析了89头中华按蚊标本。89头样本检测出39个COII单倍型,占总体样本个数43.8%,各个种群组的单倍型个数百分比都几乎超过了50%,说明云南中华按蚊具有较高的遗传性多样性。虽然不同地区的遗传多样性有一定的差别,但总体差别不大,核苷酸多样性均在0.00406—0.00496之间。中华按蚊受地理分布气候影响的同时,由于其成蚊偏好吸嗜牛血、人血,幼虫喜好水稻田等生态特点,是总体遗传多样性差别不大的主要原因。单倍型多样性和核苷酸多样性最高的是云龙种群组云龙,由于云龙的样本数较少(仅4个样本),分析结果存在一定误差;不考虑云龙遗传多样性的情况下,单倍型多样性和核苷酸多样性最高的是景洪勐腊种群组景洪勐腊。景洪勐腊地处西双版纳州,属于热带雨林气候,相对于云南其他地区,常年气温较高,气候湿润,同时西双版纳州被联合国教科文组织评为“国际生物圈保护区”,生态环境良好,这些因素是这一地区的中华按蚊遗传多样性十分丰富的重要因素。尽管元江元阳种群组的样本数是最多的,但其单倍型多样性和核苷酸多样性都不是最高,这主要是所处的元江元阳地区,常年气候干燥,气温较低,四季交替明显,并不利于中华按蚊种群遗传分化。云南地处边境,常年人口流动大,境外疟疾的输入,导致中华按蚊的传疟作用尤为突出,所以云南中华按蚊疟疾传播的防治工作一直是难点,掌握中华按蚊遗传多样性及分化,对中华按蚊及疟疾控制具有重要的作用。
3.2 种群遗传结构本研究基于mtDNA的COII序列建树分析显示(图4),云南中华按蚊各个种群的单倍型呈现出一种混杂的分布格局,没有检测到与采样点严格对应的分支,没有形成明显的系统地理格局,这与马雅军等研究的结论一致。马雅军认为中国的中华按蚊种群的分化与其地理距离或者障碍(地理隔离)没有关系[12],形成这种结果的原因主要是我国中华按蚊易变的有效种群大小和其他一些因素,比如杀虫剂和对抗疟疾的免疫基因等都可能影响到种群的分化。而单倍型网络结构图却显示(图5),主要以单倍型H1、H9、H4、H33和H2为中心呈星状分布。尽管大部分种群单倍型没有按采样地形成各自的分支,但元江元阳种群组的单倍型分布存在明显的地理分布,这说明马雅军的这一观点对云南大部分中华按蚊地理分布相符合,但在个别地区仍有差别。
为了进一步了解其地理格局和种群结构,把9个采样点的样本按地理分布划分为5个种群组,AMOVA分析显示种群组间遗传变异为12.58%,达到显著水平(P=0.04888)(表3),说明地理种群组间具有明显种群遗传结构。通过Nm和Fst分析表明,云南中华按蚊大部分种群间遗传分化较小,种群间存在广泛的基因交流,但元江元阳种群组除了和云龙种群组有基因交流外和其他种群间都缺乏明显的交流,和其他种群存在明显分化现象。从理论上讲中华按蚊体型较小不适宜长距离迁飞,受限于自身的扩散能力,一定程度的隔离即可显著限制种群的基因交流,从而导致种群间形成明显的遗传分化,但群体分化程度主要是对所在的生态条件相适应的结果,如果生态条件所产生的环境作用强度和方向大体相同,则各个分布区内的种群在遗传上将难以形成显著的分化[22]。同时一些自然因素(如风力,河流流动)和人类活动也可能使远距离的种群间产生基因交流。哀牢山是云南南北走向的大山,成为云贵高原和横断山脉两大地貌的分界线,它的出现使云南呈现东西不同地理气候,西南部地势较低,常年降水较多,气候湿润,而东部高原气候雨水少,干燥,常年平均气温较低,本次涉及的采样点元江、元阳位于云南中南部紧邻哀牢山南端,导致元江元阳种群组和其他种群组分别处于两种不同的气候区,同时哀牢山的存在也阻断了元江元阳种群组和其他种群组的基因交流,导致元江元阳种群组的中华按蚊朝着不同的方向进化,使元江元阳种群组和其他种群组间存在明显遗传差异。而其他4个种群间遗传分化较小主要是因为他们处于同一气候环境中,加上水稻田是中华按蚊主要的生活环境,而在云南省水稻田分布广泛,同时现行的水稻杀虫剂也都主要是毒死蜱、敌百虫、敌敌畏等常用杀虫剂,中华按蚊的生活环境和环境压力大体一致,导致这些地区种群并没有形成明显的分化特征。同时澜沧江是横跨云南南北处于云南西部的一条大江,本研究所涉及的采样点除了元江元阳种群组外其他种群都分布在其周围,澜沧江水流流动在一定程度上也促进了这些地区中华按蚊的基因交流。
3.3 种群历史动态Tajima′D中性检测值在一定程度内可以反应种群的历史动态,一个负D值表明存在净化选择或群体中有轻微分离的有害突变,也可能是由于群体的扩张引起的。正D值可解释为平衡选择将突变保持在平衡频率,也有可能是一个群体收缩导致的[23]。本研究中性检测结果均为负值并且显著,说明整体种群中存在许多的低频率等位基因突变,歧点分布为明显单峰分布,单倍型网络图反应种群经历过种群扩张,说明云南地区中华按蚊种群在历史上经历过复杂的种群扩张。同时从整个种群平均单倍型偏高,但核苷酸多样性偏低来看在种群发生扩张的初期云南的中华按蚊有效种群规模较小,后来经历了迅速的扩张。
引起种群扩张变化的主要因素是气候变化和地质运动,云南省地质十分复杂,在历史上也发生过多次地质变迁,特别是元古代的震旦纪到中生代的白垩纪晚期,云南经历了无数次不同的地壳运动,在第三纪始新世由于一系列的变化,一些类群的重新分布后在新的环境里得到繁衍和发展[24],中华按蚊种群可能在这个时期在适应了新的环境后发生了迅速扩张事件。到了第四纪喜马拉雅运动时期,地面大规模隆起,哀牢山的形成,阻断了元江元阳地区中华按蚊与云南其他地区种群的交流,从而逐渐形成了现在这种分布格局。
| [1] | Harrison B A, Scanlon J E. The subgenus Anopheles in Thailand (Diptera: Culicidae). Contributions of the American Entomological Institute, 1975, 12(1): 1-307. |
| [2] | Tanaka K, Mizusawa K, Saugstad E S. A revision of the adult and larval mosquitoes of Japan (Including the Ryukyu Archipelago and the Ogasawara Island) and Korea (Diptera: Culicidae). Contributions of the American Entomological Institute, 1979, 16: 1-987. |
| [3] | Rueda L M, Iwakami M, O'guinn M, Mogi M, Prendergast B F, Miyagi I, Toma T, Pecor J S, Wilkerson R C. Habitats and distribution of Anopheles sinensis and associated Anopheles hyrcanus group in Japan. Journal of the American Mosquito Control Association, 2005, 21(4): 458-463. |
| [4] | 陆宝麟. 中国动物志, 昆虫纲, 第九卷, 双翅目, 蚊科(下). 北京: 科学出版社, 1997: 12-38. |
| [5] | 周水森, 王漪, 房文, 汤林华. 2008年全国疟疾形势. 中国寄生虫学与寄生虫病杂志, 2009, 28(6): 455-457. |
| [6] | Chow C Y. Bionomics of malaria vectors in the Western Pacific region. Southeast Asian Journal of Tropical Medicine and Public Health, 1970, 1(1): 40-57. |
| [7] | Lee W J, Klein T A, Kim H C, Choi Y M, Yoon S H, Chang K S, Chong S T, Lee I Y, Jones J W, Jacobs J S, Sattabongkot J, Park J S. Anopheles kleini, Anopheles pullus, and Anopheles sinensis: potential vector of Plasmodium vivax in the Republic of Korea. Journal of Medical Entomology, 2007, 44(6): 1086-1090. |
| [8] | Ree H I, Hwang U W, Lee I Y, Kim T E. Daily survival and human blood index of Anopheles sinensis, the vector species of malaria in Korea. Journal of American Mosquito Control Association, 2001, 17(1): 67-72. |
| [9] | Coleman R E, Kiattibut C, Sattabongkot J, Ryan J, Burkett D A, Kim H C, Klein T A. Evaluation of anopheline mosquitoes (Diptera: Culicidae) from the republic of Korea for plasmodium vivax circumsporozoite protein. Journal of Medical Entomology, 2002, 39(1): 244-247. |
| [10] | 钱会霖, 汤林华, 程义亮, 杨宝金. 中华按蚊为唯一传疟媒介地区疟疾传播潜势的初步估算, 中国寄生虫学与寄生虫病杂志, 1994, 12(4): 265-267. |
| [11] | Merrell D J. Ecological Genetics. London: Longman, 1981: 453-491. |
| [12] | Ma Y J, Yang M N, Fan Y, Wu J, Ma Y, Xu J N. Population structure of the malaria vector Anopheles sinensis (Diptera: Culicidae) in China: Two gene pools inferred by Microsatellites. PLoS ONE, 2011, 6(7): e22219. |
| [13] | 董学书, 周红宁, 龚正达. 云南蚊类志(上卷). 昆明: 云南科技出版社, 2009: 144-146. |
| [14] | Joshi D, Park, M H, Saeung A, Choochote W, Min G S. Multiplex assay to identify Korean vectors of malaria. Molecular Ecology Resources, 2010, 10(4): 748-750. |
| [15] | Chen B, Pedro P M, Harbach R E, Somboon P, Walton C, Butlin R K. Mtiochondrial DNA variation in the malaria vector Anopheles minimus across China, Thailand and Vietnam: evolutionary hypothesis, population structure and population history. Heredity, 2011, 106(2): 241-252. |
| [16] | Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5: molecular evolutionary genetic analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. |
| [17] | Librado P, Rozas J. DnaSP v5: a sotfware for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452. |
| [18] | Nei M. Evolution of human races at the gene level//Bonne-Tamir B, Cohen T, Goodman R M. Human Genetics, part A: The Unfolding Genome. New York: Alan R. Liss, 1982: 167-181. |
| [19] | Roger A R, Harpending H. Population growth makes waves in the distribution of pairwise genetic differences. Molecular Biology and Evolution, 1992, 9(3): 552-269. |
| [20] | Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis. Evolutionary Bioinformatics, 2005, 1: 47-50. |
| [21] | Bandelt H J, Forster P, Rohl A. Median-joining network for inferring intraspecific phylogenies. Molecular Biology and Evolution, 1999, 16(1): 37-48. |
| [22] | Kelley S T, Farrell B D, Mitton J B. Effects of specialization on genetic differentiation in sister species of bark beetles. Heredity, 2000, 84(2): 218-227. |
| [23] | 梁日霞, 王振营, 何康来, 丛斌, 李菁. 基因线粒体COⅡ基因序列的双斑长跗萤叶甲中国北方地理种群的遗传多样性研究. 昆虫学报, 2011, 54(7): 828-837. |
| [24] | 朱世模, 黄复生. 地质变迁与云南白蚁的变化. 动物学研究, 1989, 10(1): 1-8. |
 2015, Vol. 35
2015, Vol. 35