文章信息
- 赵丹, 刘鹏飞, 潘超, 杜仁鹏, 葛菁萍
- ZHAO Dan, LIU Pengfei, PAN Chao, DU Renpeng, GE Jingping
- 生态代谢组学研究进展
- Advances in ecometabolomics
- 生态学报, 2015, 35(15): 4958-4967
- Acta Ecologica Sinica, 2015, 35(15): 4958-4967
- http://dx.doi.org/10.5846/stxb201401080064
-
文章历史
- 收稿日期:2014-01-08
- 网络出版日期:2014-09-25
2. 农业微生物技术教育部工程研究中心, 哈尔滨 150080
2. Engineering Research Center of Agricultural Microbiology Technology, Ministry of Education, Heilongjiang University, Harbin 150080, China
1935年,英国生态学家亚瑟·坦斯利(Arthur Tansley)提出生态系统概念[1]。随着分子生物学技术用于生物群落结构和丰度分析,以及高通量生物信息技术孕育的一系列系统生物学研究手段的迅猛发展,人们得以更加整体地了解生态系统的过程和功能。基因组学和转录组学考察遗传多样性和基因表达情况[2],蛋白质组学能够发现新基因,并监测代谢途径及重要蛋白对环境变化的响应[3]。但是,这些基于分子生物学原理的实验技术往往受到分子标记和检测技术的局限,难以在研究对象的基因型和表型、数量和功能间建立直接关联。
代谢物是生物信息传递的终端层次,代谢物组能直观反映生物体/细胞对所处环境的响应。1999年,伦敦帝国大学Nicholson教授发表生物系统对病原性刺激的代谢响应及基于核磁共振的多变量统计分析结果,首次将相关研究定义为代谢组学范畴[4]。代谢组学以组群指标分析为基础技术,以高通量检测和数据处理为手段,以质谱(MS)和核磁共振(NMR)技术为分析平台,以信息建模与系统整合为目标,已成为生态学研究的有力工具[5, 6, 7, 8]。近年来,代谢组学技术在土壤、海洋等传统生态学领域广泛应用,与其他系统生物学技术的结合应用也日益密切,如蛋白质组学、转录组学和基因组学等[9, 10]。代谢组学与生态学的互相融合及相关技术的交叉应用,孕育了一门新兴学科——生态代谢组学的建立与发展。本文将理清代谢组学与生态代谢组学学科发展的脉络,综述代谢组学研究中的常用技术的优势与局限性,论述代谢组学技术在生态学研究中的应用现状,展望代谢组学技术与其他系统生物学组学技术的结合应用前景,提出生态代谢组学研究者未来要完成的任务和面对的挑战。
1 生态代谢组学代谢组学概念metabonomics和metabolomics分别出现于20世纪90年代[4]和21世纪初[11],广义定义为对某一生物系统中产生的或已存在的代谢物组的研究。这两个术语描述的都是生物系统中代谢物组的定量测定过程,不同之处在于:metabonomics侧重病理性刺激及基因修饰带来的代谢响应,而metabolomics特指遗传变化或环境波动带来的代谢响应。这两个术语所涵盖的相关概念、研究方法(表 1)、分析技术和建模步骤基本相似,因此如无特殊说明,学术界对二者没有明确区分。
| 概念 Conception | 定义 Definition | ||
| 代谢物 Metabolite | 生化网络中具有生物活性的小分子物质 | ||
| 代谢物组 Metabolome | 生物系统(如一个细胞)中的所有代谢物 | ||
| 胞外代谢物组 Exo-metabolome | 细胞外(如培养物上清中)的所有代谢物 | ||
| 胞内代谢物组 Endo-metabolome | 细胞(或细胞器)内的所有代谢物 | ||
| 代谢猝灭 Metabolic quenching | (样品制备过程中)所有代谢活动的瞬间停止 | ||
| 目的物分析 Target analysis | 一个目的代谢物(如蛋白)的底物和/或产物的定量分析a | ||
| 代谢轮廓Metabolic profiling | 同类或特定路径中或相关的一系列代谢物(如糖类、脂类、有机酸类)的定量分析a | ||
| 代谢指纹 Metabolic fingerprinting | 胞内代谢物组的半定量分析b | ||
| 代谢足迹 Metabolic footprinting | 胞外代谢物组的半定量分析b | ||
| a: 定量分析指测定目的代谢物的绝对含量值,并给出基质效应误差的校正数据;b: 半定量分析由峰面积或峰高计算出的数据,数据误差源自样品本身或基质效应,数据可用于直接比较相似样品间目的代谢物含量的相对变化 | |||
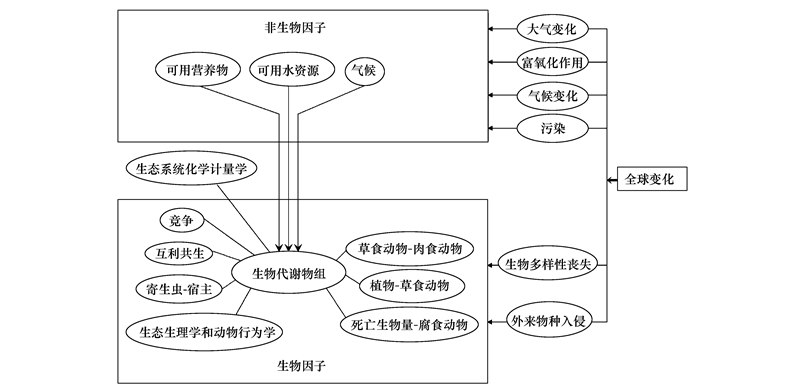
代谢组学的研究范畴是某一个生物体系中代谢物组水平的变化。在此基础上,2011年Sardans等人从生态学角度提出生态代谢组学(ecological metabolomics/ecometabolomics)概念,即研究某一个生物体对环境变化的代谢物组水平的响应(图 1)。如果研究要阐释的是生物体在某一个代谢路径上对环境变化的代谢物水平响应,则应称为局部生态代谢组研究(partial ecometabolomic studies,PEM)[17]。随着代谢组学与生态学分支学科交叉融合的日益深入,生态代谢组学研究已不局限于单一生物体代谢物组或某一代谢路径的生态学响应,而是外延到生态系统中各种生物及非生物因子的生态学过程与功能及复杂相互作用,包括生物对非生物因子如气候、营养匮乏、盐胁迫、低氧的生态代谢响应,两个或多个生物类群如植物与真菌、植物共生细菌与植物病毒、植物与动物间的相互作用等等。生态代谢组学的建立与发展,有助于深入认识物种间的相互作用机制与协同进化机制,加深对生物进化和生态系统的运作规律的理解。
2 生态学研究中应用的代谢组学主要技术本文将详细论述基于NMR和MS的两大技术平台在生态代谢组学研究中的应用现状,相关样品制备及数据处理内容请参阅Wu[18] 、Koek[19]和van Gulik[20]等人的报道。
2.1 核磁共振技术核磁共振技术的基本原理是:自旋的原子核在恒定外加磁场中做称为进动的回旋转动,当进动频率与外加电磁波频率相等时,二者产生的共振即称为核磁共振。此时,原子核吸收电磁波能量的吸收曲线即核磁共振谱(NMR-spectrum),可用于判断原子在分子中的位置与相对数量,进而对化合物进行定性与定量分析。1960年,Ohashi首次将磁共振技术用于微生物代谢研究-红曲霉((Monascus sp.)产红曲黄素(monascoflavin)及其还原产物[21]。2001年,Lefebvre等人在美国北卡罗莱纳州召开的代谢轮廓剑桥健康技术会议第一届年会(Proceedings of the 1st annual Cambridge Healthtech Conference on Metabolic Profiling,Chapel Hill,NC,USA)上报告了核磁共振氢谱技术用于大肠杆菌(Escherichia coli)代谢组学研究的成果,正式宣告核磁共振技术成为代谢组学研究的最主要技术平台。目前NMR技术在生物医学领域的代谢组学中应用最为广泛,如检测脑脊髓液、血液、尿液等体液代谢物组水平的变化用于疾病诊断,神经性疾病多发性硬化(Multiple Sclerosis,MScl)病例代谢物组中的生物标志物识别等等[22]。2013年,Rivas-Ubach等人建立并优化了用于生态代谢组学研究的NMR研究方法,包括样本间的差异识别、环境变化相关生物标志物的鉴定等等,不仅推动了NMR技术在环境样品检测中的应用,也将促进NMR技术在生态学研究中的系统化与标准化进程[23]。与其他分析技术相比,核磁共振技术具有样品前处理简单且无损伤、样品中多种成分测定同步进行的优点。
1H 100%的天然丰度赋予其在NMR谱图中的高度灵敏性。1H NMR谱图中,特征峰数量反映1H化学环境的种类,特征峰间的强度比则指示不同环境下1H的数目比。由于积分曲线高度与对应氢核数成正比,1H NMR既可对代谢物进行定量分析,也可用于结构定性。2005年,Rosenblum等人用1H NMR考察了加利福尼亚红螺(Haliotis rufescens)在食物、温度、原核生物感染等环境压力下,机体枯萎症(withering syndrome)与代谢物组水平响应间的关系,证明了1H NMR从代谢组学角度用于海洋物种病理学研究的可行性[24]。此后,1H NMR广泛应用于代谢失调、器官损伤和恶性肿瘤等疾病诊断和病理机理研究领域。Pearce 等人通过1H NMR证明了维生素B复合物相关代谢物响应水平与乳腺癌恶性程度间的耦合关系[25]。近年来,用于代谢组学研究的核磁共振技术有了飞跃式发展,如检测体液中L-左旋肉碱(L-carnitine)的高场单量子同调过滤核磁共振氢谱(high field single quantum coherence filtering 1H-NMR)[26],判定大肠癌癌变阶段的高分辨率魔角旋转核磁共振氢谱(high-resolution magic angle spinning 1H NMR,HR-MAS 1H NMR)[27]等。
2.1.2 核磁共振碳谱(13C NMR)13C 1.1%的极低天然丰度使其可以作为标记底物的示踪研究成为可能。与传统同位素示踪技术相比,13C作为稳定性同位素,具有无放射性危害、无环境污染和NMR检出灵敏等优点。13C NMR除了可提供代谢物碳核类型、碳分布、核间关系信息外,还能区分立体异构体的微细结构。此外,13C NMR能够弥补1H NMR不能测定不含氢官能团、无法区别含碳较多有机物中化学环境类似烷氢的不足。Kalaitzis等人将13C NMR与1H NMR联用,从海绵代谢物组中成功检测出环己二烯基环衍生的bromotyrosine型生物碱[28]。有研究者利用交叉极化魔角旋转核磁共振碳谱(cross-polarization magic angle spinning 13C NMR,CP-MAS 13C NMR)解析了植物有机代谢物组水平的响应与当地气候、土壤及植被间的关系[29]。
2.1.3 其他元素核磁共振波谱除了1H NMR 和13C NMR 外,其他元素的核磁共振波谱也可用于代谢组学研究。31P 天然丰度为100%,且磷元素与生物体能量代谢关系密切,因此31P NMR可用于含磷代谢物代谢途径及相关酶促反应动力学研究[30]。19F NMR 则可用于探究含氟污染物的微生物降解途径、含氟药物在人体内的作用及代谢机理[31]。此外,核磁共振技术中用于代谢组学研究的元素还有17O[32]、15N[33, 34]等。
2.2 质谱技术质谱是一种基于离子质核比测定的分析方法,因同时兼具灵敏性和特异性,目前已逐渐超越核磁共振技术,成为代谢组学研究中代谢物定性及定量分析的最有力工具,多位研究者已发表了质谱技术在代谢组学中应用的综述论文[35, 36]。近年来,质谱技术更是促进了生态代谢组学的革命性发展,覆盖领域从生态生理学、生态毒理学到化学生态学和生物地理学等生态学分支或相关学科。
2.2.1 气相色谱-质谱联用(GC-MS)GC-MS技术用于代谢组学研究始于本世纪初,尽管存在样品衍生带来定量偏差、温度等因素影响柱效带来信号损失、测定时间长等局限性,但由于GC-MS设备相对价格低,在化学及生物学研究中使用广泛,且用于化合物定性分析的数据库信息量丰富,它仍是生态代谢组学研究的最主要技术平台。GC-MS可检测三孢布拉氏霉菌(Blakeslea trispora) 代谢物组对胞外生化条件的响应,尤其是花生烯酸(arachidonic acid)对菌株产β-胡萝卜素(β-carotene)的影响[37]。Wallis等人的研究表明,美国黑松(Pinus contorta)针叶中木质素、可溶性酚类等次级代谢物的含量因当地环境条件即生态系统林地类型不同而显著变化,而与气候、地理因素不相关[38]。GC-MS为生物样本提供代谢表型图谱,通过与谱库比对鉴定代谢物,可用于探究代谢机理、识别生物标志物,在生态生理学、森林环境压力、生态毒理学和动植物间生态学关系领域有深入应用潜力[39]。
2.2.2 液相色谱-质谱联用(LC-MS)LC-MS与GC-MS相比的显著优势在于无需样品衍生化,因此可以分析的代谢物种类更多,同时灵敏度高于NMR。目前,LC-MS在微生物种属的化学分型鉴定[40]、特异性代谢物检测[41]、生物对极端环境的适应机制[42]等研究中都有应用。LC-MS技术的局限性首先在于任何一种柱材料都不能适合分离所有代谢物,其次是谱库数据量有限,将其与GC-MS结合使用,成为生态代谢组学者拓展研究深度的新选择。Doerfler 等人采用MetMAX校准工具和COVAIN统计工具同步分析了来自同一生物样品的GC-MS和LC-MS数据,通过格兰杰因果(Granger causality)检验,构建了基于GC-LC-MS的整合数据代谢模型,可用于识别初级代谢和次级代谢交界面的关键调节位点[43]。在Ciborowski等人对臭氧自血疗法(ozonated autohemotherapy)中机体血液代谢对臭氧的响应研究中,GC-MS检测到乳酸及丙酮酸含量增高,LC-MS则找出了臭氧压力下的七种生物标志物[44]。
2.2.3 直接注入式质谱(DIMS)DIMS分析中,极性代谢物样品以液体形式直接进入质谱离子源而无需色谱预分离。由于样品中的复杂代谢物分离仅基于质核比,DIMS依赖高分辨率的检测器,如TOF(time of flight)、轨道离子阱(orbitrap)和FT-ICR (Fourier transform ion cyclotron resonance)等。与需要与色谱联用的质谱技术相比,DIMS的劣势是离子抑制和质核比相同代谢物无法区分,但优势也显而易见:一是样品测定通量高,谱图相对简单;二是测定过程完全没有样品滞留现象。目前,DIMS在植物种群的自然变异[45]、生物体对环境压力及毒物的代谢指纹响应等生态代谢组学研究中都有应用[46, 47]。
2.2.4 质谱技术在生态代谢组学中的应用趋势首先,鉴于每种技术平台都有应用特异性和局限性,而环境代谢物组及其复杂,许多基于MS的技术平台在生态代谢组学中的应用有待开发,如毛细管电泳质谱(MS)、离子迁移质谱(MS)等。其次,非目的代谢组的检测耗时长、成本高,在生态代谢组学尤其是环境科学中的应用具有局限性。生态学家应针对特定种类代谢物或与环境改变相关的代谢物组分,开发用于目的代谢物组或半目的代谢物组的LC-MS和LC-MS/MS技术,建立用时更短、目的性更强的标准化检测方法,使质谱技术更广泛的用于环境监测、可控生态系统过程与功能研究、痕量化合物检测等常规生态学领域。
3 代谢组学技术在生态学研究中的应用代谢组学核心技术NMR和MS最早用于研究单个细胞或组织的代谢物组变化,进而推广到生物体乃至生物种群和群落对环境因子变化的代谢物组水平的响应。目前代谢组学技术与研究方法的应用不断拓展,如生化因子影响与生态学现象之间的关系、不同营养水平物种间的相互作用、生物群落中不同种群间的协同进化等等。代谢组学技术在环境监测、生态系统预警等应用生态学及物种进化、生态型鉴别等理论生态学研究中显示出巨大潜力。生态学涵盖范畴宽泛,分支众多,本文以生物组织层次和生物类群为划分依据,分述代谢组学技术在生态学研究中的应用。
3.1 按生物组织层次划分 3.1.1 个体生态学生物体对化学污染物的响应——陆地环境毒理学,是代谢组学技术在环境科学中应用最为广泛的领域,其中涉及最早的是蚯蚓和野生哺乳动物,其次是某些鱼类和无脊椎水生物种[48]。目前,代谢组学技术越来越多的用于监测生物体对环境变化的非病理性代谢响应,研究结果可作为相关生态系统破坏前的预警信号。例如,环境中的微量有机污染物能够破坏生物的生殖健康,传统血样检测方式只能检测已损伤生物体。Samuelsson等人以生活在合成避孕剂乙炔雌二醇(ethinyloestradiol,EE2)污染环境中的虹斑(Oncorhynchus mykiss)为研究对象,利用NMR技术找出了内分泌物代谢物组波动中的生物标志物,进而可以在鱼体生殖健康受损之前采取有效解毒措施[49]。代谢组学技术也可用于研究生物个体对气候、食物等非化学环境因子的响应。2009年,Lake等人报道了野生型拟南芥(Arabidopsis thaliana)对高强度紫外照射的代谢物组水平的适应性响应,但DIMS技术未能阐明适应机制[50]。有趣的是,研究者尝试利用代谢组学技术检测生物体与环境信号分子间的交流,这也属于化学生态学的研究范畴,如蚂蚁、蜜蜂等昆虫对来自食物或其天敌的特异性化合物的代谢物组水平的响应[51],植物感知周围草食动物或致病菌后代谢物组的变化等[52]。
3.1.2 种群生态学代谢组学研究方法中的代谢轮廓分析,使无显著表型差异的种属鉴别和分类成为可能。Bundy等人利用NMR技术成功的将几株致病性及生理特性均不相同的临床病源细菌菌株鉴别开来,值得注意的是,此前研究者尝试利用基因组学技术筛选编码已知致病毒素基因,但未能实现不同生态型的鉴别[53]。此外,代谢组学技术还可用于遗传修饰的植物种群与所处环境间的相互作用关系。如将代谢组学技术与基因组学技术结合使用,便可获得该种群对环境压力分子水平响应的全面信息,同时探明种群中沉默表型的生态学功能及农林业价值[54]。
3.1.3 群落生态学在群落生态学研究中,代谢组学技术最早用于分析土壤中小分子及大量大分子代谢物,进而了解微生物种群与植物凋落物组分间的关系[55]。虽然特异的代谢标志物可以用于指示捕食者与被捕食者的关系以及生产者与消费者间的能量流动,但是在完全开放的自然环境中寻找群落内部微小的代谢水平的变化仍是十分困难的。代谢组学技术能够实现时空连续变化环境样本中代谢物组的实时高通量分析,首先为相对简单的微生物群落生态学研究提供了便捷。Scullion 等人采用傅立叶变换红外光谱(fourier transform infrared spectrometry,FTIR spectrometry)技术,绘制了土壤蚓粪中微生物群落的代谢指纹图谱,开创了环境样品中微生物群落结构和生态学过程研究的先河[56]。此外,Gidman等人的报道称,短柄草属植物Brachypodium distachyion在竞争物种A. thaliana存在的环境下,代谢组分有显著变化,即代谢组学技术可从代谢物水平反映植物对复杂生物因素的响应[57]。
3.2 按所研究的生物类群划分 3.2.1 动物生态学代谢组学技术在野生动物和人工饲养如动物园和实验室动物的保护生态学研究中都有应用,主要包括代谢轨迹、种群健康和营养学三个方面[58]。英国伯明翰大学的Mark R. Viant教授是这一领域中的先行者。 2005年,Viant 等人提出代谢轨迹概念,应用在动物园中代谢异常动物的食谱构建和营养干预中[59]。2007年,Viant 提出NMOR (normal metabolic operating range)概念,即某一种群内95%以上个体代谢物组所构成的区间。虽然不同生境内的同一物种种群的代谢物组分存在差异,如动物园中与野生条件下非洲大羚羊的NMOR不同,这一概念仍十分有助于种群中非健康个体的检出以及指示种群功能的生物标志物的鉴别[60]。此外,动物营养学研究者则关注营养物中有哪些危及动物代谢健康的组分,以及如何从复杂的代谢系统中弄清真正的营养代谢过程[61, 62]。
3.2.2 植物生态学对于植物而言,代谢物不仅是基因表达、蛋白质相互作用以及各种生理调节过程的综合反映,代谢物也比mRNA和蛋白质与植物分子表型间的关联更加紧密。植物生态学家已将代谢组学技术成功用于研究植物对非生物环境因素的响应机制,并试图寻找特定环境压力与适应模式间的耦合机制。研究表明,糖、氨基酸和三羧酸循环中间代谢物等初级代谢物可作为光合作用功能障碍的标记物以及渗透调节的效应因子,用于(1)评估环境压力对植物代谢的影响;(2)追踪某种或某类化合物在特定生物合成/降解中的代谢路径;(3)植物样本的分类[63]。次级代谢物如抗氧化剂、活性氧(ROS)清除剂等防御性化合物和调节性代谢物,更具种属特异性,通常在特定压力条件下诱导合成,常与抗逆性和结实品质密切相关,可用于农、林业育种及种质改良研究[64, 65]。
3.2.3 微生物生态学在复杂的生态系统中,识别并定量分析微生物群落结构及其生态学功能并非易事。微生物多为单细胞存在,结构相对简单,代谢物组更能直观反映细胞对所处环境的响应。应用代谢组学技术实时收集生态系统中微生物代谢物组及非生物基质的动态数据,已经成为分析微生物菌群与环境相互作用机制的有效手段。Hong等报道牛奶中的乳杆菌属(Lactobacillus)和双歧杆菌属(Bifidobacteria)乳酸菌能够使患者血液中葡萄糖、酪氨酸和乳糖含量正常化,从而有效缓解肠易激综合症(IBS)[66]。Jung等以肠膜明串珠菌(Leuconostoc mesenterorides)作为发酵剂,利用代谢组学技术结合基因组学技术调查了泡菜发酵过程中乳酸菌群落及代谢物组的变化规律,为商品泡菜的品质均一化和益生功能性提高提供了指导[67]。
4 展望生态代谢组学概念的提出,表明人们已不局限于关注个别基因和蛋白质的分子生物学、个别物质的代谢路径,而是希望从更为宏观的视角,了解生态系统中物种之间、物种与环境之间各组分动态的结构、数量与功能。如何利用代谢组学技术实现这一愿景,生态学家仍面临诸多挑战。第一,代谢物组分数量庞大,难于在生物生态学功能与代谢响应改变之间建立关联。自然界的生命形式十分复杂,植物代谢物10万到20万种,即便是单细胞微生物,我们也无法准确给出代谢物种类的准确数目。此外,性别、年龄、健康状况、所处环境等因素变化都会导致生物体的代谢变异。已有科学家尝试将生物个体的代谢物变化及转化数据叠加分析,建立代谢物组响应的动态模型[68]。体内(in vivo) NMR、质子转移反应质谱、同位素标记以及处理高通量数据生物信息学的飞速发展,也将有助于人们理解生物体生态学功能与代谢响应间的关系[69]。第二,高通量的“组学”信息与生态学基础理论不对称。生态代谢组学研究者不应只专注于实验室的分析技术,而应该在外业研究中收集生物体生长、密度、繁殖和捕食等基本数据,这样才能结合高通量的“组学”信息,实现生物个体代谢网络和生态系统过程与功能的模型解释。第三,研究视野局限,目前的报道主要关注研究对象的生理/解剖特征与代谢物组变化间的关系。生态代谢组学概念在2011年刚被提出,代谢组学技术在生态学研究中应用尚处于起步阶段。未来的研究应充分利用代谢组学分析技术的灵敏性,以更加宏观的生态学视角设计试验,将温湿度、土壤性质等环境因素纳入研究范畴。
在未来的生态学研究实践中,代谢组学的应用将呈现以下几种发展趋势。首先是代谢组学与其他系统生物学组学技术紧密结合[42]。基因与蛋白质表达紧密相连,而代谢物则更多地反映细胞对营养状态、环境条件等外界因素变化的响应。因此有人说,“基因组学和蛋白质组学告诉你什么可能会发生,而代谢组学则告诉你什么确实发生了”。组学技术联用能够使人们从适应—选择—进化的水平深刻理解物种及其与生境间的相互作用。其次是多种代谢组学分析方法同时用于一个生物样品的分析,短时同步获得代谢物组的定性定量数据,如1H NMR和GC-MS[70],1H NMR和HPLC-MS[71],HPLC-DAD-MS-SPE-NMR (高效液相色谱-二极管阵列检测-质谱-固相萃取-核磁共振光谱)[72]等。最后,用于科学研究的开放式代谢物组数据库建立和扩容,将提高研究者对代谢物组数据的生物信息学处理和定性定量分析效率[73, 74]。随着代谢组学与其他系统生物学组学理论研究的交叉深入,以及以NMR和MS为主要技术平台的分析技术的联用发展,代谢组学技术将在压力响应、生活史变异、种群结构、营养互作、营养循环、生态位等生态学相关领域中有更加广泛和深入的应用。
| [1] | Tansley A G. The use and abuse of vegetational concepts and terms. Ecology, 1935, 16(3):284-307. |
| [2] | Gill S R, Pop M, DeBoy R T, Eckburg P B, Turnbaugh P J, Samuel B S, Gordon J I, Relman D A, Fraser-Liggett C M, Nelson K E. Metagenomic analysis of the human distal gut microbiome. Science, 2006, 312(5778):1355-1359. |
| [3] | Maron P A, Ranjard L, Mougel C, Lemanceau P. Metaproteomics:a new approach for studying functional microbial ecology. Microbial Ecology, 2007, 53(3):486-493. |
| [4] | Nicholson J K, Lindon J C, Holmes E. 'Metabonomics':understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data. Xenobiotica, 1999, 29(11):1181-1189. |
| [5] | Ram R J, VerBerkmoes N C, Thelen M P, Tyson G W, Baker B J, Blake R C, Shah M, Hettich R L, Banfield J F. Community proteomics of a natural microbial biofilm. Science, 2005, 308(5730):1915-1920. |
| [6] | Baker M. Metabolomics:from small molecules to big ideas. Nature Methods, 2011, 8(2):117-121. |
| [7] | Suthar M S, Brassil M M, Blahnik G, McMillan A, Ramos H J, Proll S C, Belisle S E, Katze M G, Gale Jr M. A systems biology approach reveals that tissue tropism to west nile virus is regulated by antiviral genes and innate immune cellular processes. PLoS Pathogens, 2013, 9(2):e1003168. |
| [8] | Geyer T. Modeling metabolic processes between molecular and systems biology. Current Opinion in Structural Biology, 2013, 23(2):218-223. |
| [9] | Mandal K, Singh B. Persistence of fipronil and its metabolites in sandy loam and clay loam soils under laboratory conditions. Chemosphere, 2013, 91(11):1596-1603. |
| [10] | Chen C, Wang J, Guo H, Hou W, Yang N, Ren B, Liu M, Dai H, Liu X, Song F, Zhang L. Three antimycobacterial metabolites identified from a marine-derived Streptomyces sp. MS100061. Applied Microbiology and Biotechnology, 2013, 97(9):3885-3892. |
| [11] | Fiehn O. Combining genomics, metabolome analysis, and biochemical modelling to understand metabolic networks. Comparative and Functional Genomics, 2001, 2(3):155-168. |
| [12] | Fiehn O. Metabolomics-the link between genotypes and phenotypes. Plant Molecular Biology, 2002, 48(1/2):155-171. |
| [13] | Allen J, Davey H M, Broadhurst D, Heald J K, Rowland J J, Oliver S G, Kell D B. High-throughput classification of yeast mutants for functional genomics using metabolic footprinting. Nature Biotechnology, 2003, 21(6):692-696. |
| [14] | Nielsen J, Oliver S. The next wave in metabolome analysis. Trends in Biotechnology, 2005, 23(11):544-546. |
| [15] | Villas-Boas S G, Højer-Pedersen J, Åkesson M, Smedsgaard J, Nielsen J. Global metabolite analysis of yeast:evaluation of sample preparation methods. Yeast, 2005, 22(14):1155-1169. |
| [16] | Villas-Boas S G, Mas S, Åkesson M, Smedsgaard J, Nielsen J. Mass spectrometry in metabolome analysis. Mass Spectrometry Reviews, 2005, 24(5):613-646. |
| [17] | Sardans J, Peñuelas J, Rivas-Ubach A. Ecological metabolomics:overview of current developments and future challenges. Chemoecology, 2011, 21(4):191-225. |
| [18] | Wu H F, Southam A D, Hines A, Viant M R. High-throughput tissue extraction protocol for NMR-and MS-based metabolomics. Analytical Biochemistry, 2008, 372(2):204-212. |
| [19] | Koek M M, Jellema R H, van der Greef J, Tas A C, Hankemeier T. Quantitative metabolomics based on gas chromatography mass spectrometry:status and perspectives. Metabolomics, 2011, 7(3):307-328. |
| [20] | van Gulik W M, Canelas A B, Seifar R M, Heijnen J J. The sampling and sample preparation problem in microbial metabolomics//Metabolomics in Practice. Weinheim, Germany:Wiley-VCH Verlag GmbH & Co. KGaA, 2013:1-19. |
| [21] | Ohashi M, Terahara A, Nakanishi K. NMR spectra of the metabolites of Monascus. Ⅱ. Monascorubrin. Bulletin of the Chemical Society of Japan, 1960, 33(9):1311-1312. |
| [22] | Smolinska A, Blanchet L, Buydens L, Wijmenga S S. NMR and pattern recognition methods in metabolomics:from data acquisition to biomarker discovery:a review. Analytica Chimica Acta, 2012, 75(31):82-97. |
| [23] | Rivas-Ubach A, Perez-Trujillo M, Sardans J, Gargallo-Garriga A, Parella T, Penuelas J. Ecometabolomics:optimized NMR-based method. Methods in Ecology and Evolotion, 2013, 4(5):464-473. |
| [24] | Rosenblum E, Viant M, Braid B, Moore J, Friedman C, Tjeerdema R. Characterizing the metabolic actions of natural stresses in the California red abalone, Haliotis rufescens using 1H NMR metabolomics. Metabolomics, 2005, 1(2):199-209. |
| [25] | Pearce J M, Mahoney M C, Lee J H, Chu W J, Cecil K M, Strakowski S M, Komoroski R A. 1H NMR analysis of choline metabolites in fine-needle-aspirate biopsies of breast cancer. Magnetic Resonance Materials in Physics, Biology and Medicine, 2013, 26(3):337-343. |
| [26] | Tsiafoulis C G, Exarchou V, Tziova P P, Bairaktari E, Gerothanassis I P, Troganis A N. A new method for the determination of free l-carnitine in serum samples based on high field single quantum coherence filtering 1H-NMR spectroscopy. Analytical and Bioanalytical Chemistry, 2011, 399(6):2285-2294. |
| [27] | Jiménez B, Mirnezami R, Kinross J, Cloarec O, Keun H C, Holmes E, Goldin R D, Ziprin P, Darzi A, Nicholson J K. 1H HR-MAS NMR spectroscopy of tumor-induced local metabolic "field-effects" enables colorectal cancer staging and prognostication. Journal of Proteome Research, 2013, 12(2):959-968. |
| [28] | Kalaitzis J A, Davis R A, Quinn R J. Unequivocal 13C NMR assignment of cyclohexadienyl rings in bromotyrosine-derived metabolites from marine sponges. Magnetic Resonance in Chemistry, 2012, 50(11):749-754. |
| [29] | Faz Cano A, Mermut A R, Ortiz R, Benke M B, Chatson B. 13C CP/MAS-NMR spectra of organic matter as influenced by vegetation, climate and soil characteristics in soils from Murcia, Spain. Canadian Journal of Soil Science, 2002, 82(4):403-411. |
| [30] | Wary C, Naulet T, Thibaud J L, Monnet A, Blot S, Carlier P G. Splitting of Pi and other 31P NMR anomalies of skeletal muscle metabolites in canine muscular dystrophy. NMR in Biomedicine, 2012, 25(10):1160-1169. |
| [31] | Bapiro T E, Richards F M, Goldgraben M A, Olive K P, Madhu B, Frese K K, Cook N, Jacobetz M A, Smith D M, Tuveson D A. A novel method for quantification of gemcitabine and its metabolites 2', 2'-difluorodeoxyuridine and gemcitabine triphosphate in tumour tissue by LC-MS/MS:comparison with 19F NMR spectroscopy. Cancer Chemotherapy and Pharmacology, 2011, 68(5):1243-1253. |
| [32] | Zhu X H, Zhang Y, Tian R X, Lei H, Zhang N Y, Zhang X L, Merkle H, Ugurbil K, Chen W. Development of 17O NMR approach for fast imaging of cerebral metabolic rate of oxygen in rat brain at high field. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(20):13194-13199. |
| [33] | Sarkar R, Comment A, Vasos P R, Jannin S, Gruetter R, Bodenhausen G, Hall H, Kirik D, Denisov V P. Proton NMR of 15N-choline metabolites enhanced by dynamic nuclear polarization. Journal of the American Chemical Society, 2009, 131(44):16014-16015. |
| [34] | Tayyari F, Gowda G N, Gu H, Raftery D. 15N-cholamine a smart isotope tag for combining NMR-and MS-Based metabolite profiling. Analytical Chemistry, 2013, 85(18):8715-8721. |
| [35] | Dunn W B, Ellis D I. Metabolomics:current analytical platforms and methodologies. TrAc Trends in Analytical Chemistry, 2005, 24(4):285-294. |
| [36] | Griffiths W J, Wang Y. Mass spectrometry:from proteomics to metabolomics and lipidomics. Chemical Society Reviews, 2009, 38(7):1882-1896. |
| [37] | Hu X, Li H, Tang P, Sun J, Yuan Q, Li C. GC-MS-based metabolomics study of the responses to arachidonic acid in Blakeslea trispora. Fungal Genetics and Biology, 2013, 57:33-41. |
| [38] | Wallis C M, Huber D P, Lewis K J. Ecosystem, location, and climate effects on foliar secondary metabolites of lodgepole pine populations from Central British Columbia. Journal of Chemical Ecology, 2011, 37(6):607-621. |
| [39] | Viant M R, Sommer U. Mass spectrometry based environmental metabolomics:a primer and review. Metabolomics, 2013, 9(1):144-158. |
| [40] | Lee J S, Kim Y S, Park S, Kim J, Kang S J, Lee M H, Ryu S, Choi J M, Oh T K, Yoon J H. Exceptional production of both prodigiosin and cycloprodigiosin as major metabolic constituents by a novel marine bacterium, Zooshikella rubidus S1-1. Applied and Environmental Microbiology, 2011, 77(14):4967-4973. |
| [41] | Southam A D, Lange A, Hines A, Hill E M, Katsu Y, Iguchi T, Tyler C R, Viant M R. Metabolomics reveals target and off-target toxicities of a model organophosphate pesticide to roach (Rutilus rutilus):Implications for biomonitoring. Environmental Science & Technology, 2011, 45(8):3759-3767. |
| [42] | Trauger S A, Kalisak E, Kalisiak J, Morita H, Weinberg M V, Menon A L, Poole Ii F L, Adams M W, Siuzdak G. Correlating the transcriptome, proteome, and metabolome in the environmental adaptation of a hyperthermophile. Journal of Proteome Research, 2008, 7(3):1027-1035. |
| [43] | Doerfler H, Lyon D, Nägele T, Sun X L, Fragner L, Hadacek F, Egelhofer V, Weckwerth W. Granger causality in integrated GC-MS and LC-MS metabolomics data reveals the interface of primary and secondary metabolism. Metabolomics, 2013, 9(3):564-574. |
| [44] | Ciborowski M, Lipska A, Godzien J, Ferrarini A, Korsak J, Radziwon P, Tomasiak M, Barbas C. Combination of LC-MS-and GC-MS-based metabolomics to study the effect of ozonated autohemotherapy on human blood. Journal of Proteome Research, 2012, 11(12):6231-6241. |
| [45] | Davey M P, Woodward F I, Quick W P. Intraspecfic variation in cold-temperature metabolic phenotypes of Arabidopsis lyrata ssp. petraea. Metabolomics, 2009, 5(1):138-149. |
| [46] | Brito-Echeverría J, Lucio M, López-López A, Antón J, Schmitt-Kopplin P, Rosselló-Móra R. Response to adverse conditions in two strains of the extremely halophilic species Salinibacter ruber. Extremophiles, 2011, 15(3):379-389. |
| [47] | Poynton H C, Taylor N S, Hicks J, Colson K, Chan S, Clark C, Scanlan L, Loguinov A V, Vulpe C, Viant M R. Metabolomics of microliter hemolymph samples enables an improved understanding of the combined metabolic and transcriptional responses of Daphnia magna to cadmium. Environmental Science & Technology, 2011, 45(8):3710-3717. |
| [48] | Vandenbrouck T, Jones O A H, Dom N, Griffin J L, De Coen W. Mixtures of similarly acting compounds in Daphnia magna from gene to metabolite and beyond. Environment International, 2010, 36(3):254-268. |
| [49] | Samuelsson L M, Förlin L, Karlsson G, Adolfsson-Erici M, Larsson D G J. Using NMR metabolomics to identify responses of an environmental estrogen in blood plasma of fish. Aquatic Toxicology, 2006, 78(4):341-349. |
| [50] | Lake J A, Field K J, Davey M P, Beerling D J, Lomax B H. Metabolomic and physiological responses reveal multi-phasic acclimation of Arabidopsis thaliana to chronic UV radiation. Plant, Cell & Environment, 2009, 32(10):1377-1389. |
| [51] | Tittiger C. Functional genomics and insect chemical ecology. Journal of Chemical Ecology, 2004, 30(12):2335-2358. |
| [52] | Jones O A H, Maguire M L, Griffin J L, Jung Y H, Shibato J, Rakwal R, Agrawal G K, Jwa N S. Using metabolic profiling to assess plant-pathogen interactions:an example using rice (Oryza sativa) and the blast pathogen Magnaporthe grisea. European Journal of Plant Pathology, 2011, 129(4):539-554. |
| [53] | Bundy J G, Willey T L, Castell R S, Ellar D J, Brindle K M. Discrimination of pathogenic clinical isolates and laboratory strains of Bacillus cereus by NMR-based metabolomic profiling. FEMS Microbiology Letters, 2005, 242(1):127-136. |
| [54] | Rischer H, Oksman-Caldentey K-M. Unintended effects in genetically modified crops:revealed by metabolomics? Trends in Biotechnology, 2006, 24(3):102-104. |
| [55] | Portais J C, Delort A M. Carbohydrate cycling in micro-organisms:what can 13C-NMR tell us? FEMS Microbiology Reviews, 2002, 26(4):375-402. |
| [56] | Scullion J, Elliott G N, Huang W E, Goodacre R, Worgan H, Darby R M, Bailey M J, Gwynn-Jones D, Griffith G W, Winson M K, Williams P A, Clegg C D, Draper J. Use of earthworm casts to validate FT-IR spectroscopy as a 'sentinel'technology for high-throughput monitoring of global changes in microbial ecology:The 7th international symposium on earthworm ecology·Cardiff·Wales·2002. Pedobiologia, 2003, 47(5):440-446. |
| [57] | Gidman E, Goodacre R, Emmett B, Smith A R, Gwynn-Jones D. Investigating plant-plant interference by metabolic fingerprinting. Phytochemistry, 2003, 63(6):705-710. |
| [58] | Dove A D. Metabolomics has great potential for clinical and nutritional care and research with exotic animals. Zoo Biology, 2013, 32(3):246-250. |
| [59] | Viant M R, Bundy J G, Pincetich C A, de Ropp J S, Tjeerdema R S. NMR-derived developmental metabolic trajectories:an approach for visualizing the toxic actions of trichloroethylene during embryogenesis. Metabolomics, 2005, 1(2):149-158. |
| [60] | Viant M R. Metabolomics of aquatic organisms:the new 'omics' on the block. Marine Ecology Progress Series, 2007, 332:301-306. |
| [61] | Coen M, Holmes E, Lindon J C, Nicholson J K. NMR-based metabolic profiling and metabonomic approaches to problems in molecular toxicology. Chemical Research in Toxicology, 2008, 21(1):9-27. |
| [62] | Gibney M J, Walsh M, Brennan L, Roche H M, German B, van Ommen B. Metabolomics in human nutrition:opportunities and challenges. The American Journal of Clinical Nutrition, 2005, 82(3):497-503. |
| [63] | Saito K, Matsuda F. Metabolomics for functional genomics, systems biology, and biotechnology. Annual Review of Plant Biology, 2010, 61:463-489. |
| [64] | Lisec J, Römisch-Margl L, Nikoloski Z, Piepho H P, Giavalisco P, Selbig J, Gierl A, Willmitzer L. Corn hybrids display lower metabolite variability and complex metabolite inheritance patterns. The Plant Journal, 2011, 68(2):326-336. |
| [65] | Schauer N, Semel Y, Roessner U, Gur A, Balbo I, Carrari F, Pleban T, Perez-Melis A, Bruedigam C, Kopka J, Willmitzer L, Zamir D, Fernie A R. Comprehensive metabolic profiling and phenotyping of interspecific introgression lines for tomato improvement. Nature Biotechnology, 2006, 24(4):447-454. |
| [66] | Hong Y S, Hong K S, Park M H, Ahn Y T, Lee J H, Huh C S, Lee J, Kim I K, Hwang G S, Kim J S. Metabonomic understanding of probiotic effects in humans with irritable bowel syndrome. Journal of Clinical Gastroenterology, 2011, 45(5):415-425. |
| [67] | Jung J Y, Lee S H, Lee H J, Seo H Y, Park W S, Jeon C O. Effects of Leuconostoc mesenteroides starter cultures on microbial communities and metabolites during kimchi fermentation. International Journal of Food Microbiol, 2012, 153(3):378-387. |
| [68] | Peñuelas J, Sardans J. Ecology:elementary factors. Nature, 2009, 460(7257):803-804. |
| [69] | Gehlenborg N, O'Donoghue S I, Baliga N S, Goesmann A, Hibbs M A, Kitano H, Kohlbacher O, Neuweger H, Schneider R, Tenenbaum D. Visualization of omics data for systems biology. Nature Methods, 2010, 7:S56-S68. |
| [70] | Srinivasan J, Kaplan F, Ajredini R, Zachariah C, Alborn H T, Teal P E, Malik R U, Edison A S, Sternberg P W, Schroeder F C. A blend of small molecules regulates both mating and development in Caenorhabditis elegans. Nature, 2008, 454(7208):1115-1118. |
| [71] | López-Gresa M P, Maltese F, Bellés J M, Conejero V, Kim H K, Choi Y H, Verpoorte R. Metabolic response of tomato leaves upon different plant-pathogen interactions. Phytochemical Analysis, 2010, 21(1):89-94. |
| [72] | Schlotterbeck G, Ceccarelli S M. LC-SPE-NMR-MS:a total analysis system for bioanalysis. Bioanalysis, 2009, 1(3):549-559. |
| [73] | Hall R D. Plant metabolomics:from holistic hope, to hype, to hot topic. New Phytologist, 2006, 169(3):453-468. |
| [74] | Allwood J W, Ellis D I, Goodacre R. Metabolomic technologies and their application to the study of plants and plant-host interactions. Physiologia Plantarum, 2008, 132(2):117-135. |
 2015, Vol. 35
2015, Vol. 35