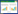文章信息
- 郭建国, 赵龙浩, 徐丹, 孙野青
- GUO Jianguo, ZHAO Longhao, XU Dan, SUN Yeqing
- 洪水对大凌河河流浮游细菌群落的影响
- Effect of the flood on the bacterial community of bacteriaplankton in Daling River
- 生态学报, 2015, 35(14): 4777-4783
- Acta Ecologica Sinica, 2015, 35(14): 4777-4783
- http://dx.doi.org/10.5846/stxb201312293053
-
文章历史
- 收稿日期:2013-12-29
- 修订日期:2014-09-09
洪水是由于气候的原因导致局部地区大雨、暴雨或大范围较长时间的霪雨天气所致,它是人类生产生活以及社会经济发展的威胁之一。洪水不仅造成大量房屋倒塌和农田被淹,危害到人民生命财产的安全,还对生态环境造成巨大的影响[1]。洪水可以造成严重的水土流失;可以携带大量泥沙、农药残留物以及工业废渣等大量污染物进入河流[2],使河道污染物总量增加;可以影响动植物栖息地的环境,而且影响动植物的生存和繁衍[3];可以冲洗盐碱化土壤表面的盐碱,补充沼泽和湖泊的水量,改进植物的生长状态和生物多样性条件[1, 4]。
微生物是地球上进行物质循环的生物地球化学过程的主力军,微生物群落的构成直接影响它们的功能[5, 6]。洪水作为一种水流运动的突发状况,对河流是一种干扰,这种干扰是否会重塑河流中的微生物群落的组成?如果这种干扰可以改变微生物群落的结构,这种驱动因素是什么?2012年8月,受第10号台风"达维"影响,辽宁省发生强降雨过程,发生了历史罕见的洪灾。大凌河是受影响严重的河流,本研究选取的研究区域位于锦州河口段,覆盖约80km,河流宽度在2—30m,深度在0.5—3m。收集了洪水暴发前后大凌河表层水样,研究洪水干扰对河流浮游细菌群落的影响。
1 材料与方法
|
| 图1 采样点分布图 Fig.1 Distribution of sampling sites in Daling River |
本研究的样品于2012年5月和2012年8月采自辽宁省锦州段大凌河流域表层水,具体布点如图 1,本研究沿河流共设17个位点,采样点多数位于郊区,由于当地各种改造工程的进行,有些位点只采集到1次,可参见表 1。水样经300目的无菌纱布过滤除去大型颗粒和杂质,取 300ml,用0.22um的硝酸纤维素膜过滤收集微生物于滤膜上,滤膜在-80℃冷冻保存。
1.2 实验方法 1.2.1 水样理化因素的测定溶解氧(DO)由溶氧仪雷磁GTB-607A测定,pH值由pH计雷磁THB-1测定,盐度S由盐度计测定,总氮量、氨氮含量、总磷量、磷酸盐的测定参照《水质分析方法国家标准汇编》中提供的方法进行。
1.2.2 DNA的提取将水样滤膜从-80℃冰箱中取出,将滤纸剪碎后,利用CTAB法提取DNA[7]。利用Nanodrop对DNA的纯度进行检测。
1.2.3 变性梯度凝胶电泳(DGGE)DNA的浓度进行定量后,以提取的水样微生物DNA为模板,用引物对U968-GC-F(5′CGCCCGGGGCGCG-CCCCGGGCGGGGCGGGGGCAGGGGGGAACGCGAAGAACCTTAC3′)和L1401-R(5′CGGTGTGTACAAGACC-C3′)扩增16SrDNA的V6-V8区,扩增程序如下:94℃ 5min,(94℃ 30s,55℃ 30s,72℃ 1min)30 cycles,72℃ 10min,4℃保存。PCR产物经琼脂糖凝胶电泳,利用灰度分析将上样量调整到400 ng,进行变性梯度凝胶电泳。聚丙烯酰胺凝胶的浓度为8%,变性浓度为45%—65%。电泳程序:120V 17h,60℃保温。电泳结束后,进行硝酸银染色,然后利用GS-800光密度扫描仪成像。
1.3 数据分析DGGE图谱利用Quantity one对主要条带进行灰度分析,导出相应的灰度值矩阵。灰度值进行对数转化后利用Canoco4.5对矩阵数据进行PCA分析,导出多样性数据进行分析;结合环境监测数据进行RDA分析,分析结果进行蒙特卡罗检验,置换次数为999,可以显著解释响应变量的环境变量被选入模型。两次采集水样间环境因素经SPSS中单因素ANOVA进行均值的显著性分析。
2 结果将样品根据位点的特征分为5个区域:R0-R4为河流的上游段;R5为工业排污口;R6,R7为河流下游段;R8,R9,R10为潮汐性淡水海水混合区;R10-R16为长期海水影响区,该区域在最小潮时仍然发生淡水海水的混合。
所有水样16SrDNA扩增产物经变性梯度凝胶电泳后得到指纹图谱,如图 2,条带分离清晰,样品间的主要微生物不同,河流中的浮游细菌群落结构存在差异。

|
| 图2 两次采集水样的微生物16SrDNA的DGGE指纹图 Fig.2 The DGGE fingerprints of microbial 16SrDNA in the samples collected in May 2012 and August 2012 |
为了进一步理解这种差异,对2012年5月和2012年8月的样品微生物群落组成进行了主成分分析(图 3)。2012年5月洪水发生前,大凌河表层水样按照样品采样点的环境特征分开,环境特征相似的样品的微生物群落结构相似(图 3);2012年8月洪水发生后,除工业排污口与其他位点的微生物群落结构有巨大差异外,其余大凌河表层水样散在分布(图 3)。以上研究表明洪水的发生改变了水流中的微生物群落结构。

|
| 图3 两次采集水样微生物群落的PCA分析 Fig.3 The PCA analysis of the microbial community in the samples |

|
|
图4
两次采集水样细菌群落的多样性分析
Fig.4 The index of diversity analysis of the bacterial community in the samples |

|
| 图5 两次采集水样理化因素条形图 Fig.5 The bar graph of environmental variables |
对5个区域中的微生物群落多样性进行了统计分析,表明在洪水发生前大凌河河流表层水中的微生物群落的香农指数H′有增加的趋势,特别是在海水影响区,洪水发生后大凌河河流表层水中的微生物群落的多样性指数有降低的趋势,海水影响区在洪水发生前后多样性指数差异显著(P<0.05)(图 4)。对河流表层细菌群落的多样性指数进行分析,洪水发生后微生物群落的多样性指数均值由3.04降到2.84,变异系数由0.06升到0.11。
对各水样的溶氧、pH值、盐度、氨氮、总氮、磷酸盐、总磷进行了测定(表 1)。对两次样品的环境变量进行均值比较,统计学分析表明:pH、S存在极显著性差异(P<0.01),TP存在显著性差异(P<0.05),其他因素存在差异,但不显著(图 5)。可见洪水的发生对于水流环境有很大的影响。
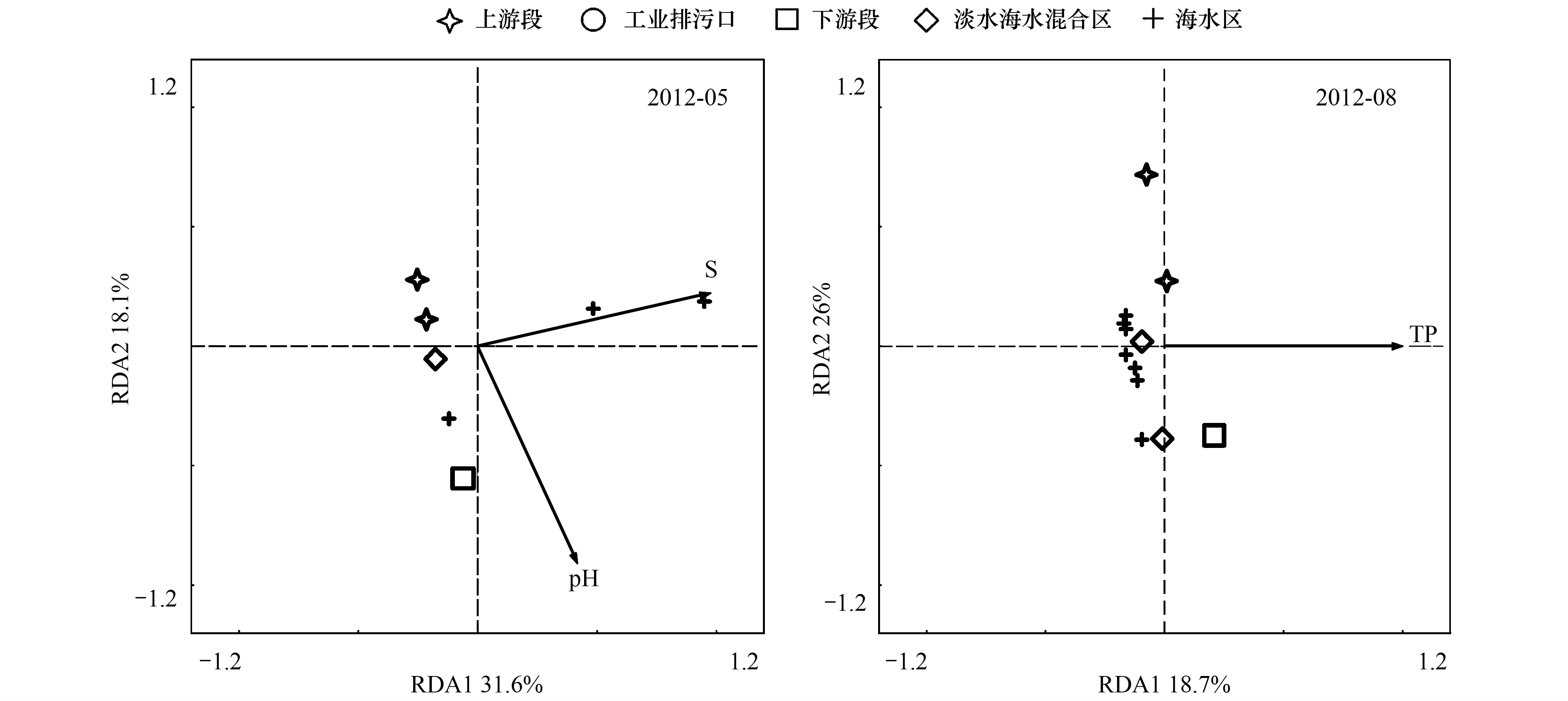
上述数据表明洪水发生前后,水环境发生了剧烈改变,而其中的微生物群落结构也发生了变化,那么水样中哪些环境变量能够解释微生物群落的结构?本文利用RDA分析对这一问题进行了进一步研究。在洪水发生前,水流中微生物群落的结构主要受pH和S的影响,在洪水发生后,水流中的微生物群落主要受TP的影响(图 6)。
| 时间 Time | 位点 Site | 溶解氧 Dissolved oxygen (DO) | pH | 盐度 Salinity (S) | 氨氮/(mg/L) NH4-N | 总氮/(mg/L) Total nitrogen (TN) | 磷酸盐/(mg/L) PO3-P | 总磷/(mg/L) Total phosphorus (TP) |
| 2012-05 | R0 | - | 8.3 | 3 | 0.87±0.031 | 2.733±0.115 | 0.67±0.036 | 0.83±0.083 |
| R2 | - | 8.1 | 3 | 0.42±0.02 | 1.97±0.058 | 0.38±0.049 | 0.53±0.056 | |
| R5 | - | 7.5 | 3 | 3.45±0.1 | 3.93±0.306 | 0.67±0.036 | 0.73±0.056 | |
| R6 | - | 9.1 | 3 | 0.32±0.035 | 2.83±0.757 | 0.39±0.051 | 0.84±0.085 | |
| R7 | - | 9.1 | 3 | 0.11±0.012 | 2.1±0.2 | 0.63±0.045 | 0.83±0.055 | |
| R8 | - | 8.5 | 3 | 0.23±0.012 | 1.0±0.1 | 0.33±0.035 | 0.37±0.01 | |
| R10 | - | 8.5 | 3 | 0.26±0.035 | 2.37±0.252 | 0.28±0.023 | 0.36±0.072 | |
| R11 | - | 8.8 | 3 | 0.86±0.035 | 2.47±0.115 | 1.25±0.081 | 1.33±0.05 | |
| R12 | - | 8.8 | 8 | 0.12±0.02 | 1.67±0.058 | 0.45±0.051 | 0.74±0.029 | |
| R13 | - | 8.6 | 6 | 0.1±0.001 | 1.5±0.265 | 0.66±0.079 | 1.08±0.1 | |
| 2012-08 | R3 | 6.77 | 7.63 | 0.32 | 1.12±0.113 | - | 0.04±0.011 | 0.41±0.035 |
| R4 | 1.92 | 7.71 | 0.3 | 1.31±0.06 | 2.17±0.181 | 0.03±0.002 | 0.5±0.08 | |
| R5 | 2.35 | 6.76 | 1.23 | 5.86±0.096 | 3.20±0.-2 | 0.09±0.015 | 1.49±0.09 | |
| R6 | 2.04 | 7.62 | 0.3 | 1.56±0.076 | - | 0.05±0.004 | 0.71±0.061 | |
| R8 | 7.48 | 8.28 | 0.92 | 1.2±0.142 | 1.53±0.-042 | 0.03±0.003 | 0.39±0.015 | |
| R9 | 4.21 | 7.7 | 0.36 | 1.19±0.087 | 2.83±0.-163 | 0.03±0.006 | 0.48±0.061 | |
| R10 | 3.84 | 7.7 | 0.45 | 1.13±0.046 | 1.70±0.-182 | 0.02±0.008 | 0.37±0.066 | |
| R11 | 4 | 7.75 | 0.44 | 1.34±0.093 | 1.40±0.-053 | 0.03±0.009 | 0.39±0.074 | |
| R12 | 4.18 | 7.79 | 0.4 | 1.23±0.128 | - | 0.02±0.003 | 0.32±0.044 | |
| R13 | 5.11 | 7.69 | 2.53 | 1.54±0.106 | 1.33±0.-115 | 0.03±0.003 | 0.31±0.036 | |
| R14 | 5.46 | 7.65 | 7.26 | 1.34±0.092 | - | 0.03±0.005 | 0.32±0.032 | |
| R15 | 5.77 | 7.72 | 5.78 | 1.26±0.078 | - | 0.02±0.004 | 0.32±0.026 | |
| R16 | 5.34 | 7.7 | 0.67 | 1.27±0.085 | - | 0.03±0.003 | 0.36±0.072 |
|
| 图6 两次采集水样微生物群落和环境变量的RDA分析 Fig.6 The RDA analysis between the microbial community and the environmental variables |
微生物广泛地存在于各种环境中,在生物地球化学过程中发挥着重要作用。微生物时刻受到各种自然压力的选择[8, 9]。洪水作为一种剧烈的自然干扰对农业,畜牧业[10]及生态环境有很大的影响。NATHAN R. DE JAGER[11]发现随着洪水的发生频率和时间的增长,小树种减少,耐受性物种规模增大。Chih-Ching Chung[12]发现长江洪灾影响聚球藻属在中国东海表层水中的生态分布。这些提示洪水可以对生态环境形成选择压力。
洪水发生前,PCA分析显示水样依据水流环境聚集在一起,分别为河流上游、排污口、河流下游、淡水海水混合区、海水影响区五类。有研究发现微生物群落的类型具有栖息地选择性[13, 14]。在洪水发生前,表层水中的微生物群落结构非常符合这一现象,也说明河流表层微生物群落在各自的栖息环境中进行进化,形成了环境依赖的特征。对这5个区域的微生物多样性的趋势分析也表明:洪水改变了微生物群落多样性沿河流的升高的趋势,与环境因素的改变和洪水的稀释作用有关。RDA分析洪水发生前的环境因素对于表层水中微生物群落结构的解释作用,发现pH和S是主要的环境解释因素。pH和S是已研究的环境因素中对生物群落的形成具有非常重要的因子[15, 16, 17]。已有研究表明:土壤pH对湿地植物的分布具有很大的影响[18],土壤微生物群落对pH变化敏感[19],海洋的酸化过程会影响海洋微生物的多样性[20]。在水生态系统中,盐度可以控制微生物的分布[21, 22],形成明显的淡水微生物群和海水微生物群。洪水发生前水流中微生物的解释变量为pH和S,也进一步说明该河段表层水中微生物群落形成了环境依赖性的结构。
洪水发生后,河道水位急速上升,水量增大,环境数据显示水流中的pH和S都明显下降,这与洪水的稀释作用有关,氨氮、总氮、磷酸盐、总磷的浓度也发生变化。对河流表层微生物群落的PCA分析表明:微生物群落在洪水后各样点根据河道自然环境特征聚集的特点消失,而是趋于散在分布。洪水流经农田,道路等将非土著性微生物带入河道,而河流的稀释导致原环境中微生物浓度的稀释,在这种条件下微生物在改变的新环境中迅速建立起新的群落结构,这也就解释了散在性分布的原因。从RDA分析的结果中发现,洪水后,影响微生物群落结构的主要解释变量为TP。而河流表层水中的TP在洪水发生前后有显著性差异。磷是浮游植物和细菌生长的必需元素,磷的缺失可以限制他们的生长[23]。磷是农田的主要肥料之一,突发洪水将大量农田中的磷元素带入河流,从而促进了微生物群落的重塑。磷的富集是导致淡水和近海岸赤潮的发生的原因之一[24, 25]。在一些富营养区海湾,磷是具有限制性的元素[26],突发洪水可能将大量磷元素带入,提高了风险。
结论:在自然条件下,河流表层水中的浮游细菌群落在各自的环境条件下进行进化,这种进化导致的结果与所处生态位的环境特征相关,当强烈的外界因素如洪水介入后,会改变河流固有的环境特征,特别是限制性元素磷的重新分布,从而对微生物群落造成重塑,重塑导致的微生物群落变化,会带来未知的危害,如赤潮等灾害的发生,本研究可以为淡水和近海岸灾害的发生提供依据。
| [1] | 许士国, 党连文, 牟志录. 嫩江1998年特大洪水环境影响分析. 大连理工大学学报, 2003, 43(1): 114-118. |
| [2] | Wölz J, Brack W, Moehlenkamp C, Claus E, Braunbeck T, Hollert H. Effect-directed analysis of Ah receptor-mediated activities caused by PAHs in suspended particulate matter sampled in flood events. Science of the Total Environment, 2010, 408(16): 3327-3333. |
| [3] | 高宗强. 洪水对生态环境的影响. 山西水土保持科技, 2002, (2): 25-25, 26-26. |
| [4] | Tan H Z, Luo Y J, Wen S W, Liu A Z, Li S Q, Yang T B, Sun Z Q. The effect of a disastrous flood on the quality of life in Dongting Lake area in China. Asia Pacific Journal of Public Health, 2004, 16(2): 126-132. |
| [5] | Stahl D A, Flowers J J, Hullar M, Davidson S. Structure and function of microbial communities//The Prokaryotes. Berlin Heidelberg: Springer, 2013: 3-30. |
| [6] | Nannipieri P, Ascher J, Ceccherini M T, Landi L, Pietramellara G, Renella G. Microbial diversity and soil functions. European Journal of Soil Science, 2003, 54(4): 655-670. |
| [7] | Sambrook J, Russell D W. Molecular Cloning: A Laboratory Manual. 3rd ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2001:512-513. |
| [8] | Dornelas M. Disturbance and change in biodiversity. Philosophical Transactions of the Royal Society B: Biological Sciences, 2010, 365(1558): 3719-3727. |
| [7] | Sambrook J, Russell D W. Molecular Cloning: A Laboratory Manual. 3rd ed. Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press, 2001:512-513. |
| [10] | Inchaisri C, Supikulpong P, Vannametee E, Luengyosluechakul S, Khanda S, Tashnakajankorn T, Ajariyakhajorn K, Sasipreeyajan J, Techakumpu M. The effect of a catastrophic flood disaster on livestock farming in Nakhon Sawan province, Thailand. Tropical Animal Health and Production, 2013, 45(4): 917-922. |
| [11] | De Jager N R. Effects of flood frequency and duration on the allometry of community-level stem size-density distributions in a floodplain forest. American Journal Botany, 2012, 99(9): 1572-1576. |
| [12] | Chung C C, Huang C Y, Gong G C, Lin Y C. Influence of the Changjiang River Flood on synechococcus ecology in the surface waters of the East China Sea. Microbial Ecology, 2014, 67(2): 273-285. |
| [13] | Pontarp M, Canback B, Tunlid A, Lundberg P. Phylogenetic analysis suggests that habitat filtering is structuring marine bacterial communities across the globe. Microbial Ecology, 2012, 64(1): 8-17. |
| [14] | Wang J, Shen J, Wu Y C, Tu C, Soininen J, Stegen J C, He J Z, Liu X Q, Zhang L, Zhang E. Phylogenetic beta diversity in bacterial assemblages across ecosystems: deterministic versus stochastic processes. The Isme Journal, 2013, 7(7): 1310-1321. |
| [15] | McCarthy S A. Effects of temperature and salinity on survival of toxigenic Vibrio cholerae O1 in seawater. Microbial Ecology, 1996, 31(2): 167-175. |
| [16] | Belnap C P, Pan C, Denef V J, Samatova N F, Hettich R L, Banfield J F. Quantitative proteomic analyses of the response of acidophilic microbial communities to different pH conditions. The Isme Journal, 2011, 5(7): 1152-1161. |
| [15] | McCarthy S A. Effects of temperature and salinity on survival of toxigenic Vibrio cholerae O1 in seawater. Microbial Ecology, 1996, 31(2): 167-175. |
| [18] | Shang W, Yang Y X. Degradation characteristics, patterns, and processes of lakeside wetland in Napahai of northwest Yunnan Plateau, Southwest China. Chinese Journal of Applied Ecology, 2012, 23(12): 3257-3265. |
| [19] | Anderson J A H, Hooper M J, Zak J C, Cox S B. Molecular and functional assessment of bacterial community convergence in metal-amended soils. Microbial Ecology, 2009, 58(1): 10-22. |
| [20] | Kroeker K J, Micheli F, Gambi M C, Martz T R. Divergent ecosystem responses within a benthic marine community to ocean acidification. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(35): 14515-14520. |
| [21] | Herlemann D P, Labrenz M, Jurgens K, Bertilsson S, Waniek J J, Andersson A F. Transitions in bacterial communities along the 2000 km salinity gradient of the Baltic Sea. The ISME Journal, 2011, 5(10): 1571-1579. |
| [22] | Abed R M M, Kohls K, De Beer D. Effect of salinity changes on the bacterial diversity, photosynthesis and oxygen consumption of cyanobacterial mats from an intertidal flat of the Arabian Gulf. Environmental Microbiology, 2007, 9(6): 1384-1392. |
| [23] | Zhou Q X, Gibson C E, Zhu Y M. Evaluation of phosphorus bioavailability in sediments of three contrasting lakes in China and the UK. Chemosphere, 2001, 42(2): 221-225. |
| [24] | Godfrey P, Mitchell D. Eutrophication and phosphate detergents. Science, 1972, 177(4051): 816-817. |
| [25] | Lee G F, Jones R A. Detergent phosphate bans and eutrophication. Environment Science & Technology, 1986, 20(4): 330-331. |
| [26] | Qu L M, Yao D, Cong P F. Inorganic nitrogen and phosphate and potential eutrophication assessment in Liaodong bay. Environmental Science, 2006, 27(2): 263-267. |
 2015, Vol. 35
2015, Vol. 35