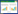文章信息
- 阮宇, 吕佳, 李俊清, 肖国生
- RUAN Yu, LÜ Jia, LI Junqing, XIAO Guosheng
- 利用近似贝氏计算推论台湾海峡沿岸秋茄种群的拓殖路线
- Kandelia obovata Colonization route along Taiwan Strait Inferenced by approximate Bayesian computation
- 生态学报, 2015, 35(13): 4304-4313
- Acta Ecologica Sinica, 2015, 35(13): 4304-4313
- http://dx.doi.org/10.5846/stxb201402200297
-
文章历史
- 收稿日期:2014-02-20
- 修订日期:2015-01-30
2. 北京林业大学, 北京 100083
2. The Academy of Forestry of Beijing Forestry University, Beijing 100083, China
中国台湾(以下简称“台湾”)位于欧亚大陆东南端,约250万年前,由于地壳褶皱不断上升,构成台湾岛现代地形。台湾与大陆之间的台湾海峡形成于更新世冰期[1, 2],第四纪末次盛冰期(Last Glacial Maximum,LGM),海水下降100—120m,台湾海峡成为大陆与台湾之间的陆桥,即著名的东山陆桥[3],陆桥成为两岸物种交流的通道。由于第四纪冰期与间冰期反复出现,大陆与台湾之间也反复出现相接、间断,直至距今约8500年以前,陆桥才最后被淹没[4]。因此,台湾植物区系的形成与大陆植物有非常紧密的关系。台湾植物共1201属,其中与大陆共有的属有1156属[5]。高等植物中生物多样性相似度为79.3%[6]。还出现台湾岛内和横断山区(东喜马拉雅)的高山植物景观极其相似的现象[7]。但台湾特有种也非常丰富,约1070种,占全部种数的29.3%。特有种比例高[5]。台湾的植物区系是新老植物成分并存且共同发展的古老区系,是多次地质事件侵袭后,再活化的历史事件演变的结果。
红树植物在海峡两岸都有分布,且具有非常高的相似性[4],分布于台湾北部竹围的秋茄(Kandelia obovata)与福建福鼎的秋茄种群遗传结构具有较高的相似性,但遗传多样性却低于福鼎种群,且分布于大陆东南沿海秋茄种群的遗传多样性呈现由北向南不断降低的趋势[8]。大多数红树植物的繁殖体具有漂浮能力,随洋流进行漂移。胎生苗漂移是红树植物基因交流的主要方式[9, 10, 11, 12],是红树植物地理分布格局的重要影响因子[13, 14, 15, 16]。当陆桥形成时,有助于两岸陆地物种交流,但对于繁殖体借助于海水进行漂移的红树植物来说,陆桥却可能成为阻碍其扩散的主要因子。因此台湾海峡的陆桥出现便可能成为台湾红树植物起源、拓殖方向的影响因子。黑潮(Kuroshio current)是西太平洋由南向北流动的洋流,将热带的温暖海水带往北方较为寒冷的海域,也同时推动红树植物由南向北迁移[17]。
本研究以分布于两岸的红树植物秋茄种群为研究对象,利用简单重复序列 (simple sequence repeats,SSR)分子标记,通过近似贝氏计算(approximate-Bayesian-computation,ABC)推测台湾红树植物的起源,以期为红树植物保护提出生态保护单元,为红树植物生态恢复与保护提出科学依据。 1 材料与方法 1.1 材料的采集
针对海峡两岸秋茄的自然分布的特点,选取自然分布于广州深圳、福建漳州、福建福鼎、福建宁德、台湾北部竹围及台湾南部四草的秋茄样地。共6个样地,依地理分布位置区分为大陆东南沿海北部种群(N. CN),大陆东南沿海南部种群(S. CN),台湾北部种群(N. TW),台湾南部种群(S. TW)。采集地相关资料分别见表 1及图 1。采集方法:在野外每个样地采集5—10株个体叶片,每株间距至少10m以上。采集的嫩叶片立即放入装有硅胶的塑料袋中干燥,并记录采集地生境类型、海拔高度、个体编号等信息。
| 采集地代号 Population code |
采集地点 Collecting sites |
样品数 Sample size |
经纬度 Coordinate(N/E) |
种群特征 Population characteristics | |
| S.CN | ZJ | 福建漳州漳江口红树林自然保护区竹塔村 | 5 | N 23°55.452′ E 117°24.893′ |
原生群落 |
| SZ | 广东深圳福田红树林保护区 | 5 | N 22°32′ E 114°03′ |
原生群落 | |
| N.CN | FD | 福建宁德福鼎沙埕港 | 5 | N 26°52′—27°26′ E 119°55′—120°43′ | 原生群落 |
| ND | 福建宁德三都湾 | 5 | N 26°18′—27°4′ E118°32′—120°44′ | 原生群落 | |
| N.TW | TP | 台湾北部竹围红树林保护区 | 10 | N 25°09′ E 120°16′ | 原生群落 |
| S.TW | TN | 台湾南部台江国家公园(四草) | 10 | N 23°00′ E 120°08 | 原生群落 |
| N. CN: 大陆东南沿海北部种群,S. CN: 大陆东南沿海南部种群, N. TW: 台湾北部种群,S. TW台湾南部种群 | |||||

|
| 图 1 台湾种群起源及拓殖路线假说 Fig.1 The origin hypotheses and probable migration routes of mangroves in Taiwan,China A:采样地地理位置分布图;B—F:分别为5种可能的起源地及迁移路径;虚线代表隔离事件,箭头方向为可能的拓殖方向和路径;t1—t3分别为各隔离、拓殖事件发生的时间;N.CN:大陆东南沿海北部种群,N. CN: 大陆东南沿海北部种群,S. CN: 大陆东南沿海南部种群,N.TW:台湾北部种群,S.TW:台湾南部种群;FD:福建省福鼎沙埕港、ND:福建省宁德三都湾、ZJ:福建漳州漳江口红树林自然保护区竹塔村、SZ:深圳福田红树林保护区、TP:台湾北部竹围红树林保护区、TN:台湾南部台江国家公园(四草) |
DNA的提取分别采用2×CTAB 法[18]和改良的4×CTAB[19]法进行。改良的4×CTAB 法改进之处主要是在常规2×CTAB 法的基础上,将Tris和EDTA 的浓度都增加1倍。而盐离子(NaCl) 浓度不变。
从已发表的文献中选取18对核基因SSR引物,其中8对引物扩增出较强的多态位点(表 2)。PCR反应体系为20 μL,其中一组的PCR反应条件是94℃变性30s,48℃退火30s,72℃延伸30s,循环数35次。反应前94℃预变性3min,反应后72℃延伸5min。另一组的PCR反应条件是94℃变性30s,53—62℃退火(依引物不同而异,表 2) 30s,72℃延伸1min,循环数40次。反应前94℃预变性9min,反应后72℃延伸5min。
| 引物点 Locus | 序列(5′—3′) Primer sequence |
PCR反应条件 PCR amplification conditions |
| Kcan004 | 5′-CTTGGGGAGGTTTCTCCATTTG-3′ 5′-GCGCCCCTAATTAATGCTT-3′ | 引自Sugaya等2002[20] |
| Kcan009 | 5′-CCCGAACTGATGCAGATTCA-3′ 5′-AGGATGGGTCTTTACAGGTTATTT-3′ | |
| Kcan011 | 5′-AGCCACTCAGGTGTTCTATG-3′ 5′-CAGGTCTCATGGCTGTGTCC-3′ | |
| Kcan034 | 5′-CAGAAGCAGCAAGTAAGGAA-3′ 5′-GAAGAACGTGAAGACAGTGA-3′ | |
| Kaca01a | 5′-ATTCACACTAGTCCACTTCTCCGC-3′ 5′-ACACACACACACACAGAGAG-3′ | 引自Islam等2006[21] |
| Kaca04b | 5′-ATCGTAGCGCACGAATGCTTAATA-3′ 5′-AGAGAGAGAGAGAGACACAC-3′ | |
| Kaca05c | 5′-GTAGTGCGGTGAGGTTTATAGGAA-3′ 5′-ACACACACACACACTCTCTC-3′ | |
| Kaca10d | 5′-TTTTTAGATCTGGCCGGCACGTGC-3′ 5′-TCTCTCTCTCTCACACACACAC-3′ | |
| a、b、c、d: 退火温度分别为60、57、58、62℃ | ||
PCR反应产物经1%琼脂糖凝胶电泳检测确认后,送北京睿博兴科生物技术有限公司,由该公司利用ABI3730自动测序仪(Applied Biosystems)配合Genemarker V1.75软件分析确定微卫星片断长度和识别。微卫星数据用软件MICRO-CHECHER[22]检测非期望突变、大空位(gap)、不正常大小的等位基因或无效等位基因。分析位点都未发现异常等位基因。无异常位点等位基因数据用于数据处理。
1.3 数据分析 1.3.1 遗传多样性参数遗传多样性的估算采用GenALEx6.2[23]计算有效等位基因数、观测杂合度、期望杂合度、种群分化指数FST和遗传距离。并以ARLEQUIN3.0[24]对种群遗传结构分子变异 (AMOVA)进行分析,程序运行中进行10000次的重复模拟检验后取得遗传变异的期望值分布区间,并检测观测值是否偏离期望值,以评估台湾海峡两岸的秋茄是否呈明显种群结构。
1.3.2 遗传组成相似性及归群分析种群遗传相似度归群分析采用STRUCTURE 2.3.3[25, 26, 27]进行种群遗传结构分析,该程序基于贝叶斯聚类分析方法(Bayesian clustering analysis)计算种群的遗传组成,并估计种群间遗传混杂程度。本研究运用马尔可夫链蒙特卡尔法(Markov chain Monte Carlo)针对K=1—7进行运算。每个K值进行10次独立运算。每次运算进行1000000次模拟,再选择舍弃100000资料(burn-in),并使用选择混合祖先模型(admixture model)。各K值利用STRUCTURE HARVESTER V 0.6.8[28],收集其概率L(K)并以ΔK作为标准判断最好的K值。求最大可能K值,将该K值对应的10次独立运算结果以CLUMPP V1.1.2[29]进行10000次随机顺序整合。最后用DISTRUCT[30]软件绘制成种群遗传结构图。
1.3.3 贝叶斯近似计算法(简称ABC)中拓殖路线假说构建假说1:起源受到陆桥的影响,台湾红树植物南、北种群呈多次起源,有多条迁移路线。假说2:起源未受到陆桥的影响,台湾红树植物呈一次性起源,台湾种群从东南北部起源,向东迁移至台湾北部再沿台湾西海岸拓殖到台湾南部。假说3:起源未受到陆桥的影响,台湾红树植物呈一次性起源,东南南部是起源地,向东迁移至台湾南部再沿台湾西海岸拓殖到台湾北部。假说4:起源未受到陆桥的影响,台湾红树植物呈一次性起源,东南北部是起源地,向东南迁移至台湾南部再沿台湾西海岸拓殖到台湾北部。假说5:起源未受到陆桥的影响,台湾红树植物呈一次性起源,东南南部是起源地,向东北迁移至台湾北部再沿台湾西海岸拓殖到台湾南部(图 1)。
1.3.4 贝叶斯近似计算模型的构建与评估为检测台湾秋茄种群的起源及拓殖路线假说,采用DIYABC软件[31]进行检测(图 1,图 2)。每一个假说(假设为一个情境)构建成一个模型,分别进行12000000次模拟,模拟参数的估计范围设定为默认值。DIYABC模拟完成后,选取最接近观测值的35000个数据计算逻辑回归概率值,以评估最佳情境。DIYABC参数设定(1)N1,N2,N3和N4分别为大陆东南沿海北部种群、大陆东南沿海南部种群、台湾北部种群及台湾南部种群的有效种群大小。(2)设定秋茄种群历史时间。由于末次盛冰期(距今约2万年前)台湾岛与大陆通过陆桥相连,因此现今的台湾西部海岸当时是陆地而无红树林分布,故可假设台湾的秋茄种群的拓殖历史小于2万年,由于秋茄胎生苗从萌发至结实最快为两年,秋茄的每一世代最短为3a。因而台湾秋茄种群拓殖历史时间t2及t1上限设定为1万代(约3万年),而大陆东南沿海南北种群t3上限则设定为10万代(约20万年前),且根据种群发展的时间顺序设定t3> t2 > t1。
|
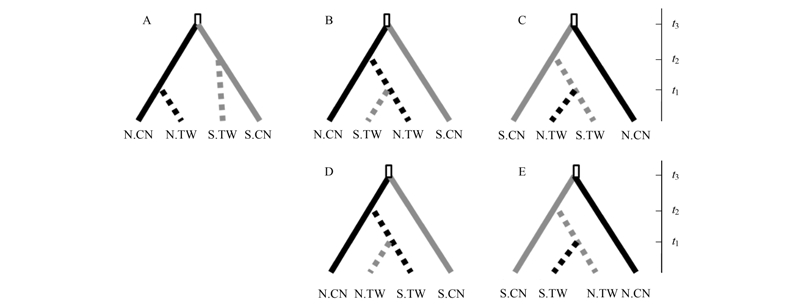
| 图 2 5种可能的情境模型 Fig.2 Five colonization scenarios of K. obovata 实线为大陆东南沿海种群,虚线为台湾种群;黑线为北部种群、灰线为南部种群; t1—t3分别为各隔离、拓殖事件发生的时间情境 |
4个区域8个SSR位点分析结果表明,8个位点多态性为100%。有效等位基因平均数是4.537±0.453,变化范围是6.537±1.019(S.CN)至3.364±0.522(C.CN)。Shannon′s index是1.526±0.079,变化范围是1.892±0.173(S.CN)至1.27±0.149(C.CN)。期望杂合度是0.728±0.022,变化范围是0.811±0.036(S.CN)至0.648±0.054(C.CN)(表 3)。4个种群中分布于大陆东南沿海南部的遗传多样性最高,而分布于大陆东南沿海北部多样性最低。从分子变异分析(AMOVA)可得,13.08%的分化来源于种群间,86.98%的分化来源种群内的个体间。群体间差异明显(FST=0.1308,P=0.000)(表 4)。但种群间两两比较,大陆东南沿海北部种群与其他种群差异最大,大陆东南沿海南部种群与台湾南、北种群的差异较小分别为0.05103和0.09491(表 5)。
|
自然群落 Populations code | Na | Ne | I | Ho | He | FST |
| S.CN | 8.375±1.179 | 6.537±1.019 | 1.892±0.173 | 0.925±0.037 | 0.811±0.036 | -0.164±0.088 |
| N.CN | 4.875±0.639 | 3.364±0.522 | 1.27±0.149 | 0.858±0.067 | 0.648±0.054 | -0.338±0.068 |
| N.TW | 5.125±0.515 | 4.045±0.437 | 1.451±0.098 | 1.000±0.000 | 0.736±0.023 | -0.368±0.042 |
| S.TW | 5.875±0.693 | 4.2±0.687 | 1.493±0.14 | 0.820±0.083 | 0.718±0.041 | -0.133±0.103 |
| 总计Total | 6.063±0.453 | 4.537±0.397 | 1.526±0.079 | 0.901±0.029 | 0.728±0.022 | -0.251±0.042 |
| Na:等位基因Allele, Ne:有效等位基因Effective allele, I:香农指数Shannon′s index, Ho:观察杂合度Observed heterozygosity, He:期望杂合度Expected heterozygosity, FST:种群分化指数Population differentiation coefficient | ||||||
| 变异来源 Source of variation |
自由度 df |
方差和 Sum of squares |
方差分量 Variance components |
变异百分比 % Variation |
GST |
| 种群间Among populations | 3 | 22.163 | 0.27688 | 13.02 | 0.13018 |
| 个体间Among individuals | 76 | 140.600 | 1.85000 | 86.98 | |
| 总计Total | 79 | 162.762 | 2.12688 |
| S.CN | N.CN | N.TW | S.TW | S.CN | N.CN | N.TW | S.TW | |||
| S.CN | 0.00000 | N.TW | 0.09491** | 0.2442** | 0.00000 | |||||
| N.CN | 0.13946** | 0.00000 | S.TW | 0.05103** | 0.17343** | 0.06022** | 0.00000 | |||
|
* |
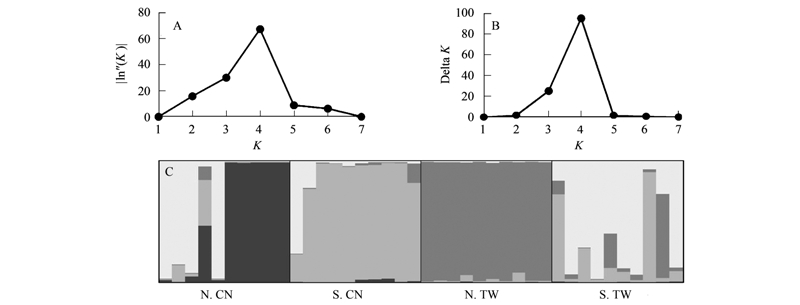
贝叶斯聚类分析的结果呈现四大地理分区6个种群的归群方式中,当归群数(K)为4时,ΔK值最大,其中漳江口自然保护区与福田红树林保护区两个种群的遗传组成归为同群,福鼎沙埕港与三都湾两个种群归群为同群(图 3),表示本研究根据地理分布而归群的大陆东南沿海北部、南部地理区与遗传组成的分布一致。然而台湾南、北两种群的遗传组成较大陆东南沿海两地理区要复杂,其中台湾北部的竹围红树林自然保护区的秋茄在遗传组成可分成两群,呈现种群内的遗传分化现象,检测出其中部分个体来自大陆东南沿海南部的遗传成分,呈现两地理区间可能存在一定程度的基因交流;而台湾南部的台南四草种群的遗传组成多为台湾北部种群的其中之一(图 3白色部分),而少数个体也有来自大陆东南沿海南部及北部的遗传成分,表明该种群可能在近代有较频繁的基因交流。
|
| 图 3 利用Structure对4个种群进行结构分析结果 Fig.3 Results of the Bayesian clustering analysis of sampling areas 图A、B分别是以ln”(K)与delta K判断最佳分群数(K)为4时的图形,图C是当K=4时的各种群的遗传组成 |
利用DIYABC软件评估情境中,以逻辑回归计算最接近观测值的35000次模拟值与观测值的偏差所得到的情境1至情境5的后验概率密度,得到最高的后验概率密度为情境5,其平均值为0.5515(95%CI: 0.5403—0.5627),比情境1至情境4的后验概率密度高(情境1—4的值0.0001—0.2054)(图 4)。情境5即近似贝氏计算评估大陆东面沿海南北种群分岐早,可能有各自不同的来源,且台湾秋茄种群从大陆东南沿海南部种群起源向东北迁移至台湾北部后,再向南拓殖到台湾南部的假说获得较高的支持。

|
| 图 4 图 1及图 2中5种情境的后验概率逻辑回归分析结果 Fig.4 Logistic regression analysis of posterior probabilities for five biogeographic scenarios illustrating in Fig. 1 and Fig. 2 |
根据情境5的模拟参数结果显示大陆南部种群具有较大的有效种群大小 (NS.CN = 77000,95%CI = 15600—98000),其次为台湾南部种群(NS.TW = 11500,95%CI = 3180—47300),两岸的北部种群皆小于南部种群(大陆北部种群NN.CN = 1930,95%CI = 701—9500;台湾北部种群NN.TW = 2010,95%CI = 720—8730)(表 6)。根据有效种群大小的估算结果也显示南部种群的遗传多样性高于北部种群。种群拓殖的时间估计表明,台湾北部种群拓殖至南部种群的时间(t1)约为2520个世代前(95%CI = 870—7540),倘若秋茄一个世代时间约为3—5a(自胎生苗萌发至成树结实),则南迁时间约为7560a至12600a前。然而台湾秋茄种群的起源时间(t2)的估计值为9670个世代前(95%CI=2230—9890),约为29000—48400a前。而大陆东南沿海南部种群与北部种群的溯祖时间为50400个世代前(95%CI = 9280—97600),约为15.1万年至25.2万年前,约为更新世中期末,早于后更新世(late-Pleistocene,12.3万年前至1万年前)时的全球气温下降至末次全冰时期(约11.5万年前至1万年前)的时间。该溯祖时间显示大陆东南沿海南、北种群可能有不同的起源,或受末次冰期时海平面下降的隔离而分化。
| 参数 Parameter |
平均值 Mean |
中值 Median |
众数 Mode |
q025 | q975 |
| NN.CN | 4.24×103 | 3.74×103 | 1.93×103 | 7.01×102 | 9.50×103 |
| NS.CN | 6.01×104 | 6.11×104 | 7.70×104 | 1.56×104 | 9.80×104 |
| NN.TW | 3.58×103 | 3.16×103 | 2.01×103 | 7.20×102 | 8.73×103 |
| NS.TW | 1.97×104 | 1.71×104 | 1.15×104 | 3.18×103 | 4.73×104 |
| t1 | 3.62×103 | 3.39×103 | 2.52×103 | 8.70×102 | 7.54×103 |
| t2 | 7.02×103 | 7.40×103 | 9.67×103 | 2.23×103 | 9.89×103 |
| t3 | 5.35×104 | 5.32×104 | 5.04×104 | 9.28×103 | 9.76×104 |
| NN.CN,NS.CN,NN.TW,NS.TW分别表示大陆东南沿海北部种群、南部种群及台湾北部种群、南部种群的有效种群大小; t1,t2,t3分别为各隔离、拓殖事件发生的时间, q025为95%置信区间的下限值, q975表示为95%置信区间的上限值 | |||||
本研究利用8组SSR分子标记估算了秋茄整体遗传多样性程度(He = 0.728±0.022)与Geng等[32]针对广东湛江种群的估算相似(He = 0.704—0.738),但若只针对大陆东南沿海南部种群的估算(He = 0.811±0.036)略高于Geng的估算值。而对台湾地区的一般多样性评估上,本研究的所估算的遗传多样性各指数在Na,Ne及I上北部种群皆略小于南部种群,但杂合子比例期望值(He)呈现北部种群较高的现象,贝叶斯聚类分析的结果中呈现台湾北部种群内有明显的种群内分化现象,可解释其等位基因数低但杂合子比例期望值高的原因。而此结果也与Chiang等[33]的叶绿体DNA序列分析结果类似。在整体评估上,大陆东南沿海南部种群遗传多样性最为丰富,该地区气候与生境有利于秋茄的生长,且大陆东南沿海南部也可能是亚洲红树植物分布区的多样性中心之一,Liao等[12]曾研究南中国海周边的角果木(Ceriops tagal)时发现,分布在海南岛的角果木拥有较为古老的基因型。此外,大陆东南沿海北部是秋茄的分布临界,生境温度较低,在生境因子中温度对秋茄的影响最大,它不但限制秋茄的生理生长,也限制了秋茄的遗传结构的复杂性。
3.2 台湾种群的起源及秋茄的迁移路线由5个情境模型模拟计算结果可以推测台湾种群起源于大陆东南南方种群(图 3,表 6)。其起源时间大约在29000—48400年前,早于末次盛冰期(大约2万年前),东南南部种群拓殖至台湾北部的时间早于末次盛冰期,意味着现今台湾北部秋茄种群的起源可能并未受到东山陆桥的影响,可能在末次冰期前与东南南部种群共同起源自南方的共同祖先。就全球尺度来看,红树科植物在第三纪进入了地质历史最繁盛的时期[34]。但到了第三纪后期,从上新世开始由于气候呈现较大幅度的变化,而影响全球植物的分布,至更新世冰期的出现,红树植物的分布范围大大缩小,据1994年张玉兰对红树植物花粉在我国东南部海域沉积物中的分布及古环境意义的研究中表明,南海沿岸沉积了更新世中、晚期的花粉,这些花粉主要由本地原生的红树植物花粉沉积而成[17]。表明红树科植物南部种群起源古老这些研究与本文的研究一致 (表 3)。随着冰期结束海退,黑潮(Kuroshio current,流经台湾东部,沿琉球群岛至日本东岸或黑潮支流(Kuroshio branch current,自台湾南端进入台湾海峡)对胎生苗迁徙的影响非常明显,许多研究表明[17, 35],黑潮影响了秋茄的分布,大陆沿海南部海域周围的秋茄胎生苗也可能随黑潮支流向北漂移并定居于台湾北部,而后进向台湾南部拓殖。而贝叶斯聚类分析的结果亦显示台湾的秋茄种群遗传结构受近期基因交流的影响高于大陆东南沿海种群,故检测出部分个体来自大陆沿岸的遗传成分,显示海岛种群受外来基因的影响高于大陆种群的现象。
3.3 东南沿海南、北种群的分化原因东南沿海原始的秋茄种群的溯祖时间(15.1万年至25.2万年前)早于晚更新世时期,则意味(1)东南沿海南、北种群的遗传分化可能受到气候变化与海侵海退的影响,或(2)东南沿海南、北种群可能来自不同的起源。结果与分子变异分析 (AMOVA)和STRUCTURE的分析结果相同,4个种群间都已出现遗传分化,但大陆东南北部种群的遗传差异与其他3个种群间分化最大(表 5与图 3)。分布于东南北部种群的起源可能与南部种群起源不同,这个结果与AFLP的分析研究结果相同[8]。种群的分化也可能来自各种群不同的栖息地环境条件差异所引起,Ruan等[8]也利用AFLP为分子标记,区分出适应性基因座与中性基因座,再进行了分析比较,推论出种群间的分化可能受到部分的区域性适应(local adaptation)所致,因为该区域的选择压力造成的分化可能与地理分布因子无关。温度是影响秋茄分布的主要因子[36, 37, 38, 35],东南北部是秋茄的分布北界,因而温度因子也可能影响秋茄南、北种群分化。然而适应性分化的推论还需要更多的分子及生理证据支持。地理环境及其他因子的影响使秋茄种群出现明显分化的结果与Chen等[39]用ISSR分子标记进行遗传结构分析所得推论一致。
4 结语与种群遗传结构保护建议秋茄种群的起源古老,特别大陆东南沿海南部种群可能是台湾海峡周围秋茄的起源中心,也是遗传多样性的中心,所以保护大陆东南沿海南部种群是红树林保护的重点。大陆东南沿海北部种群出现与其他种群分化程度高的现象,显示了一独立于其他种群之外的演化显著单元,应视为独立的保护经营单元。进行人工引种栽培时,需注意南、北种群引种地的选择。避免引种栽培时将南、北种群的种苗混杂栽植(即南苗北栽或北苗南栽)而造成基因混杂,导致原生基因被外来基因取代而使遗传多样性丧失。从DIYABC研究推断台湾南部种群是由台湾北部种群拓殖定居形成的,而北部种群与大陆东南沿海南部种群可能同时起源于一个中心,然而因长期的海峡隔离以及可能来自其他地区(如琉球)的基因交流,台湾地区的秋茄种群也明显与大陆东南沿海种群有遗传上的分化。台湾地区虽对西部沿海各红树林重要分布地成立了保护区或国家公园以维持原生种群,但仍需注意在其他河口滩地进行引种栽培时的种苗来源。避免来源不明的种苗进行人工栽培。
致谢:台湾师范大学廖培钧副教授及黄生教授对本研究给予指导,北京林业大学谢磊老师及吴江梅老师建议与帮助,特此致谢。
| [1] | Lin C C. Geology and ecology of Taiwan prehistory. Asian Perspectives, 1963, 7(1/2): 203-213. |
| [2] | Yu H S. Geological characteristics and distribution of submarine physiographic features in the Taiwan region. Marine Georesources and Geotechnology, 2003, 21(3/4): 139-153. |
| [3] | 林观得. 台湾海峡海底地貌的探讨. 台湾海峡, 1982, 1(2): 58-63. |
| [4] | Huang Z Y, Yu H S. Morphology and Geologic Implications of Penghu Channel off southwest Taiwan. Terrestrial, Atmospheric and Oceanic Sciences, 2003, 14(4): 469-485. |
| [5] | 应俊生, 徐国士. 中国台湾种子植物区系的性质、特点及其与大陆植物区系的关系. 植物分类学报, 2002, 40(1): 1-51. |
| [6] | 张娆挺, 胡慧娟. 台湾海峡两岸高等植物区系研究. 台湾海峡, 1998, 17(4): 417-425. |
| [7] | 陈毅峰, 陈宜瑜, 何德奎, 隋晓云. 台湾海峡两岸生物地理学现状与展望. 动物学杂志, 2006, 41(1): 118-122. |
| [8] | Ruan Y, Huang B H, Lai S L, Wan Y T, Li J Q, Huang S, Liao P C. Population genetic structure, local adaptation, and conservation genetics of Kandelia obovata. Tree Genetics & Genomes, 2013, 9(4): 913-925. |
| [9] | Giang L H, Geada G L, Hong P N, Tuan M S, Lien N T H, Ikeda S, Harada K. Genetic variation of two mangrove species in Kandelia (Rhizophoraceae) in Vietnam and surrounding area revealed by microsatellite markers. International Journal of Plant Sciences, 2006, 167(2): 291-298. |
| [10] | Liao P C, Havanond S, Huang S. Phylogeography of Ceriops tagal (Rhizophoraceae) in Southeast Asia: the land barrier of the Malay Peninsula has caused population differentiation between the Indian Ocean and South China Sea. Conservation Genetics, 2007, 8(1): 89-98. |
| [11] | Liao P C, Chiang Y C, Huang S, Wang J C. Gene flow of Ceriops tagal (Rhizophoraceae) across the Kra Isthmus in the Thai Malay Peninsula. Botanical Studies, 2009, 50(2): 193-204. |
| [12] | Liao P C, Hwang S Y, Huang S, Chiang Y C, Wang J C. Contrasting demographic patterns of Ceriops tagal (Rhizophoraceae) populations in the South China Sea. Australian Journal of Botany, 2011, 59(6): 523-532. |
| [13] | Rabinowitz D. Dispersal properties of mangrove propagules. Biotropica, 1978, 10(1): 47-57. |
| [14] | Ellison J C. Pollen evidence of Late Holocene mangrove development in Bermuda. Global Ecology and Biogeography Letters, 1996, 5(6): 315-326. |
| [15] | Delgado P, Hensel P F, Jiménez J A, Day J W. The importance of propagule establishment and physical factors in mangrove distributional patterns in a Costa Rican estuary. Aquatic Botany, 2001, 71(3): 157-178. |
| [16] | Steinke T D, Ward C J. Use of plastic drift cards as indicators of possible dispersal of propagules of the mangrove Avicennia marina by ocean currents. African Journal of Marine Science, 2003, 25(1): 169-176. |
| [17] | 张玉兰, 王开发. 红树植物花粉在我国东南部海域沉积物中的分布及古环境意义. 海洋与湖沼, 1994, 25(1): 23-28. |
| [18] | Doyle J J, Doyle J L. A rapid DNA isolation Procedure for small quantities of fresh leaf tissue. Phytochemical Bulletin, 1987, 19(1): 11-15. |
| [19] | 庞雅文, 段立柱, 宋德应, 任竹梅. 盐肤木基因组DNA提取方法改进及AFLP体系的建立. 广西植物, 2011, 31(1): 36-38. |
| [20] | Sugaya T, Takeuchi T, Yoshimaru H, Katsuta M. Development and polymorphism of simple sequence repeat DNA markers for Kandelia candel (L.) Druce. Molecular Ecology Notes, 2002, 2(1): 65-66. |
| [21] | Islam M S, Tao J M, Geng Q F, Lian C L, Hogetsu T. Isolation and characterization of eight compound microsatellite markers in a mangrove tree Kandelia candel (L.) Druce. Molecular Ecology Notes, 2006, 6(4): 1111-1113. |
| [22] | Oosterhout C V, Hutchinson W F, Wills D P M, Shipley P. MICRO-CHECKER: software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 2004, 4(3): 535-538. |
| [23] | Smouse P E, Peakall R, Gonzales E. A heterogeneity test for fine-scale genetic structure. Molecular Ecology, 2008, 17(14): 3389-3400. |
| [24] | Excoffier L, Laval G, Schneider S. Arlequin (version 3. 0): An integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, 2005, 1: 47-50. |
| [25] | Falush D, Stephens M, Pritchard J K. Inference of population structure: Extensions to linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587. |
| [26] | Falush D, Stephens M, Pritchard J K. Inference of population structure using multilocus genotype data: dominant markers and null alleles. Molecular Ecology Notes, 2007, 7(4): 574-578. |
| [27] | Pritchard J K, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155(2): 945-959. |
| [28] | Earl D A, von Holdt B M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method. Conservation Genetics Resources, 2012, 4(2): 359-361. |
| [29] | Jakobsson M, Rosenberg N A. CLUMPP: a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure. Bioinformatics, 2007, 23(14): 1801-1806. |
| [30] | Rosenberg N A. DISTRUCT: a program for the graphical display of population structure. Molecular Ecology Notes, 2004, 4(1): 137-138. |
| [31] | Cornuet J M, Ravigné V, Estoup A. Inference on population history and model checking using DNA sequence and microsatellite data with the software DIYABC (v1. 0). BMC Bioinformatics, 2010, 11: 401. |
| [32] | Geng Q F, Lian C L, Goto S, Tao J M, Kimura M, Islam M D S, Hogetsu T. Mating system, pollen and propagule dispersal, and spatial genetic structure in a high-density population of the mangrove tree Kandelia candel. Molecular Ecology, 2008, 17(21): 4724-4739. |
| [33] | Chiang T Y, Chiang Y C, Chen Y J, Chou C H, Havanond S, Hong T N, Huang S. Phylogeography of Kandelia candel in East Asiatic mangroves based on nucleotide variation of chloroplast and mitochondrial DNAs. Molecular Ecology, 2001, 10(11): 2697-2710. |
| [34] | 金建华. 红树科植物的化石记录及植物地理史. 生态学报, 2005, 25(4): 676-681. |
| [35] | Kao W Y, Shih C N, Tsai T T. Sensitivity to chilling temperatures and distribution differ in the mangrove species Kandelia candel and Avicennia marina. Tree Physiology, 2004, 24(7): 859-864. |
| [36] | Analuddin K, Sharma S, Suwa R, Hagihara A. Crown foliage dynamics of mangrove Kandelia obovata in Manko Wetland, Okinawa Island, Japan. Journal of Oceanography, 2009, 65(1): 121-127. |
| [37] | Gwada P, Makoto T, Uezu Y. Leaf phenological traits in the mangrove Kandelia candel (L.) Druce. Aquatic Botany, 2000, 68(1): 1-14. |
| [38] | Hoque A T M R, Sharma S, Suwa R, Mori S, Hagihara A. Seasonal variation in the size-dependent respiration of mangroves Kandelia obovata. Marine Ecology Progress Series, 2010, 404: 31-37. |
| [39] | Chen S B, Ding W Y, Qiu J B, Wang G Y, Zhou Z M, Chen J F, Ai W M, Wang C Y, Xie Q L. The genetic diversity of the mangrove Kandelia Obovata in China revealed by ISSR analysis. Pakistan Journal of Botany, 2010, 42(6): 3755-3764. |
 2015, Vol. 35
2015, Vol. 35