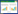文章信息
- 周宁宁, 王梦馨, 崔林, 潘铖, 张新亭, 韩宝瑜
- ZHOU Ningning, WANG Mengxin, CUI Lin, PAN Cheng, ZHANG Xinting, HAN Baoyu
- 基于CO I基因全长序列的假眼小绿叶蝉地理种群遗传分化研究
- Genetic variation of Empoasca vitis (Göthe) (Hemiptera: Cicadellidae) among different geographical populations based on mtDNA CO I complete sequence
- 生态学报, 2014, 34(23): 6879-6889
- Acta Ecologica Sinica, 2014, 34(23): 6879-6889
- http://dx.doi.org/10.5846/stxb201404160745
-
文章历史
- 收稿日期:2014-4-16
- 网络出版日期:2014-03-18
假眼小绿叶蝉Empoasca vitis (GÜthe) 隶属于半翅目(Hemiptera) 、叶蝉科(Cicadellidae)、小绿叶蝉属(Empoasca),广泛分布于我国大陆茶区,繁殖快,世代重叠严重,抗药性强,防治难度大,是我国茶树上为害最重的害虫。常年假眼小绿叶蝉可致长江中下游茶区夏、秋茶减产10%—15%,严重发生年份达50%以上[1]。研究者们对于假眼小绿叶蝉生物学习性、地理分布、危害损失估计和防治等进行了深入的研究[2, 3],但关于其地理种群遗传和分化情况的报道甚少,仅应用 RAPD-PCR 技术鉴定了茶园小绿叶蝉类的优势种[4]。我国幅员辽阔,茶树广泛种植于秦岭淮河以南的祖国南半壁,各茶区的自然地理条件差别大,茶树种植制度不同,可以造就不同地理区域的种群或生态型;另一方面,假眼小绿叶蝉成虫善爬善跳还具有一定的飞行能力,若虫也善于爬行和跳跃,成、若虫能适应各种茶园生境。如果这种非远距离的扩散连续地或渐次地发生,则有可能使得不同地理区域的种群发生基因交流[5]。
随着分子系统地理学的发展,分子标记技术越来越多地应用到分子系统学研究中。mtDNA具有严格遵循母系遗传,不发生重组、倒位、易位等突变现象以及进化速率快等特点,而常被用于分析昆虫物种间及物种内种群的分化程度[6, 7]。CO I基因是线粒体蛋白编码基因之一,密码子第三位碱基可相对自由变异[8]。且CO I的演化速率是其他线粒体蛋白编码基因的3倍,两端序列相对保守,是较好的分子标记[9, 10],已被用于研究玉米黄翅叶蝉Dalbulus maidis、黄条脊冠叶蝉Aphrodes leafhoppers等叶蝉的遗传多样性[11, 12]。
目前主要以CO I基因的部分序列进行遗传多样性研究。本实验首次扩增得到我国13个主要茶叶种植省份的茶园假眼小绿叶蝉线粒体CO I基因的全长序列,通过滑动窗口分析确定其突变位点的分布,并发现了茶园假眼小绿叶蝉线粒体CO I基因存在不完整的终止密码子。再利用此序列分析了茶园假眼小绿叶蝉种群间的遗传多样性、遗传分化程度、基因流水平及分子变异情况等,以探讨长期的地理阻隔对其种群遗传特性的影响及其生态适生性、以及该叶蝉自身扩散和人工助迁等因素造成的种群变异,为针对不同地理区域假眼小绿叶蝉种群的控制策略提供参考。
1 材料和方法 1.1 供试虫源本实验所用茶园假眼小绿叶蝉,在2011年10月—2013年10月期间采集于山东青岛、河南西九华山、江苏苏州、安徽敬亭山、湖北英山、四川乐山、重庆永川、江西婺源、浙江兰溪、湖南长沙、福建武夷山、广西桂林、和云南勐海等13个产茶省的重点产茶县市,详情见表 1。采集的成虫和若虫浸泡于无水乙醇中,放置于-20℃长期保存备用。
| 种群代码 Population code | 采集地点 Sampling location | 地理坐标 Geo-coordinates | 采集时间 Collection date | 分析个体数 Number of individuals assayed |
| SDQD | 山东青岛 | 36.28°N,120.32°E | 2013.1 | 12 |
| HNXJHS | 河南西九华山 | 31.81°N,115.81°E | 2013.08 | 12 |
| JSSZ | 江苏苏州 | 31.06°N,120.37°E | 2012.11 | 12 |
| AHJTS | 安徽敬亭山 | 30.97°N,118.69°E | 2012.07 | 18 |
| HBYS | 湖北英山 | 30.80°N,115.78°E | 2013.08 | 15 |
| SCLS | 四川乐山 | 29.60°N,103.55°E | 2011.1 | 15 |
| CQYC | 重庆永川 | 29.38°N,105.90°E | 2012.11 | 11 |
| JXWY | 江西婺源 | 29.26°N,117.85°E | 2013.05 | 12 |
| ZJLX | 浙江兰溪 | 29.12°N,119.47°E | 2011.1 | 17 |
| HNCS | 湖南长沙 | 28.18°N,113.08°E | 2012.11 | 12 |
| FJWYS | 福建武夷山 | 27.65°N,117.96°E | 2013.06 | 11 |
| GXGL | 广西桂林 | 25.29°N,110.35°E | 2013.06 | 12 |
| YNMH | 云南勐海 | 21.99°N,100.42°E | 2011.1 | 18 |
取单头假眼小绿叶蝉,放在加有适量液氮的1.5 mL离心管中,用无菌的研磨棒磨成粉末,采用动物组织快速提取试剂盒(VWI),按照试剂盒说明书步骤进行基因组DNA提取。从提取的DNA样品中取1 μL用Nanodrop 2000c 仪器(Thermo Scientific公司)和1%琼脂糖凝胶电泳检测其浓度和纯度,基因组DNA保存于-20℃备用。
1.3 CO I基因全长扩增和序列测定根据叶蝉科线粒体CO I基因两端的保守区域,设计特异性引物用于扩增 CO I基因全长序列(扩增产物包括CO I基因两端的侧翼序列,5′包括基因NADH 脱氢酶亚基2的部分序列,以及tRNATrp、tRNACys、tRNATyr3个相连的tRNA序列;3′端包括tRNALeu(UUR)序列以及CO II基因部分序列)。引物序列:CO I-F(正向引物):5′- AACAACAGC TTTCTAGAAATTTGC-3′,CO I-R(反向引物):5′-TTCCATAGTTGGTGAAGAAGC-3′。扩增片段的长度约为1773 bp。
PCR反应体系总体积为25 μL,含2.0 μL的基因组DNA作为模板,2.5 μL 10×PCR Buffer(Mg2+ plus),2 μL dNTP,16 μL ddH2O,1 μL CO I-F,1 μL CO I-R,0.5 μL Taq DNA聚合酶。PCR扩增程序:94℃预变性3 min,94℃变性 30 s,52℃退火30 s,72℃延伸2 min,35个循环,最后72℃延伸10 min。PCR产物经电泳检测确认成功扩增后,委托上海桑尼生物科技有限公司测序。
1.4 数据整理与分析测序结果通过Chromas软件读取,用DNAMAN7.0 对序列进行编辑,去除两端的侧翼序列。用Clustal X 1.8软件进行多序列同源比对,并辅以人工校对,生成用于MEGA5.1软件分析的数据格式[13]。将整理后的序列在NCBI网站上进行BLAST比对,确定目标序列。使用MEGA5.1软件分析碱基组成、变异位点及碱基转换颠换比;基于Kimura-2-Parameter模型计算种群间的遗传距离;以琉璃叶蝉Homalodisca vitripennis CO I基因全长序列作为外群,采用NJ邻接法构建单倍型系统发育树,系统发育树各分支的置信度(bootstrap)均进行1000次重复检验[14]。采用NETWORK4.6.1.2软件绘制单倍型间的进化网络图[15]。利用DnaSP5.0软件分析13个种群内的单倍型多样性Hd、核苷酸多样性Pi、核苷酸平均差异数k,种群间核苷酸平均差异数Kxy、核苷酸歧义度Dxy、遗传分化系数Gst与固定系数Fst等分子遗传学系数,同时生成滑动窗口,分析突变位点在全长序列中的分布,并进行Tajima′s D中性检验[16, 17]。应用Arlequin 3软件进行AMOVA分子变异分析,判断遗传分化的主要来源是种群内或种群间[18]。通过GenALEx 6.41软件检验种群间的遗传距离与地理距离的自然对数矩阵之间的相关性[19]。
2 结果与分析 2.1 茶园假眼小绿叶蝉线粒体CO I基因全长序列及滑动窗口分析通过整理去除测序结果两端侧翼序列,得到茶园假眼小绿叶蝉线粒体CO I基因全长序列,为1534 bp。该基因没有完整的终止密码子,最后一个碱基为T。从已报道的研究中可知,许多半翅目昆虫,如琉璃叶蝉Homalodisca vitripennis(GenBank登录号:NC_006899.1)、青蛾蜡蝉Geisha distinctissima(GenBank登录号:NC_012617.1)[20, 21]以及红头凤沫蝉Paphnutius ruficeps(GenBank登录号:NC_021100.1)[22],其线粒体CO I基因以不完全终止密码子(T)作为停止信号,与本研究所得结果一致。在13个种群176个个体中共发现突变位点113个,约占全长的7.37%,其中有7个位点的突变引起了氨基酸的改变(359,798,967,1175,1316,1330,1422)。单一多态位点40个,简约信息位点73个,未发现碱基缺失或插入现象。由表 2可知,在所有核苷酸替换中,转换的发生频率占87.61%,而颠换仅占12.39%。其中腺嘌呤(A)与鸟嘌呤(G)之间的转换占总转换数的30.8%,胞嘧啶(C)与胸腺嘧啶(T)之间的转换占56.81%,值得注意的是,T转换为C的比例高达43.68%。总体转换/颠换偏倚率R值为7.01。据研究发现,通常亲缘关系较近的分类阶元之间核苷酸替换主要为转换,而在亲缘关系较远的分类阶元之间,核苷酸替换率则以颠换为主[23, 24]。本研究发现茶园假眼小绿叶蝉种内线粒体CO I基因核苷酸突变位点的变异类型以转换为主,验证了上述规律。
| 碱基Basic group | 腺嘌呤 A Adenine A | 胸腺嘧啶 T Thymine T | 胞嘧啶 C Cytosine C | 鸟嘌呤 G Guanine G |
| 腺嘌呤 A Adenine A | - | 2.74 | 0.82 | 11.4 |
| 胸腺嘧啶 T Thymine T | 1.66 | - | 13.13 | 0.98 |
| 胞嘧啶 C Cytosine C | 1.66 | 43.68 | - | 0.98 |
| 鸟嘌呤 G Guanine G | 19.4 | 2.74 | 0.82 | - |
通过软件DnaSP5.0滑动窗口分析CO I全长序列,结果见图 1。从图中可以看到,序列间的突变位点在绝大部分区段内均有分布。其中,在400 bp左右、700 bp到800 bp之间,1200 bp和1300 bp左右有较多的突变位点;前200 bp,900 bp左右,1100 bp左右的突变位点较少。由此可见,与使用基因的部分序列进行遗传分化分析相比,使用序列的全长能够包含更丰富的信息。
|
| 图 1 序列间突变位点的滑动窗口分析 Fig. 1 Sliding window analysis of variable sites among all sequences 窗口长度为75 bp,相邻窗口相互覆盖1/3窗口长度 |
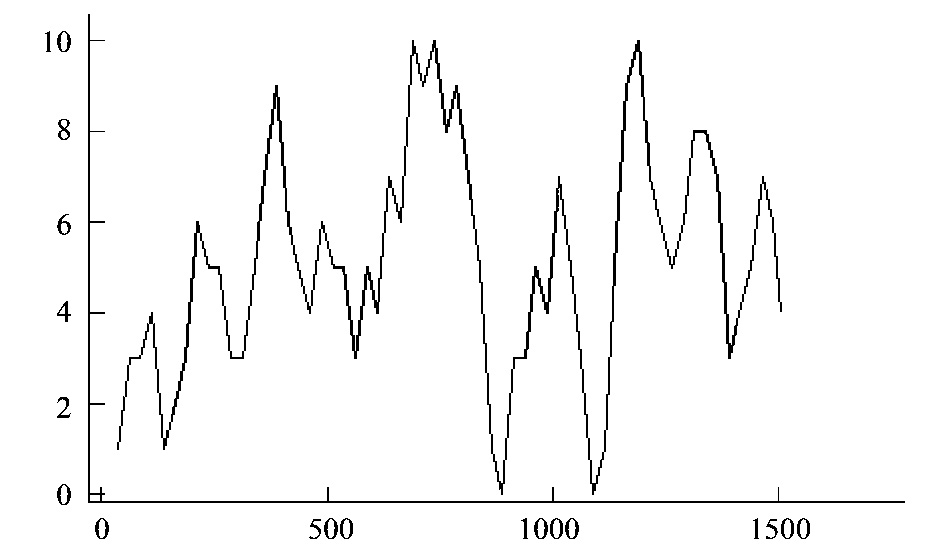
从已获得的176条CO I基因全长序列中定义了105种单倍型:Hap1—Hap105。其中Hap5为共享单倍型,在所有种群中出现24次,占所有种群的13.64%。其余频率较高的单倍型有:Hap16出现13次(7.39%),Hap3和Hap26出现9次(5.11%)。在105种单倍型中,有84种单倍型是由单个种群所特有的独享单倍型。由单倍型系统发育树(图 2,图中仅显示大于30%的置信度值bootstrap)可以看出,在几个大分支中置信度较低,说明这些单倍型之间的差异较小,不能形成可靠的分支。而各种群的单倍型分散在不同的分布群中,未形成明显的系统地理结构。图 3是茶园假眼小绿叶蝉mtDNA CO I基因105 种单倍型间的进化网络图,表现了各地理种群单倍型的分布情况,以及单倍型之间的演化关系。每种颜色代表一个种群的单倍型组成。没有单倍型存在的那些节点是单倍型进化过程中的中间节点。单倍型Hap71在进化网络的较为中心位置,而其他几种出现频率较高的单倍型则处于边缘。由图 3中可以看到,各地理种群的单倍型散布于整个网络图中,每个种群由多种较为原始的单倍型演化产生的单倍型共同组成。说明种群间存在基因交流,分化程度较小。
|
| 图 2 利用邻接法构建的假眼小绿叶蝉不同地理种群CO I单倍型间的系统发育树 Fig. 2 Neighbor joining phylogenetic tree of haplotypes of different geographic populations of Empoasca vitis (GÜthe) based on CO I gene sequence |

|
| 图 3 105 种 mtDNA CO I基因 单倍型的进化网络关系 Fig. 3 Median-joining network of 105 mtDNA CO I gene haplotypes H1—H105表示单倍型,圆的面积表示单倍型在样品中出现的频率,彩色扇形面积代表各种群在同一单倍型中所占的比例 |
各种群CO I基因单倍型、核苷酸多样性分析及Tajima′s D中性检验结果如表 2所示。由表可知,13个种群的总体单倍型多样性指数Hd为0.9720,种群间核苷酸多样性指数Pi为0.00607。种群内核苷酸多样性在0.00413—0.00651范围内,平均为 0.00546。其中单倍型多样性最高的种群是福建武夷山(FJWYS),而云南勐海(YNMH)最低。核苷酸多样性最高和最低的种群分别为湖南长沙(HNCS)和安徽敬亭山(AHJTS)。
对各个地理种群的茶园假眼小绿叶蝉CO I基因全长序列基于Tajima′s D值进行中性检验(表 3),检测结果均不显著,全部种群均为P>0.10,说明茶园假眼小绿叶蝉种群的进化过程遵循中性进化模型,各地理种群在进化过程中没有出现群体扩张,群体大小保持相对稳定。
| 种群代码 Population code | 单倍型 Haplotype | 单倍型多样性 Haplotype d iversity (Hd) | 核苷酸多样性 Nucleotide diversity (Pi) | 核苷酸平均 差异数 Average number of Nucleotide difference (k) | Tajima′s D | 中性检验显著性 Statistical significance |
| ZJLX | Hap1—13 | 0.926 | 0.00541 | 8.294 | 1.30855 | P > 0.10 |
| YNMH | Hap14—22 | 0.804 | 0.00437 | 6.699 | 0.3107 | P > 0.10 |
| SDQD | Hap16,23—28 | 0.879 | 0.00497 | 7.62121 | -1.15821 | P > 0.10 |
| SCLS | Hap5,29—38 | 0.933 | 0.00614 | 9.419 | -1.15821 | P > 0.10 |
| JXWY | Hap5,39—47 | 0.955 | 0.00595 | 9.121 | -1.1604 | P > 0.10 |
| JSSZ | Hap5,7,48—56 | 0.985 | 0.0065 | 9.985 | -1.03223 | P > 0.10 |
| HNXJHS | Hap5,16,52,57—62 | 0.939 | 0.00597 | 9.152 | -1.05377 | P > 0.10 |
| HNCS | Hap26,34,48—49,63—68 | 0.97 | 0.00651 | 9.985 | -1.3644 | P > 0.10 |
| HBYS | Hap5,16,69—78 | 0.962 | 0.00422 | 6.476 | -0.92055 | P > 0.10 |
| GXGL | Hap5,8,12,16,26,79—83 | 0.97 | 0.00582 | 8.924 | -0.93822 | P > 0.10 |
| CQYC | Hap5,26,43,84—90 | 0.945 | 0.00535 | 8.2 | 0.65308 | P > 0.10 |
| FJWYS | Hap5,52,56,91—96 | 1 | 0.00556 | 8.533 | -0.80342 | P > 0.10 |
| AHJTS | Hap5,26,28,71,89,97—105 | 0.967 | 0.00413 | 6.333 | -1.37285 | P > 0.10 |
| 总群体 Total | Hap1—105 | 0.972 | 0.00607 | 9.311 | -1.65533 | 0.10 > P > 0.05 |
应用DnaSP5.0软件计算反映茶园假眼小绿叶蝉种群间基因差异程度的遗传学参数。各种群间核苷酸差异数Kxy在6.47778—13.37500之间,均值为8.98383;核苷酸歧义度Dxy在0.00422—0.00872之间,均值为0.00586(表 4)。各地理种群总遗传分化系数Gst为0.03652,总固定系数Fst为0.10876,总基因流Nm为4.097,固定系数Fst在-0.04168—0.43061之间。其中云南勐海(YNMH)种群与其他种群的遗传分化程度最大(表 5)。说明种群间分化程度低,基因交流频繁。
| 种群代码 Population code | ZJLX | YNMH | SDQD | SCLS | JXWY | JSSZ | HNXJHS | HNCS | HBYS | GXGL | CQYC | FJWYS | AHJTS |
| ZJLX | 12.28758 | 9.01961 | 10.14118 | 8.54412 | 8.85784 | 8.62745 | 9.67157 | 7.49804 | 8.63235 | 8.17647 | 8.30481 | 7.12745 | |
| YNMH | 0.00801 | 10.25000 | 10.16667 | 12.72222 | 13.37500 | 11.62037 | 12.58333 | 9.71481 | 10.49537 | 12.83333 | 12.77778 | 11.44444 | |
| SDQD | 0.00588 | 0.00668 | 9.81667 | 9.55556 | 10.00000 | 9.16667 | 9.40972 | 7.79444 | 8.88194 | 9.68333 | 9.71970 | 7.76852 | |
| SCLS | 0.00661 | 0.00663 | 0.0064 | 10.58889 | 11.35556 | 10.32222 | 10.70000 | 8.90222 | 10.07778 | 10.36000 | 10.53939 | 9.51852 | |
| JXWY | 0.00557 | 0.00829 | 0.00623 | 0.0069 | 9.18750 | 8.77083 | 9.83333 | 7.87778 | 9.15972 | 8.90000 | 8.96212 | 7.58333 | |
| JSSZ | 0.00577 | 0.00872 | 0.00652 | 0.0074 | 0.00599 | 9.22917 | 10.44444 | 8.36111 | 9.72222 | 9.08333 | 9.02273 | 7.99074 | |
| HNXJHS | 0.00562 | 0.00758 | 0.00598 | 0.00673 | 0.00572 | 0.00602 | 9.77778 | 7.67222 | 9.03472 | 8.95833 | 8.71212 | 7.68981 | |
| HNCS | 0.0063 | 0.0082 | 0.00613 | 0.00698 | 0.00641 | 0.00681 | 0.00637 | 8.75000 | 9.95833 | 10.22500 | 9.90152 | 8.64352 | |
| HBYS | 0.00489 | 0.00633 | 0.00508 | 0.0058 | 0.00514 | 0.00545 | 0.005 | 0.0057 | 7.69444 | 7.80667 | 7.89697 | 6.47778 | |
| GXGL | 0.00563 | 0.00684 | 0.00579 | 0.00657 | 0.00597 | 0.00634 | 0.00589 | 0.00649 | 0.00502 | 9.29167 | 9.28030 | 7.70833 | |
| CQYC | 0.00533 | 0.00837 | 0.00631 | 0.00675 | 0.0058 | 0.00592 | 0.00584 | 0.00667 | 0.00509 | 0.00606 | 8.30909 | 7.46667 | |
| FJWYS | 0.00541 | 0.00833 | 0.00634 | 0.00687 | 0.00584 | 0.00588 | 0.00568 | 0.00645 | 0.00515 | 0.00605 | 0.00542 | 7.66162 | |
| AHJTS | 0.00465 | 0.00746 | 0.00506 | 0.00621 | 0.00494 | 0.00521 | 0.00501 | 0.00563 | 0.00422 | 0.00502 | 0.00487 | 0.00499 |
| 种群代码 Population code | ZJLX | YNMH | SDQD | SCLS | JXWY | JSSZ | HNXJHS | HNCS | HBYS | GXGL | CQYC | FJWYS | AHJTS |
| ZJLX | 0.07237 | 0.05047 | 0.02645 | -0.00624 | -0.00385 | -0.00231 | 0.02698 | 0.00881 | 0.00752 | 0.00623 | -0.00659 | 0.00217 | |
| YNMH | 0.38989 | 0.06663 | 0.07042 | 0.06408 | 0.05593 | 0.04985 | 0.05999 | 0.04741 | 0.02245 | 0.05185 | 0.06624 | 0.06066 | |
| SDQD | 0.11774 | 0.30144 | 0.04897 | 0.04348 | 0.03529 | 0.04414 | 0.0253 | 0.03848 | 0.01811 | 0.01464 | 0.04602 | 0.01919 | |
| SCLS | 0.12667 | 0.20729 | 0.13208 | 0.02066 | 0.0156 | 0.02462 | 0.02228 | 0.02243 | 0.02228 | 0.01478 | 0.02248 | 0.01994 | |
| JXWY | -0.01914 | 0.37823 | 0.12394 | 0.12454 | -0.00589 | -0.00503 | 0.01931 | 0.00479 | 0.00878 | -0.00504 | -0.00956 | 0.00009 | |
| JSSZ | -0.03180 | 0.37629 | 0.1197 | 0.14562 | -0.03979 | -0.00927 | 0.00453 | 0.00269 | 0.0045 | -0.00418 | -0.0134 | -0.00051 | |
| HNXJHS | -0.01105 | 0.31797 | 0.08512 | 0.10046 | -0.04168 | -0.03673 | 0.02326 | 0.00586 | 0.00559 | 0.00309 | -0.01375 | 0.00387 | |
| HNCS | 0.05502 | 0.33705 | 0.06448 | 0.09328 | 0.02851 | 0.04401 | 0.02144 | 0.01764 | 0.0119 | 0.00381 | 0.02172 | 0.01226 | |
| HBYS | 0.01506 | 0.32188 | 0.09568 | 0.10723 | 0.01004 | 0.01562 | -0.01846 | 0.05937 | 0.00655 | 0.00445 | 0.00599 | -0.00442 | |
| GXGL | 0.00268 | 0.25569 | 0.06859 | 0.08991 | 0.01496 | 0.02753 | -0.00035 | 0.05059 | -0.00075 | -0.00039 | 0.01024 | 0.00549 | |
| CQYC | -0.02902 | 0.40652 | 0.16586 | 0.13357 | 0.00817 | -0.01935 | 0.01294 | 0.09447 | 0.03867 | 0.06058 | 0.00011 | -0.00482 | |
| FJWYS | 0.00695 | 0.41698 | 0.18613 | 0.16413 | 0.03364 | -0.00772 | 0.00417 | 0.08171 | 0.07077 | 0.07739 | -0.00693 | 0.00123 | |
| AHJTS | -0.02613 | 0.43061 | 0.10185 | 0.17254 | -0.01898 | -0.02107 | -0.00684 | 0.05605 | 0.01127 | 0.01032 | 0.00446 | 0.05155 |
通过AMOVA分子变异分析结果可以看出,不同地理种群之间的变异方差组分为0.59490(占方差比率的12.66%),而种群内的方差组分为4.10510(占方差比率的87.33%)且差异显著(P<0.05)(表 6)。说明这13个茶园假眼小绿叶蝉地理种群的遗传分化主要来自于种群内部,种群间的差异较小。
| 变异来源 Source of variation | 自由度 df | 平方和 Sum of squares | 方差组分 Variance of components | 方差比率 Percentage of variation | P 值 P value |
| 固定系数Fst: 0.10876 | |||||
| 种群间Among populations | 12 | 145.608 | 0.59490 Va | 12.66 | P <0.001 |
| 种群内Within populations | 163 | 669.131 | 4.10510 Vb | 87.34 | P <0.001 |
| 合计Total | 175 | 814.739 | 4.70000 | 100 | |
从表 7可知,各个地理种群之间的遗传距离在0.0042—0.0088之间。应用Mantel相关性检验计算两个矩阵之间的相关系数r为-0.0697(P=0.5373>0.05,1000次随机抽样),表明这两个矩阵之间没有显著的相关性,即不同种群之间的遗传分化程度与地理隔离没有显著关系。
| 种群代码 Population code | ZJLX | SDQD | HNCS | GXGL | FJWYS | AHJTS | JSSZ | CQYC | JXWY | HNXJHS | HBYS | SCLS | YNMH |
| ZJLX | 6.6858 | 6.4494 | 6.9058 | 5.3961 | 5.3901 | 5.4498 | 7.1835 | 5.0639 | 6.1344 | 5.9964 | 7.3423 | 7.6343 | |
| SDQD | 0.0059 | 7.0296 | 7.3454 | 6.8938 | 6.4136 | 6.3649 | 7.3458 | 6.7030 | 6.4745 | 6.6081 | 7.4564 | 7.8223 | |
| HNCS | 0.0063 | 0.0062 | 6.0423 | 6.1813 | 6.4386 | 6.6524 | 6.5695 | 6.1757 | 6.1784 | 5.9707 | 6.8479 | 7.2788 | |
| GXGL | 0.0057 | 0.0058 | 0.0065 | 6.6876 | 6.9410 | 7.0678 | 6.4506 | 6.7608 | 6.8031 | 6.7003 | 6.7156 | 6.9818 | |
| FJWYS | 0.0054 | 0.0064 | 0.0067 | 0.0061 | 5.9304 | 6.0999 | 7.0856 | 5.1904 | 6.2296 | 6.0152 | 7.2610 | 7.5383 | |
| AHJTS | 0.0047 | 0.0051 | 0.0057 | 0.0051 | 0.0049 | 5.0789 | 7.1251 | 5.3319 | 5.6671 | 5.6299 | 7.2877 | 7.6366 | |
| JSSZ | 0.0058 | 0.0066 | 0.0069 | 0.0064 | 0.0060 | 0.0052 | 7.2466 | 5.7513 | 6.0892 | 6.0850 | 7.3918 | 7.7070 | |
| CQYC | 0.0054 | 0.0064 | 0.0065 | 0.0061 | 0.0054 | 0.0050 | 0.0059 | 7.0557 | 6.8946 | 6.8714 | 5.4338 | 6.8967 | |
| JXWY | 0.0056 | 0.0063 | 0.0064 | 0.0060 | 0.0058 | 0.0050 | 0.0060 | 0.0059 | 5.8427 | 5.5723 | 7.2342 | 7.5625 | |
| HNXJHS | 0.0057 | 0.0060 | 0.0064 | 0.0059 | 0.0059 | 0.0050 | 0.0061 | 0.0057 | 0.0058 | 4.7227 | 7.0886 | 7.5364 | |
| HBYS | 0.0049 | 0.0051 | 0.0057 | 0.0050 | 0.0051 | 0.0042 | 0.0055 | 0.0052 | 0.0052 | 0.0050 | 7.0763 | 7.5042 | |
| SCLS | 0.0067 | 0.0064 | 0.0070 | 0.0066 | 0.0068 | 0.0062 | 0.0075 | 0.0069 | 0.0069 | 0.0068 | 0.0058 | 6.8060 | |
| YNMH | 0.0081 | 0.0067 | 0.0083 | 0.0069 | 0.0084 | 0.0075 | 0.0088 | 0.0084 | 0.0084 | 0.0076 | 0.0064 | 0.0067 |
同行专家们也发表了少量关于茶园叶蝉类种群分化和基因流的研究报道。在印度茶小绿叶蝉Empoasca flavescens F. 是茶树上的优势种,印度孟加拉邦的茶小绿叶蝉因茶园管理模式的不同引起了种群的遗传分化[26]。在普通茶园中主要依靠喷施化学农药进行防治,而有机茶园主要依靠茶树品种的抗虫性与施用微生物农药防治,在这两种管理模式下,茶小绿叶蝉面临不同的选择压力,造成了不同管理模式的种群间差异大于相同管理模式下的种群间差异。在我国也有关于茶园假眼小绿叶蝉种群分化的初步研究,采用RAPD分子标记技术,探讨了我国7个省份的茶园假眼小绿叶蝉的遗传差异及其亲缘关系[4]。
基于前人的相关成果,本研究采用线粒体CO I基因全长序列,对我国主要产茶省份的茶园假眼小绿叶蝉不同地理种群的遗传多样性、遗传分化程度、基因流水平及分子变异进行了解析,建立了单倍型系统发育树和进化网络图。依据几十年来的茶园叶蝉类发生和为害观察、标本收集及鉴定,茶园中约有10余种叶蝉,但优势种是假眼小绿叶蝉,其个体数占茶园叶蝉个体数的99.99%以上[27],这个百分率很稳定,不随时间、地点的变化而改变,本研究的供试虫态都是成虫,供试标本采集时间从2011年10月至2013年10月,采到的标本立即浸入无水乙醇中,数小时之后就保存于-20℃备用,可尽量消除采集时间因素对于试验结果的影响。在13个种群176个个体中共发现突变位点113个,核苷酸替换以转换为主。突变位点在整个区段内均有分布,但不同位置的进化速率有所不同。共产生105种单倍型,具有较高的单倍型多态性。其中单倍型Hap5为共享单倍型,这种单倍型可认为是能够适应环境变化并在种群中稳定存在的优势单倍型,该单倍型很有可能就是祖先单倍型[28, 29];在所有的单倍型中,有84种是独享单倍型,是由单个种群所特有的。各地理种群内具有较为丰富的单倍型多样性,表明茶园假眼小绿叶蝉对于不同环境具有较强的生存能力,这与假眼小绿叶蝉广泛存在于各种茶园环境中的情况一致。从单倍型系统发育树来看,单倍型的分布与其所在的地理单元之间不存在明显的对应关系。Tajima′s D检验是基于种内多态性的一种中性检验方法,可反映出物种种群变化动态的历史。统计值为正值时说明序列进化方式为平衡选择,且有一些单倍型分化;负值时为负向选择,种群的扩张存在瓶颈效应。本实验中总群体和各个地理种群的检测结果均不显著,说明茶园假眼小绿叶蝉种群的进化过程遵循中性进化模型,过去没有出现群体扩张和持续性增长,群体大小保持相对稳定[17]。固定系数Fst是一种种群间遗传距离的测度参数[30],从本实验的Fst值上看不同地理种群之间存在一定程度的遗传分化。基因流即群体间基因交流,是种群遗传结构均质化的主要因素之一,具有高水平基因流的种群间往往比具有有限基因流的种群间的遗传分化程度小[31]。本实验中总基因流Nm为4.097>4,当Nm>4时,种群间的基因交流比较充分,均质化作用足以抵制遗传漂变的作用,一定程度上抵消种群间的遗传分化[32]。通过AMOVA分子变异分析可以看出,假眼小绿叶蝉的遗传分化主要来自于种群内部,种群间的差异较小。通过Mnatel检验可知,不同种群之间的遗传分化程度与地理距离没有显著关系。
综上所述,认为假眼小绿叶蝉种群的进化过程遵循中性进化模型,群体大小保持相对稳定,这与茶园中假眼小绿叶蝉每年保持高发生量的情况相吻合,因为许多茶区每年假眼小绿叶蝉发生时间、发生量和施药次数基本不变,每年都是10多次。假眼小绿叶蝉种群间存在一定程度的遗传分化,这是因为我国幅员广大,不同茶园所在的自然地理条件存在较大差异,不同地理种群之间的假眼小绿叶蝉可能存在着生物学和生态学特征的差异而有一定遗传分化。而较为充分的基因交流带来的均质化作用,在一定程度上抵消了种群间的遗传分化。种群间假眼小绿叶蝉充分的基因交流主要源于假眼小绿叶蝉自然迁移和人为传播两方面的原因。假眼小绿叶蝉成虫本身具有一定的飞行扩散能力,能适应各种地理环境,这种近距离的扩散行为一直在进行中,能够引起不同种群间一定程度的基因交流。相比于茶园假眼小绿叶蝉的自然迁移,人为助迁的作用十分突出。在许多茶人们的印象中,20世纪50年代省际茶区之间假眼小绿叶蝉的体色等特征是有差异的,可能意味着大尺度的自然地理景观条件下该叶蝉种群存在生态型的差异。然而,20世纪90年代以后,省内茶区之间、省际之间茶树鲜叶的异地加工十分活跃,帮助了茶园假眼小绿叶蝉的大规模迁移,比如,近些年来从早春至深秋,黔、川、渝、皖南、豫南茶区鲜叶源源不断地调往浙、苏茶区加工以获得高价出售,这些鲜叶夹带了大量的假眼小绿叶蝉成虫、若虫和卵,它们随着茶树鲜叶在1—3日内迁移几百—几千km;闽南、闽北茶区茶苗不断调往浙、苏南、皖南、鲁南和豫南等茶区,浙西茶区的安吉白茶茶苗持续调往周边各省茶区。到达新区的假眼小绿叶蝉很快适应了当地茶园环境,经过几百代的世代更替,使得假眼小绿叶蝉不同地理种群生态型的差异逐渐消失。这也解释了为何假眼小绿叶蝉的遗传分化主要来自于种群内部、种群间的差异较小、以及种群之间的遗传分化程度与地理距离没有显著关系。
需要一提的是:来自西南边陲的云南勐海种群(YNMH)在所有种群中单倍型多样性Hd最小,与其他种群的遗传分化程度最大,与其他种群之间的遗传距离也最大,而除云南以外其他种群之间的差异比较一致。这说明了云南的茶园假眼小绿叶蝉相比较于其他种群来说处于一个相对封闭的环境中,与其他种群基因交流较小。这是因为云南的茶树多为乔木型、半乔木型,茶园地理环境、气候和植被、以及茶叶内含物等显著区别于其他假眼小绿叶蝉种群所在茶区的相应因素。而且采集YNMH种群的云南的茶园处于原始森林,交通不便、人迹罕至,该假眼小绿叶蝉种群较为独立,与其他种群的交流较少。
本研究首次扩增得到茶园假眼小绿叶蝉CO I基因的全长序列,并将其应用到种群间的遗传分化研究中。与使用CO I基因的部分序列进行遗传分化分析相比,使用序列的全长包含了更丰富的信息,获得了充分的变异位点及较为全面的遗传学数据。在下一步的研究中,还需要配合其他分子标记,比如线粒体的控制区以及核基因上的分子标记,增加试验采样点及样本数量,进一步加深对中国茶园假眼小绿叶蝉种群间关系及遗传机制的研究。
| [1] | Wang Q S, Wang D F, Wu G Y. Research advances on Empoasca vitis (Göthe) in tea trees in China. Fujian Journal of Agricultural Sciences, 2013, 28(6): 615-623. |
| [2] | Wang Y G, Li L J, Ran L X, Yu X S, Wu W W, Han B Y, Li Z M. Investigation on population dynamics of Empoasca vitis (Göthe) of different planting patterns. Southwest China Journal of Agricultural Science, 2010, 23(2): 413-415. |
| [3] | Liu L F, Xu D L, Mu D, Han B Y. Analysis of the feeding behaviours of tea green leafhoppers on resistant to sensible cultivars of tea plants by electrical penetration graphy techniques. Journal of Anhui Agricultural University, 2011, 38(2): 281-286. |
| [4] | Fu J Y, Han B Y. Studies on genetic relationships among populations of Empoasca vitis (Göthe) from tea gardens in seven provinces based on RAPD analysis. Acta Agriculturae Zhejiangensis, 2007, 19(1): 11-14. |
| [5] | Johnson K B, Radcliffe E B, Teng P S. Effects of interacting populations of Alternaria solani, Verticillium dahliae, and the potato leafhopper (Empoasca fabae) on potato yield. Phytopathology, 1986, 76: 1046-1052. |
| [6] | Wang D M, Lin Y. Application of mitochondrial DNA gene order in molecular systematics of insects. Guangdong Journal of Agricultural Sciences, 2010, 37(6): 188-190. |
| [7] | Wang B X, Yang L F. Phylogenetic utilites of mitochondrial DNA sequences in the study of insect systematic. Entomological Knowledge, 2002, 39(2): 88-92. |
| [8] | Lv H J, Huang Y. Phylogenetic relationship among some groups of orthopteran based on complete sequences of the mitochondrial COI gene. Zoological Research, 33(3): 319-328. |
| [9] | Pavan-Kumar A, Gireesh-Babu P, Babu P S, Jaiswar A K, Krishna V H, Prasasd K P, Chaudhari A, Raje S G, Chakraborty S K, Krishna Gopal, Lakra W S. Molecular phylogeny of elasmobranchs inferred from mitochondrial and nuclear markers. Molecular Biology Reports, 2014, 41(1): 447-457. |
| [10] | Zanol J, Halanych K M, Struck T H, Fauchald K. Phylogeny of the bristle worm family Eunicidae (Eunicida, Annelida) and the phylogenetic utility of noncongruent 16S, CO I and 18S in combined analyses. Molecular Phylogenetics and Evolution, 2010, 55(2): 660-676. |
| [11] | Palomera V, Bertin S, Rodríguez A, Bosco D, Virla E, Moya-Raygoza G. Is there any genetic variation among native mexican and argentinian populations of Dalbulus maidis (Hemiptera: Cicadellidae)? Florida Entomologist, 2012, 95(1): 150-155. |
| [12] | luemel J K, Andrew K R, Virant-Doberlet M, Symondson W O C. Primers for identification of type and other archived specimens of Aphrodes leafhoppers (Hemiptera, Cicadellidae). Molecular Ecology Resources, 2011, 11(5): 770-774. |
| [13] | Thompson J D, Gibson T J, Plewniak F, Jeanmougin F, Higgins D G. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 1997, 25(24): 4876-4882. |
| [14] | Tamura K, Dudley J, Nei M, Kumar S. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4. 0. Molecular Biology and Evolution, 2007, 24(8): 1596-1599. |
| [15] | Polzin T, Daneshmand S V. On Steiner trees and minimum spanning trees in hypergraphs. Operations Research Letters, 2003, 31(1): 12-20. |
| [16] | Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data. Bioinformatics, 2009, 25(11): 1451-1452. |
| [17] | Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics, 1989, 123(3): 585-595. |
| [18] | Excoffier L, Laval G, Schneider S. Arlequin (version 3. 0): an integrated software package for population genetics data analysis. Evolutionary Bioinformatics Online, 2007, 1: 47-50. |
| [19] | Peakall R, Smouse P E. GENALEX 6: genetic analysis in Excel. Population genetic software for teaching and research. Molecular Ecology Notes, 2006, 6(1): 288-295. |
| [20] | Song N, Liang A P. The complete mitochondrial genome sequence of Geisha distinctissima (Hemiptera: Flatidae) and comparison with other hemipteran insects. Acta Biochimica et Biophysica Sinica, 2009, 41(3): 206-216. |
| [21] | Bengtsson-Palme J, Ryberg M, Hartmann M, Branco S, Wang Z, Godhe A, De Wit P, Sánchez-García M, Ebersberger I, de Sousa F, Amend A, Jumpponen A, Unterseher M, Kristiansson E, Abarenkov K, Bertrand Y J K, Sanli K, Eriksson K M, Vik U, Veldre V, Nilsson R H. Improved software detection and extraction of ITS1 and ITS2 from ribosomal ITS sequences of fungi and other eukaryotes for analysis of environmental sequencing data. Methods in Ecology and Evolution, 2013, 4(10): 914-919. |
| [22] | Liu J, Liang A P. The complete mitochondrial genome of spittlebug Paphnutius ruficeps (Insecta: Hemiptera: Cercopidae) with a fairly short putative control region. Acta Biochimica et Biophysica Sinica, 2013, 45(4): 309-319. |
| [23] | Simon C, Frati F, Beckenbach A, Crespi B, Liu H, Flook P. Evolution, weighting, and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers. Annals of the Entomological Society of America, 1994, 87(6): 651-701. |
| [24] | Frati F, Simon C, Sullivan J, Swofford D L. Evolution of the mitochondrial cytochrome oxidase II gene in Collembola. Journal of Molecular Evolution, 1997, 44(2): 145-158. |
| [25] | Slatkin M. A measure of population subdivision based on microsatellite allele frequencies. Genetics, 1995, 139(1): 457-462. |
| [26] | Saha D, Mukhopadhyay A, Bahadur M. Genetic diversity of Empoasca flavescens Fabricius (Homoptera: Cicadellidae), an emerging pest of tea from sub-himalayan plantations of West Bengal, India. Proceedings of the Zoological Society, 2012, 65(2): 126-131. |
| [27] | Zhao D X, Chen Z M, Cheng J A. Belongingness of tea leafhopper dominant species. Journal of Tea Science, 2000, 20(2): 101-104. |
| [28] | Wen L Y, Zhang L X, Liu N F. Phylogenetic relationship of Perdix dauuricae inferred from mitochondrial Cytochrome b gene. Zoological Research, 2005, 26(1): 69-75. |
| [29] | Li J B, Ren Z M. Genetic diversity among schlechtendalia chinensis individuals revealed by Cyt b sequences. Journal of Fudan University: Natural Science, 2009, 48(5): 680-686. |
| [30] | Rousset F. Genetic differentiation and estimation of gene flow from F-statistics under isolation by distance. Genetics, 1997, 145(4): 1219-1228. |
| [31] | Millar C I, Libby W J. Strategies for conserving clinal, ecotypic, and disjunct population diversity in widespread species//Genetics and Conservation of Rare Plants. New York: Oxford University Press, 1991: 149-170. |
| [32] | Whitlock M C, McCauley D E. Indirect measures of gene flow and migration: FST≠1/(4Nm+1). Heredity, 1999, 82(2): 117-125. |
| [1] | 王庆森, 王定锋, 吴光远. 我国茶树假眼小绿叶蝉研究进展. 福建农业学报, 2013, 28(6): 615-623. |
| [2] | 汪云刚, 李良静, 冉隆珣, 玉香甩, 吴文伟, 韩宝瑜, 李忠美. 不同种植模式下茶假眼小绿叶蝉种群动态的调查. 西南农业学报, 2010, 23(2): 413-415. |
| [3] | 刘丽芳, 徐德良, 穆丹, 韩宝瑜. EPG 技术分析不同品种茶树抗假眼小绿叶蝉取食行为的差异. 安徽农业大学学报, 2011, 38(2): 281-286. |
| [4] | 付建玉, 韩宝瑜. 七省茶园假眼小绿叶蝉的 RAPD 分析及其亲缘关系探讨. 浙江农业学报, 2007, 19(1): 11-14. |
| [6] | 王德明, 林杨. 线粒体DNA基因序列在昆虫分子系统学研究中的应用. 广东农业科学, 2010, 37(6): 188-190. |
| [7] | 王备新, 杨莲芳. 线粒体DNA序列特点与昆虫系统学研究. 昆虫知识, 2002, 39(2): 88-92. |
| [27] | 赵冬香, 陈宗懋, 程家安. 茶小绿叶蝉优势种的归属. 茶叶科学, 2000, 20(2): 101-104. |
| [28] | 文陇英, 张立勋, 刘迺发. 以mtDNA细胞色素b基因探讨斑翅山鹑的分类地位. 动物学研究, 2005, 26(1): 69-75. |
| [29] | 李继变, 任竹梅. 角倍蚜mtDNA Cyt b 基因遗传多样性分析. 复旦学报: 自然科学版, 2009, 48(5): 680-686. |
 2014, Vol. 34
2014, Vol. 34