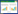文章信息
- 于基成, 刘秋, 邵阳, 刘长建, 闫建芳, 齐小辉
- YU Jicheng, LIU Qiu, SHAO Yang, LIU Changjian, YAN Jianfang, QI Xiaohui
- 大连海域沉积物中可培养海洋链霉菌活性及其多样性
- Activity and diversity of culturable Streptomyces isolated from marine sediments of sea areas of Dalian
- 生态学报, 2013, 34(20): 5896-5906
- Acta Ecologica Sinica, 2013, 34(20): 5896-5906
- http://dx.doi.org/10.5846/stxb201303240500
-
文章历史
- 收稿日期:2013-3-24
- 网络收稿日期:2014-8-2
放线菌是一类非常重要的药源微生物,目前已发现的数万种微生物来源的生物活性物质中,约有45%是由放线菌产生的[1]。海洋环境所具有的高盐度、高压、低温和低营养等特点,因此海洋放线菌具有不同于陆生放线菌的种群特点和独特的代谢途径,使其成为新型放线菌和放线菌代谢产物的重要新资源[2, 3]。自从1926 年Aronson 首次描述了海洋放线菌Mycobacterium marinum 新种开始,人们陆续从海洋环境中分离筛选到多种海洋放线菌[4]。但筛选到的海洋放线菌仍和陆地放线菌相似,以Streptomyces、Micromonospora和Rhodococcus 数量最多。尤其是海洋链霉菌,仍是海洋放线菌中的主要类群。近年来的研究也表明,从海洋放线菌中发现结构新颖并具有强生理活性的物质已达100多个,其中90%以上产生于链霉菌属[5]。但目前对海洋放线菌活性的研究多集中筛选新型放线菌种群或针对某一菌株进行活性代谢产物的分离纯化、结构分析以及功能性评价等,以期获得新型化合物。但对海洋放线菌整体活性的统计分析报道较少。本文以海洋链霉菌作为研究对象,分别以植物病原真菌、革兰氏阴性和阳性细菌作为指示菌,通过对海洋链霉菌活性分析,拟分析海洋链霉菌的抑菌活性特点,为目标性开发利用海洋放线菌资源奠定基础。
1 材料与方法 1.1 样品的采集于大连的泊石湾、王家庄、杏树屯、小窑湾、大连湾、拉树房、毛莹子、庄河花园村、龙王塘西村附近海域,采用抓斗式采样器采集海洋沉积物样品。采集后的样品用冰盒保存并立即带回实验室进行海洋放线菌菌株的分离。
1.2 抗菌活性筛选指示菌及土壤链霉菌分别以大肠杆菌(Escherichia coli),金黄色葡萄球菌(Staphyloccocus aureus)和尖孢镰刀菌(Fusarium oxysporum F. sp. cucumarinum)作为指示菌。所有指示菌均来自于大连民族学院生命科学学院放线菌资源与应用实验室。试验所用土壤链霉菌(表 1)菌株编号分别为H86、K1、LQ1、LQ23、LQ37、LQ69、 LQ137、M3、M5、V6、R11、高30和高F-9,均为大连民族学院生命科学学院放线菌资源与应用实验室分别从温室土壤和大田作物土壤分离筛选及鉴定获得。
1.3 分离培养基及海洋放线菌的分离以常用的陆生和海洋放线菌分离培养基作为基础培养基[6, 7, 8],并进行适当的改良。各种培养基的配方见表 1。分别称取各海洋沉积物样品5 g,转入装有45 mL无菌天然海水的150 mL三角瓶中,将三角瓶置55 ℃水浴锅中加热6 min。然后采用稀释分离法分别涂布于9种培养基上。28 ℃下培养5—15 d。根据菌落形态,挑选类似链霉菌的菌株,分别初步统计各培养基中分离获得的链霉菌菌株数目。
1.4 放线菌16SrRNA基因序列分析根据形态差异,对分离获得的放线菌通过16S rRNA基因序列分析进行初步鉴定。采用微波法提取分离到的放线菌基因组DNA,PCR扩增后,进行16S rRNA基因序列分析。所用引物为细菌通用引物[9],由上海生工合成。F27: 5′-AGAGTTTGATCCT GGCTCAG-3′;R1492: 5′-TACCTTGTTACGAC TT-3′。 1.5 可培养海洋放线菌的抗菌活性筛选 将分离的海洋放线菌分别接种于用天然海水和蒸馏水配制的高氏Ⅰ号培养基,28 ℃培养10—14 d(不同菌株的培养时间不同),用打孔器切下直径约为1 cm菌饼,测定其抑菌活性[10]。细菌在37℃培养24h,真菌在28 ℃培养72 h后,观察并测量抑菌圈直径的大小,进行记录。
1.6 海洋链霉菌的活性比较 1.6.1 不同种海洋链霉菌的活性分析参照1.5抗菌活性测定方法,比较海洋链霉菌的抑菌活性。
| 培养基名称Medium name | 培养基组分Medium components |
| VB母液:维生素B1、核黄素、烟酸、维生素B6、肌醇、泛酸、对氨基苯酸各0.1 g,生物素0.05 g,去离子水定容至100 mL;微量盐溶液:FeSO4 ·7H2O 、MnCl2· 4H2O、 ZnSO4各0.1 g,去离子水定容至100 mL | |
| 高氏Ⅰ号培养基Gause No.1 medium (GM) | 可溶性淀粉 20 g,KNO3 1 g,MgSO4·7H2O 0.5 g,FeSO4 0.01 g,琼脂 20 g,天然海水定容至 1000 mL,pH 7.2 |
| 甘油精氨酸培养基Glycerol arginine medium (M2) | 甘油 5 mL,精氨酸 0.5 g,K2HPO4 1 g,MgSO4 0.5 g,琼脂 18 g,天然海水定容至1000 mL,pH 7.2 |
| M13培养基M13 medium | 酵母浸出粉 4 g,可溶性淀粉 15 g,K2HPO4 1 g,MgSO4 0.5 g,琼脂 18 g,天然海水定容至1000 mL,pH 7.2 |
| 燕麦琼脂培养基Oat agar medium (OM) | 燕麦粉 20 g,微量盐溶液1 mL,琼脂18 g,天然海水定容至1000 mL,pH7.2 |
| 葡萄糖天冬氨酸培养基Glucose aspartic acid medium (GAM) | 葡萄糖 10 g,天冬氨酸0.5 g,K2HPO4 0.5 g,琼脂 15 g,天然海水定容至1000 mL,pH 7.2 |
| 腐殖酸维生素培养基 Humic acid vitamin medium (HV-1) | 腐殖酸 1.0 g,CaCO3 0.02 g,Na2HPO4 0.5 g,MgSO4·7H2O 0.5 g,KCl 1.7 g,FeSO 4·7H2O 0.01 g,VB母液1 mL,天然海水定容至1000 mL,pH 7.2 |
| 改良腐殖酸维生素培养基Improved humic acid vitamin medium (HV-2) | 虾皮壳 2 g,腐殖酸 1 g,CaCO3 0.02 g,Na2HPO4 0.5 g,MgSO4·7H2O 0.5 g,KCl 1.7 g,FeSO 4·7H2O 0.01 g,VB母液1 mL,天然海水定容至1000 mL,pH 7.2 |
| 改良LSE-SE-1培养基Improved LSE-SE medium-1 (LSE-SE-1) | 虾皮壳 2 g,CaCO3 0.02 g,Na2HPO4 0.5 g,MgS04·7H2O 0.5 g,KCl 1.7 g,FeSO 4·7H2O 0.01 g,VB母液1 mL,天然海水定容至1000 mL,pH 7.2 |
| 改良LSE-SE-2培养基Improved LSE-SE medium-2 (LSE-SE-2) | 虾皮壳 2 g,豆饼粉 0.2 g,CaCO3 0.02 g,Na2HPO4 0.5 g,MgSO4·7H2O 0.5 g,KCl 1.7 g,FeSO 4·7H2O 0.01 g,VB母液1 mL,天然海水定容至1000 mL,pH 7.2 |
将有抗菌活性的放线菌的16S rDNA PCR扩增产物测序,测序结果提交到NCBI的GeneBank数据库,并利用Blast软件和MEGA软件构建活性海洋放线菌及其相似种的系统发育树,分析具有活性海洋放线菌种群多样性。同时,利用SciFinder物质数据库查询同源种的生物活性物质情况,分析获得的海洋链霉菌潜在的生物活性物质。
1.7 同种海洋链霉菌与土壤链霉菌的活性比较按照1.5抗菌活性测定方法,比较本实验分离到的海洋链霉菌和来自土壤的同种链霉菌的抑菌活性。同时以50%海水配制的高氏Ⅰ号培养基培养土壤链霉菌后,以同种的海洋链霉菌为对照,比较其抑菌活性。分析相同培养条件下同种不同来源的链霉菌的抑菌活性。
2 结果与分析 2.1 不同培养基对海洋放线菌的分离效果利用9种培养基共分离获得放线菌165株(图 1)。结果表明,分离效果较好的培养基是改良LSE-SE -2培养基(LSE-SE-2)和燕麦琼脂培养基(OM)。但细菌在这两种培养基上生长迅速,影响后期放线菌的分离纯化。培养基甘油精氨酸培养基(M2)中有放线菌偏好的两种营养成分甘油和精氨酸,分离效果也较好,且污染较少。培养基改良腐殖酸维生素培养基(HV-2)分离的放线菌数目较LSE-SE-2少,但培养基受污染程度小,放线菌的检出率最高,是一种高效的分离培养基。高氏Ⅰ号培养(GM)、葡萄糖天冬氨酸培养基(GAM)和腐殖酸维生素培养基(HV-1)也获得一定量的放线菌种群。HV-1、M13培养基上只分离到了少量海洋放线菌。因此,HV-2和M2是两种高效分离海洋放线菌的培养基,其次为LSE-SE-2和OM培养基。
|
| 图 1 不同培养基对海洋放线菌的分离效果 Fig. 1 The separation effect for marine actinomyces on different medium 图中字母均为各培养基名称简写,如GM为高氏Ⅰ号培养基名称简写,其余与表 1所列培养基一致 |
根据形态特征及其放线菌的16S rDNA 基因序列分析,经排重,共获得165株放线菌,其中7株为拟诺卡氏菌(Nocardiopsis sp.),其余158株全部为链霉菌,部分菌株NCBI登录号为HQ696525—HQ696549,HM007148—HM007161,HM209312—HM209318。
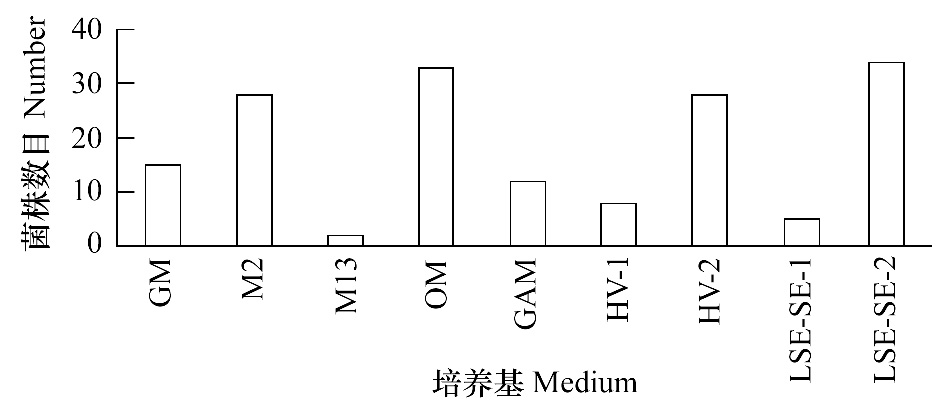
2.3 可培养海洋放线菌的抗菌活性分析采用琼脂块法对分离到的海洋放线菌分别进行抗金黄色葡萄球菌、大肠杆菌以及尖孢镰刀菌活性试验,结果表明,分离获得的158株海洋链霉菌中,95株菌株分属不同的链霉菌种,且表现不同的抗菌活性。不同种的链霉菌中,85株放线菌对金黄色葡萄球菌具有抗菌活性,占总分菌数的51.5%;其中抑菌圈直径大于30 mm的菌4株。27株放线菌对大肠杆菌具有抗菌活性,占总分菌数的16.4%。其中抑菌圈直径为27 mm的菌株编号分别为YH6和YH21,对尖孢镰刀菌有活性的菌株分别为M95、M108、HV14、HL9、HE16、HE66,仅占总分菌数的3.6%。由图 2可见,海洋放线菌更多地显示抗金黄色葡萄球菌的活性,且高活性菌株也多表现抗金黄色葡萄球菌。另外,27株表现抗大肠杆菌活性的菌株中有23株同时表现抗革兰氏阳性菌活性,6株抗尖孢镰刀菌活性的菌株也均同时具有抗革兰氏阳性菌活性。
|
| 图 2 海洋放线菌对金黄色葡萄球菌、大肠杆菌以及尖孢镰刀菌抑菌活性比较 Fig. 2 Antagonism comparison of marine actinomyces against F. oxysporium f sp. cucumarinum, S.aureus and E. coli |
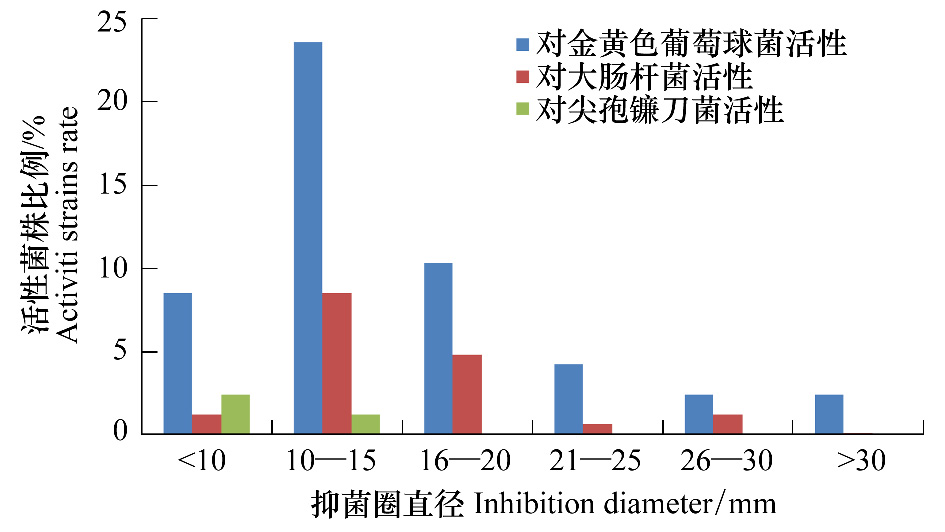
以上述拮抗放线菌及再利用Blast软件从GenBank,EzTaxon server 2.1数据库中搜索出的亲缘关系近的已报道菌株为基础,分别构建不同拮抗活性菌株的系统发育树,分析拮抗海洋放线菌多样性(图 3和图 4)。可以看出,拮抗海洋放线菌涵盖链霉菌各个类群如粉红孢类群、金色类群、白孢类群、烬灰类群、黄色类群蓝色类群、淡紫灰类群、青色类群、灰红紫类群、灰褐类群和绿色类群,而7种拟诺卡氏菌独立成支。

|
| 图 3 拮抗大肠杆菌的海洋放线菌种群多样性分析 Fig. 3 Species diversity analysis of antagonistic marine actinomyces against E. coli 图中字母+数字如M10、YH21等为分离的海洋放线菌菌株编号 |
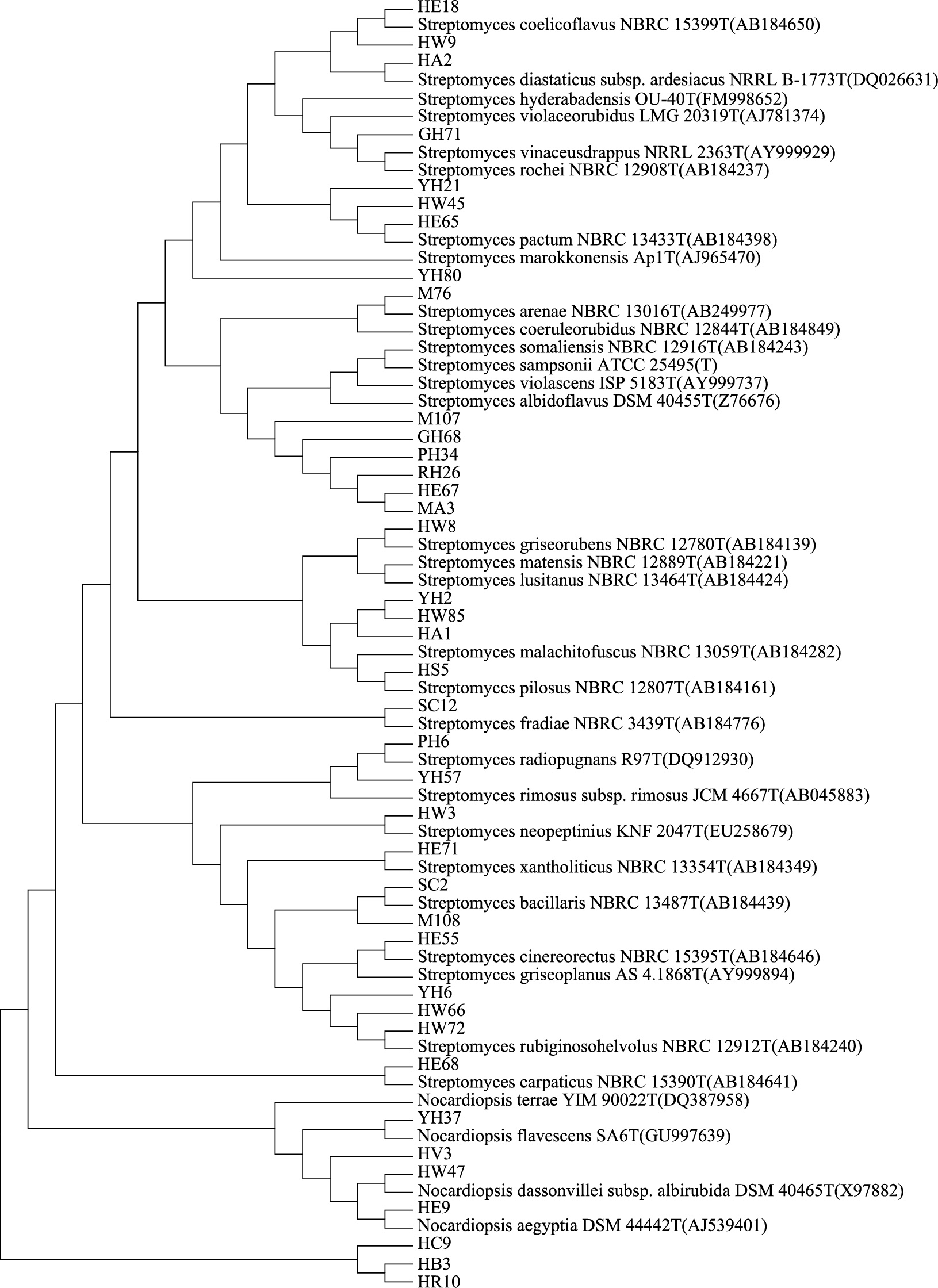
|
| 图 4 拮抗金黄色葡萄球菌的放线菌多样性分析 Fig. 4 Species diversity analysis of antagonistic marine actinomyces against S.aureus |
同时,根据各分离菌株的16S rDNA序列及其生理生化特性,确定各分离菌株的同源近似种(表 2)。结合分离菌株对革兰氏阴性细菌、阳性细菌和植物病原真菌的抑菌活性测定结果,比较SciFinder物质数据库报道的各同源近似种的生物活性。结果表明,95株分离菌株中具有抑菌活性,而其同源近似种未见活性报道的有24株。在24株分离菌株中,其中23株均表现抑制金黄色葡萄球菌活性,且10株同时表现抑制大肠杆菌活性;菌株PH26仅表现抑制大肠杆菌活性,菌株M95、HE66同时表现抑制尖孢镰刀菌活性,HV14对3种指示菌均表现抑菌活性(表 2)。在95株分离菌株中,其近似同源种报道具有生物活性,而分离菌株未检测出相应活性的有13株,近似同源种报道的具有抑制革兰氏阴性菌活性,而分离菌株未检出相应活性的9株,同源近似种报道的具有抑制丝状真菌活性,而分离菌株未检出相应活性的2株(HE11、HE71)。
| 菌株名称Strain name | 分离菌株的抑菌活性*Antagonism activity of isolates | 同源近似种Homologous species | 相似率/%Similarityrate | 同源种的生物活性*Bioactivity of homologous species | ||
| Es△ | St | Fu | ||||
| 表中数据为3次重复实验,抑菌圈直径平均值; 表示没有抑菌活性;*同源种的生物活性由SciFinder物质数据库查询,网址:http://scifinder.cas.org/ ;#分离菌株具有抑菌活性,而其同源近似种未见活性报道的菌株;●近似同源种报道具有生物活性,而分离菌株未检测出相应活性的菌株;△ Es表示各菌株对大肠杆菌(E. coli)的抑菌活性;St表示各菌株对金黄色葡萄球菌(S.aureus)的抑菌活性;Fu表示对尖孢镰刀菌(F. oxysporum F sp. cucumarinum)的抑菌活性 | ||||||
| HA1 | 30 | S.malachitofuscus | 99.156 | 抑制金黄色葡萄球菌 | ||
| HA2 | 39 | S.diastaticus | 100.000 | 拮抗性有限,轻度抑制芽孢杆菌 | ||
| HA5 | 37 | S.matensis | 98.875 | 产生抗革兰氏阳性菌的马塔霉素(Matamycin) | ||
| HA9● | S.corchorusii | 100.00 | 抑制真菌、革兰氏阳性菌 | |||
| HA14 | S.mexicanus | 99.697 | 未见报道 | |||
| HA15 | S.tricolor | 99.789 | 未见报道 | |||
| HB2 | S.coerulescens | 99.472 | 抑制小球藻 | |||
| HB3# | 10 | S.sampsonii | 99.699 | 未见活性报道 | ||
| HB12● | S.violaceoruber | 100.00 | 抑制革兰氏阳性菌、阴性菌 | |||
| HB13# | 11 | S.sannanensis | 99.328 | 未见活性报道 | ||
| HB14● | S.toxytricini | 99.925 | 抑制金黄色葡萄球菌、枯草杆菌、大肠杆菌、白色假丝酵母、颗粒青霉 | |||
| HB21● | S. flavotricini | 100.00 | 强烈抑制革兰氏阳性菌、阴性菌、酵母菌和丝状真菌 | |||
| HC5 | S.ciscaucasicus | 100.000 | 未见报道 | |||
| HC7● | S.atrovirens | 98.140 | 抑制革兰氏阳性菌 | |||
| HC9# | 22 | S.coerulescens | 99.925 | 抑制小球藻 | ||
| HC10 | S.violaceochromogenes | 100.00 | 抑制革兰氏阳性菌和大肠杆菌 | |||
| HC12 | 20 | S.flavotricini | 98.883 | 强烈抑制革兰氏阳性菌、阴性菌、酵母菌和丝状真菌 | ||
| HC13● | S.collinus | 99.925 | 产生抑制革兰氏阳性菌的覆盖霉素(Collinomycin) | |||
| HR10 | 13 | 24 | S.venezuelae | 100.000 | 多种活性,产生氯霉素(Chloramphenicol)、核酸霉素(Rinamycin)、去氧核酸霉素(Derinamycin)、双氢苦霉素(Dihydropicromycin)和莱马杀菌素(Lemacidin) | |
| HR13● | S.violaceoruber | 100.000 | 抑制革兰氏阳性菌、阴性菌 | |||
| HS1 | S.violaceorubidus | 99.851 | 未见报道 | |||
| HS5 | 16 | S.pilosus | 100.00 | 具有拮抗作用 | ||
| YH2 | 18 | S.cyaneofuscatus | 99.225 | 抑制革兰氏阳性细菌 | ||
| YH6 | 27 | 17 | S.griseoplanus | 99.857 | 抑制革兰氏阳性\\阴性细菌和肉瘤,产生新碱性抗生素SF-1739 | |
| YH17 | 17 | S.lateritius | 98.967 | 强抑制革兰氏阳性细菌 | ||
| YH21# | 27 | 32 | S.hyderabadensis | 99.033 | 未见活性报道 | |
| YH23 | 17 | 29 | S.afghaniensis | 99.364 | 产生抑制革兰氏阳性菌的泰东霉素(Taitomycin) | |
| YH37# | 12 | 21 | N. flavescens | 100.00 | 未见活性报道 | |
| YH66# | 11 | S.phaeogriseichroma-togenes | 97.994 | 未见活性报道 | ||
| YH58 | 14 | S.resistomycificus | 99.152 | 产生抑制革兰氏阳性细菌、分枝杆菌的拒霉素(Resistomycin) | ||
| YH68 | 17 | 11 | S.rimosus | 99.820 | 广谱活性,产生土霉素(Oxytetracycline)、龟裂杀菌素(Rimocidin)、巴龙霉素(Paramomycin)、中性霉素(Neytramycin)、抗生素K-16 | |
| YH73 | 10 | S.griseoaurantiacus | 99.489 | 抑制革兰氏阳性菌、分枝杆菌和德巴利氏酵母 | ||
| YH80 | 12 | S.coerulescens | 99.864 | 抑制小球藻 | ||
| YH91 | 9 | S.diastaticus | 99.142 | 拮抗性有限,轻度抑制芽孢杆菌 | ||
| M53 | 10 | S.finlayi | 99.855 | 对藤黄八叠球菌和某些放线菌有效 | ||
| M8# | 11 | 13 | S.bacillaris | 98.947 | 未见活性报道 | |
| M10 | 11 | 16 | S.violascens | 99.146 | 对金黄色葡萄球菌、大肠杆菌十分微弱且不稳定的活性 | |
| M71 | 9 | S.ramulosus | 99.680 | 产生醋霉素(Acetomycin),抑制革兰氏阳性菌、阴性菌、毛滴虫和阿米巴 | ||
| M76 | 10 | 23 | S.arenae | 97.863 | 抑制结核杆菌 | |
| M77# | 17 | 11 | S.radiopugnans | 98.892 | 未见活性报道 | |
| M95# | 8 | 8 | S.specialis | 99.155 | 未见活性报道 | |
| M105 | 12 | S.vinaceusdrappus | 99.272 | 产生友霉素(Amicetin),抑制革兰氏阳性菌、分枝杆菌 | ||
| M107 | 7 | S.griseosporeus | 99.174 | 产生泰东霉素,抑制革兰氏阳性菌 | ||
| M108 | 16 | 8 | S.laurentii | 98.856 | 产生硫链菌素(Thiostrepton),抑制革兰氏阳性菌、分枝杆菌 | |
| M109 | 16 | 7 | S.luteogriseus | 99.928 | 产生对Hela细胞有毒性的佩里霉素(Peliomycin),有报道对革兰氏阳性菌有作用 | |
| MH6 | 17 | 34 | S.lydicus | 99.568 | 产生利迪链菌素(Streptolydigin),抑制革兰氏阳性、阴性、真菌、滴虫和抗真菌的利迪霉素(Lydimycin) | |
| MH28 | N. dassonvillei | 97.445 | 未见活性报道 | |||
| GH1 | 13 | S.turgidiscabies | 99.331 | 引起植物病害 | ||
| GH27# | 10 | S.haliclonae | 99.398 | 未见活性报道 | ||
| GH31 | 16 | S.capillispiralis | 97.366 | 产生头孢菌素C-4 | ||
| GH68 | 8 | S.bellus | 99.378 | 产生抗革兰氏阳性菌的马塔霉素(Matamycin) | ||
| GH69 | S.asterosporus | 98.796 | 未见活性报道 | |||
| GH71 | 17 | S.rochei | 99.481 | 拮抗作用广泛,产生疏螺菌素(Borrelidin)、抗生素T-2636、束菌素(Bundin)、兰卡杀菌素(Lankacidin) | ||
| GH72 | 9 | S.scabiei | 98.575 | 引起植物病害 | ||
| GH74● | S.lincolnensis | 97.837 | 抑制革兰氏阳性、阴性细菌,产生林肯霉素(Lincomycin)、郎西纳霉素(Rancinamycins) | |||
| PH6# | 11 | 25 | S.radiopugnans | 98.822 | 未见活性报道 | |
| PH26# | 16 | N. dassonvillei | 97.516 | 未见活性报道 | ||
| PH33 | 22 | 28 | S.parvus | 97.486 | 产生抗革兰氏阳性细菌和肿瘤的放线菌素(Actinomycin) | |
| PH34 | 16 | S.ambofaciens | 97.937 | 抑制革兰氏阳性、阴性细菌、原虫、真菌和肿瘤,产生螺旋霉素(Spiramycin)、刚果杀锥虫素(Congocidin)、纺锤菌素(Netropsin)、偶氮霉素(Duazomycin)和偶氮亮氨酸(DON) | ||
| RH26 | 26 | S.anulatus | 99.711 | 抑制革兰氏阳性、阴性细菌和真菌 | ||
| HV3# | 11 | 13 | N. terrae | 98.552 | 未见活性报道 | |
| HV14# | 11 | 10 | 9 | N. dassonvillei | 98.657 | 未见活性报道 |
| HW3# | 12 | S.neopeptinius | 99.182 | 未见活性报道 | ||
| HW6 | 14 | S.griseorubens | 99.634 | 抑制革兰氏阳性细菌和颗粒青霉,对阴性细菌和白色假丝酵母无作用 | ||
| HW8 | 17 | S.tauricus | 98.804 | 对革兰氏阳性细菌微弱作用,在菌丝体内产生抑制肿瘤的公牛菌素(Tavromycetin) | ||
| HW9 | 10 | S.violaceolatus | 100.00 | 对部分链霉菌微弱作用 | ||
| HW16 | 13 | S.cuspidosporus | 98.254 | 产生抑制革兰氏阳性、阴性细菌以及肿瘤的稀疏菌素(Sparsomycin)和抑制分枝杆菌的杀结核菌素(Tubercidin) | ||
| HW17 | 13 | S.cinereorectus | 99.927 | 抑制革兰氏阳性和阴性细菌 | ||
| HW27 | 10 | S.fimicarius | 98.341 | 具有拮抗活性 | ||
| HW45# | 13 | 11 | S.heliomycini | 99.703 | 未见活性报道 | |
| HW47# | 10 | N. dassonvillei | 99.641 | 未见活性报道 | ||
| HW66 | 16 | S.pluricolorescens | 98.287 | 产生抑制革兰氏阳性细菌的色硫霉素(Chrothiomycin)和抑制肿瘤的多色霉素(Pluramycin) | ||
| HW72 | 11 | S.rubiginosohelvolus | 99.139 | 抑制革兰氏阳性细菌和颗粒青霉 | ||
| HW83 | 11 | S.lusitanus | 99.298 | 产生四环素(Tetracycline)和金霉素(Chlorotetracycline),抑制革兰氏阳性、阴性细菌、分枝杆菌和大型病毒 | ||
| HW85 | 13 | S.regensis | 100.00 | 产生放线菌素(Actinomycin),抑制革兰氏阳性细菌和肿瘤 | ||
| HW87# | 17 | 17 | S.marinus | 99.104 | 未见活性报道 | |
| HL9 | 11 | 12 | S.fradiae | 97.474 | 广泛抗菌活性,产生新霉素(Neomycin)、弗氏菌素(Fradicin)、泰乐菌素(Tylosin)、大环内酯类抗生素B-61269A、B-587和氨基苷菌素(Aminosidine) | |
| HE9# | 8 | N. aegyptia | 98.028 | 未见活性报道 | ||
| HE11 | 13 | 13 | S.cellulosae | 98.867 | 产生抑制真菌、酵母菌的制霉色基素(Fungichromin)和制霉色质素(Fungichromatin)和放线菌素(Actinomycin) | |
| HE16 | 9 | 16 | 9 | S.atrovirens | 98.821 | 抑制革兰氏阳性细菌 |
| HE18# | 11 | S.coelicoflavus | 97.945 | 产生淀粉酶抑制剂 | ||
| HE23 | 12 | S.rishiriensis | 99.129 | 产生香豆霉素(Coumermycin)、新核苷抗生素AT-265、枝三烯菌素(Mycotrienins),对革兰氏阳性、阴性细菌、真菌、酵母菌均有抑制作用,对肿瘤作用微弱 | ||
| HE34● | 11 | S.pristinaespiralis | 98.961 | 产生抑制革兰氏阳性细菌的原始霉素(Pristinamycims) | ||
| HE55 | 8 | S.cinereorectus | 98.922 | 具有抑制革兰氏阳性和阴性细菌活性 | ||
| HE65 | 10 | S.pactum | 98.433 | 广谱抗菌活性,产生抗肿瘤抗生素密旋霉素(Pactamycin)、密旋菌素(Pactacin)和杀虫的杀粉蝶菌素(Piericidin) | ||
| HE66# | 12 | 10 | S.thermocarboxydus | 99.561 | 未见活性报道 | |
| HE67 | 9 | S.somaliensis | 99.031 | 寄生菌,引起足菌病 | ||
| HE68# | 18 | S.carpaticus | 99.564 | 未见活性报道 | ||
| HE69 | 9 | S.avermitilis | 99.600 | 产生除虫菌素复合体,杀线虫活性 | ||
| HE71● | 13 | 14 | S.xantholiticus | 99.869 | 抗革兰氏阳性细菌、分枝杆菌、酵母菌和丝状真菌 | |
| SC2# | 16 | 17 | S.bacillaris | 99.383 | 未见活性报道 | |
| SC3# | 12 | S.carpinensis | 99.284 | 未见活性报道 | ||
| SC12 | 14 | 24 | S.fradiae | 99.905 | 广泛抗菌活性,产生新霉素(Neomycin)、弗氏菌素(Fradicin)、泰乐菌素(Tylosin)、大环内酯类抗生素B-61269A、B-587和氨基苷菌素(Aminosidine) | |
| MA2 | 10 | S.tateyamensis | 99.033 | 强抑制革兰氏阳性细菌,产生硫肽菌素(Thiopeptins) | ||
| MA3 | 7 | S.corchorusii | 99.368 | 抑制真菌、革兰氏阳性细菌,产生盐屋霉素(Siomycin) | ||
由表 3可见,9个种分别来源于土壤和海洋链霉菌的24株菌株中,其抗菌活性表现差异较大。来自海洋的链霉菌除菌株HR13外,均对金黄色葡萄球菌表现抗菌活性,分别分属链霉菌的9个种。而来自土壤的链霉菌则只有4株分属链霉菌的3个种。表现抗真菌活性的6株链霉菌均来自土壤样品,分属4个种。对大肠杆菌具有抑菌活性的12株链霉菌中,10株来自土壤,来自海洋样品的仅有2株,其中,土壤源占6个种,海洋源占2个种。研究结果进一步表明,海洋源链霉菌更多地表现为抗革兰氏阳性菌活性,土壤源链霉菌更多地表现抗真菌活性。同时,研究也表明,利用海水培养的与不利用海水培养的土壤链霉菌其活性没有差异。
| 菌株名称Strain name | 菌株编号Strain number | 抑菌活性Antagonism activity/mm | 来源Sources | ||
| 黄瓜枯萎病菌 | 大肠杆菌 | 金黄色葡萄球菌 | |||
| 表中数据为3次重复实验,抑菌圈直径平均值; :表示没有抑菌活性 | |||||
| 浅天蓝链霉菌S.coerulescens | YH80 | 12 | 海洋 | ||
| LQ137 | 11 | 温室土壤 | |||
| LQ1 | 9 | 温室土壤 | |||
| 粪生链霉菌S.fimicarius | HW27 | 10 | 海洋 | ||
| M5 | 大田土壤 | ||||
| 毛链霉菌S.pilosus | HS5 | 16 | 海洋 | ||
| V6 | 13 | 大田土壤 | |||
| 紫阔链霉菌S.violaceolatus | HW9 | 10 | 海洋 | ||
| K1 | 12 | 大田土壤 | |||
| HR13 | 海洋 | ||||
| LQ69 | 9 | 温室土壤 | |||
| R11 | 16 | 大田土壤 | |||
| 淀粉酶链霉菌S.diastaticus | HA2 | 39 | 海洋 | ||
| LQ37 | 13 | 14 | 12 | 温室土壤 | |
| 高30 | 16 | 15 | 13 | 大田土壤 | |
| 龟裂链霉菌S.rimosus | YH68 | 17 | 11 | 海洋 | |
| 高F-9 | 26 | 18 | 大田土壤 | ||
| 葡萄牙链霉菌S.lusitanus | HW85 | 13 | 海洋 | ||
| LQ123 | 11 | 温室土壤 | |||
| 深红紫链霉菌S.violaceorubidus | HS1 | 海洋 | |||
| M3 | 11 | 10 | 大田土壤 | ||
| 弗氏链霉菌S.fradiae | SC12 | 14 | 24 | 海洋 | |
| HL9 | 13 | 11 | 海洋 | ||
| H86 | 12 | 8 | 大田土壤 | ||
Pisano 等[11]对海洋沉积物放线菌抗菌活性进行研究时发现,尽管拮抗放线菌的比例有的低至15.8%,有的高至73%,但均表现为主要抑制革兰氏阳性细菌,其次是真菌,对革兰氏阴性细菌的拮抗最小。Sujatha从南印度Tuticorin海岸沉积物样品中分离的64株链霉菌中,44株链霉菌表现抗细菌活性,占分离到的链霉菌株数的68.8%[12]。Zhang等对黄海海洋沉积物中24株需要海水才能生长很好的放线菌活性时发现,其中17株均具有抗细菌活性[13]。在本实验中,具有拮抗活性的菌株共有95株,占总分菌数的57.6%。其中抑制革兰氏阳性细菌的比例最高,占总分菌比例的51.5%,其次为革兰氏阴性细菌,占总分菌比例的16.4%,对真菌的抑制比例很小,只有3.6%,且活性也很低。本研究结果与Pisano的研究结果相似,即海洋链霉菌更多地表现抗革兰氏阳性细菌活性,且活性远远高于相应的土壤链霉菌。而对真菌和革兰氏阴性细菌的活性均较低,尤其对真菌活性,其数量及活性大小均远远低于土壤链霉菌。由于许多革兰氏阳性细菌多分布于海洋沉积物和海洋生物共生系统中,本研究分离获得的165株链霉菌中均来自渤海和黄海海域-20—-40 m水下海洋沉积物样品,50%以上的菌株表现抗革兰氏阳性细菌活性,可能与样品中存在大量革兰氏阳性菌有关[14]。是否这些革兰氏阳性菌株与海洋放线菌在营养或空间分布上存在竞争,而促使海洋放线菌进化为更多的表现抗革兰式阳性活性还有待进一步研究。但本文的研究结果,对于研究者更好地理解微生物多样性,科学解释微生物种群间的关系,利用共培养技术提高微生物次级代谢产物的产量,发现新型活性代谢产物以借鉴。
95株分离菌株中具有抑菌活性,而其同源近似种未见活性报道的有23株。这一结果说明,在海洋链霉菌中可能存在着大量的新型活性物质。近似同源种报道具有生物活性,而分离菌株未检测出相应活性的菌株有13株。同时,相应近似种报道具有抑制革兰氏阴性菌活性,而其对应的分离菌株则未检出相应活性。究其原因,可能是由于微生物在不同的环境条件下通过不同途径代谢不同的活性代谢产物。有关同种海洋链霉菌与土壤链霉菌活性比较结果也进一步表明,即使同一种链霉菌,由于其栖境不同,其活性也存在很大差异。同时,9个种土壤和海洋链霉菌的24株菌的活性比较也再次证明海洋链霉菌多表现抗革兰氏阳性菌活性。
3.2 活性海洋放线菌的系统发育地位链霉菌属的种群异常繁多,阎逊初先生根据国内外大量的分类鉴定资料和多年的实践经验,将链霉菌分为12个类群[15],本研究筛选的95株活性海洋放线菌涵盖除吸水类群外的所有链霉菌类群,且多表现广谱抗性。在27株表现阴性菌的活性菌株中23株同时表现抗革兰氏阳性菌,6株抗尖孢镰刀菌活性的菌株也同时抗革兰氏阳性菌,据结论可推测抗革兰氏阳性菌的活性海洋放线菌菌株表现更为广谱的抗菌活性,具备产生广谱性代谢产物的潜能,进一步说明海洋链霉菌是获得新型广谱性活性代谢产物的重要资源。
9 5株分离菌株中具有抑菌活性,而其同源近似种未见活性报道的23株分离菌株,对目标菌株均将作为新型活性化合物筛选的潜在菌株。同时,即使分离菌株的生物活性与近似同源种报道的生物活性相近,具体分析其活性也有一定的差异。如根据筛选菌株的亲缘关系分析,如图 3和表 3中M10与S.violascens亲缘关系最近(同源性99.146%),S.violascens的文献资料表明其对大肠杆菌有十分微弱且不稳定的活性[15],但菌株M10的对革兰氏阳性和阴性菌株的抑菌活性都很稳定,3次重复实验结果均表现抑菌活性。同样,菌株HA2与 S.diastaticus亲缘关系最近(100%同源性),但相关文献报道该链霉菌拮抗活性有限,只有轻度抑制枯草芽孢杆菌的活性,并未发现抑制金黄色葡萄球菌的活性[15, 16],而菌株HA2对金黄色葡萄球菌的抑菌圈直径高达39.0 mm,且活性稳定。因此,有待于对这些菌株的活性代谢产物进行进一步的研究。
| [1] | Bérdy J. Bioactive microbial metabolites. The Journal of Antibiotics, 2005, 58(1): 1-26. |
| [2] | Bull A T, Stach J E M, Ward A C, Goodfellow M. Marine actinobacteria: perspectives, challenges, future directions. Antonie Van Leeuwenhoek, 2005, 87(1): 65-79. |
| [3] | Mincer T J, Jensen P R, Kauffman C A, Fenical W. Widespread and persistent populations of a major new marine actinomycete taxon in ocean sediments. Applied and Environmental Microbiology, 2002, 68(10): 5005-5011. |
| [4] | Tian X P, Zhang S, Li W J. Advance in marine actinobacterial research-A review. Acta Microbiologica Sinica, 2011, 51(2): 161-169. |
| [5] | Lam K S.Discovery of novel metabolites from marine actinomycetes. Current Opinion in Microbiology, 2006, 9(3): 245-251. |
| [6] | Zhang H T, Lee Y K, Zhang W, Lee H K. Culturable actinobacteria from the marine sponge Hymeniacidon perleve: isolation and phylogenetic diversity by 16S rRNA gene-RFLP analysis. Antonie Van Leeuwenhoek, 2006, 90(2): 159-169. |
| [7] | Lin L, Tan Y, Chen F F, Zhou H X, Wang Y G, He W Q, Wang Y. Diversity of culturable actinomycetes in Sea deposit of Tiger beach at Bohai bay, Dalian, China. Acta Microbiologica Sinica, 2011, 51(2): 262-269. |
| [8] | He J, Zhang D F, Xu Y, Zhang X M, Tang S K, Xu L H, Li W J. Diversity and bioactivities of culturable marine actinobacteria isolated from mangrove sediment in Indian Ocean. Acta Microbiologica Sinica, 2012, 52(10): 1195-1202. |
| [9] | Xu P, Li W J, Xu L H, Jiang C L. A microwave-based method for genomic DNA extraction from actinomycetes. Microbiology, 2003, 30(4): 82-84. |
| [10] | Zhao B X, Gao Z G, Shao Y, Yan J F, Hu Y C, Yu J C, Liu Q, Chen F. Diversity analysis of typeⅠketosynthase in rhizosphere soil of cucumber. Journal of Basic Microbiology, 2011, 52(2): 224-231. |
| [11] | Pisano M A, Sommer M J, Taras L. Bioactivity of chitinolytic actinomycetes of marine origin. Applied Microbiology and Biotechnology, 1992, 36(4): 553-555. |
| [12] | Selvakumar D. Marine Streptomyces as a novel source of bioactive substances. World Journal of Microbiology & Biotechnology, 2010, 26(12):2123-2139. |
| [13] | Zhang S M, Ye L, TANG X X, Diversity and Bioactivity of Actinomycetes from Marine Sediments of the Yellow Sea. Journal of Ocean University of China (Oceanic and Coastal Sea Research), 2012, 11 (1): 59-64. |
| [14] | Lin B X, Huang Z Q, Xie L H. Advance in researches of drugs derived from marine bacteria. Acta Microbiologica Sinica, 2005, 45(4): 657-660. |
| [15] | Yan X C. Classification and Identification of Actinomyces. Beijing: Science Press, 1992: 296-299, 473, 961. |
| [16] | Seco E M, Cuesta T, Fotso S, Laatsch H, Malpartida F. Two polyene amides produced by genetically modified Streptomyces diastaticus var. 108. Chemistry & Biology, 2005, 12(5): 535-543. |
| [5] | 田新朋, 张偲, 李文均. 海洋放线菌研究进展. 微生物学报, 2011, 51(2): 161-169. |
| [7] | 林灵, 谭亿, 陈菲菲, 周红霞, 王以光, 赫卫清, 王勇. 大连渤海老虎滩海域沉积物可培养放线菌的多样性. 微生物学报, 2011, 51(2): 262-269. |
| [8] | 何洁, 张道锋, 徐盈, 张晓梅, 唐蜀昆, 徐丽华, 李文均. 印度洋红树林沉积物可培养海洋放线菌多样性及其活性. 微生物学报, 2012, 52(10): 1195-1202. |
| [9] | 徐平, 李文均, 徐丽华, 姜成林. 微波法快速提取放线菌基因组DNA. 微生物学通报, 2003, 30(4): 82-84. |
| [12] | 林白雪, 黄志强, 谢联辉. 海洋细菌活性物质的研究进展. 微生物学报, 2005, 45(4): 657-660. |
| [13] | 阎逊初. 放线菌的分类与鉴定. 北京: 科学出版社, 1992: 296-299, 473, 961. |
 2013, Vol. 34
2013, Vol. 34