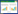文章信息
- 陈明明, 王少璞, 韦梦, 何佳颖, 郑邦旭, 裘琼芬
- CHEN Mingming, WANG Shaopu, WEI Meng, HE Jiaying, ZHENG Bangxu, QIU Qiongfen
- 象山港网箱养殖区沉积物的古菌空间分布
- The archaeal spatial distribution in sediments of cage culture area in Xiangshan Bay, Zhejiang, China
- 生态学报, 2014, 34(14): 4099-4106
- Acta Ecologica Sinica, 2014, 34(14): 4099-4106
- http://dx.doi.org/10.5846/stxb201212041745
-
文章历史
- 收稿日期:2012-12-4
- 修订日期:2014-2-27
象山港位于宁波市东南部,是一个由东北向西南向内陆嵌入的狭长型半封闭海湾,是浙江省重要的水产养殖基地[2]。网箱养殖是其中主要的养殖形式。网箱养殖是国内近些年来发展起来的一种高度集约化的养殖模式,但在带来巨大经济效益的同时,港内水域和底质环境也受到显著影响[3, 4]。研究表明,2007年象山港内海水中的硝酸盐比1992年增加了一倍,磷酸盐也有大幅度的增加[5]。目前,对象山港网箱养殖区区域环境的研究大多以水质分析为主[6],生态学相关报道也主要针对于浮游植物和底栖生物[7],对微生物尤其是古菌的研究较为缺乏[1, 8, 9]。完整且精确地描述生物体的多样性和相对丰富对于了解任何生态系统都是至关重要的[10]。
古菌作为海洋微生物的一大类群,广泛生活在各类海域环境中,包括高盐、高热等极端环境,在海洋生态系统的物质与能量循环中扮演着重要角色[11]。古菌界主要由泉古菌门(Crenarchaeota)和广域古菌门(Euryarchaeota)组成[9]。已培养的泉古菌包括暖球形菌目(Caldisphaerales)、硫还原球菌目(Desulfurococcales)、硫化叶菌目(Sulfolobales)和热变形菌目(Thermoproteales)[10]。泉古菌门中除了氨氧化古菌[11]外,大多数纯培养都属于嗜热菌。已培养的广域古菌有嗜盐菌、嗜热菌、产甲烷菌和硫酸盐还原菌[10]。其中,产甲烷古菌、氨氧化古菌、硫酸盐还原菌等都在海洋碳、氮、硫等元素循环中必不可少,也是目前研究的热点[12, 13, 14, 15]。然而,基因组的研究表明,可培养的古菌仅代表了自然界中非常小一部分的古菌多样性[16]。Lipp等的研究表明,深度在1m以下的沉积物中,古菌生物量占所有活细胞生物量的87%[17]。沉积物作为微生物和养殖废弃物的重要聚集地,直接影响着上层水质和养殖生物的健康。基于分离培养的传统微生物研究方法存在很大局限性而无法真实反映沉积物中的古菌群落结构,从而影响对海洋生态系统的正确评价[18]。磷脂脂肪酸(phospholipid fatty acid,PLFA)等化学方法又无法获得古菌的生物标志物[19]。因此,非培养的分子生物学方法是研究古菌群落结构和多样性的有效手段。以原核生物16S rRNA基因作为系统发育分析的标签[20],通过DNA序列间的差异计算出核苷酸多态性,由此构建进化关系的系统发育树,从分子水平上直观、形象地表现进化规律,是目前古菌研究的常用手段[21]。此外,末端限制性片段长度多态性(T-RFLP)、变性梯度凝胶电泳(DGGE)等技术是目前研究微生物群落结构和多样性比较普遍的方法[22]。T-RFLP技术将RFLP技术和荧光标记技术相结合后发展的一种较先进的生态学指纹分析方法,具有高灵敏度、高通量等特点,且因其易操作、能获得半定量的结果等原因,在微生物研究中被广泛应用。
本文通过T-RFLP技术与克隆测序的方法,分析古菌分布与环境因子之间的关系,比较象山港网箱养殖区与周边区域沉积物中古菌的群落结构和多样性,及其在不同深度上的分布规律,探讨象山港网箱养殖活动对沉积物中古菌群落结构和多样性的影响,为象山港网箱养殖的生态效应评价提供有用信息。
1 材料和方法 1.1 试验材料沉积物样品于2011年7月采自浙江省宁波市象山港内的3个不同区域:象山港口非养殖区(CK,29°36′11.94″N,121°46′18.00″E)、西沪港口交汇区(IS,29°33′0.66″N,121°44′10.86″E)和西沪港内网箱养殖区(AC,29°32′33.66″N,121°46′6.42″E)。采样位点的部分理化背景以及沉积物的采集方式已在前面的研究中有所描述[1]。简单地说,每个区域采集了6个不同深度的沉积物样品:0—5 cm,5—10 cm,10—15 cm,15—20 cm,20—25 cm,50—55 cm。所有样品用液氮冷冻,带回实验室-20℃保存至分析。
1.2 理化分析总磷的测定采用过硫酸钾氧化法。硅酸盐的测定采用硅钼蓝比色法(GB/T 9742—2008)。可交换铵离子浓度采用次溴酸钠氧化法测定[23],采用2 mol/L KCl溶液振荡1 h进行提取。亚硝酸盐和硝酸盐浓度采用双波长法测定[24]。温度、氧化还原电位(Eh)、pH和硫酸盐(SO2-4)的测定在前面研究中已有所描述[1]。
1.3 沉积物总DNA的提取和古菌16S rRNA基因的扩增称取0.5 g核心沉积物样品,采用MP公司土壤DNA提取试剂盒(FastDNA SRIN kit for soil),按照说明书提取沉积物总DNA,得到的总DNA最终溶于100 μL DES(试剂盒提供)中。用带FAM荧光标记的古菌16S rRNA基因通用引物[25]:109F:5′-AC(gt) GCT CAG TAA CAC GT-3′,934R-FAM:5′-FAM-AGG AAT TGG CGG GGG AGC AC-3′,取1 μL总DNA作为模板进行古菌16S rRNA基因的PCR扩增。反应体系(50 μL):5 μL10×Buffer,4 μL 2.5 mmol/L dNTP,1 μL 10 μmol/L 引物,1 μL 2.5 U Taq DNA聚合酶(TakaRa公司),1 μL模板DNA,加灭菌水至终体积50 μL,并混匀。扩增条件:94℃预变性5 min;94℃变性30 s,52℃退火45 s,72℃延伸90 s,30个循环,再72℃延伸8 min。将PCR产物经1%琼脂糖凝胶电泳检测后,用DNA纯化试剂盒(北京天根生化科技有限公司)纯化。
1.4 古菌16S rRNA基因的限制性酶切用限制性内切酶Tag I(T^CGA)对古菌16S rRNA基因PCR产物纯化产物进行酶切。反应体系(40 μL):4 μL 10×Buffer,4 μL 0.1% BSA,10 U限制性内切酶Tag I(TakaRa公司),30 μL PCR纯化产物,加灭菌水至终体积40 μL,并混匀。在65℃酶切3 h后,立即加入3×40 μL的预冷无水乙醇和0.1×40 μL的3 mol/L NaAc(pH 5.2),采用乙醇沉淀法纯化脱盐,最后溶于10 μL的灭菌水中。
1.5 末端限制性片段基因扫描取1—2 μL酶切纯化产物,与10 μL HiDi-甲酰胺和0. 2 μL内标(MapMarker 1000)混合后在95℃变性3 min,马上置于冰浴,然后通过ABI 3130 Genetic Analyzer进行限制性片段基因扫描,并由ABI GeneMap分析软件获取T-RFLP结果。
1.6 古菌16S rRNA基因克隆/测序和系统发育分析根据古菌16S rRNA基因T-RFLP分析结果,选取CK区域表层0—5 cm样品,构建古菌16S rRNA基因克隆文库。采用无荧光标记的109F/934R引物扩增,其余PCR条件同上(1. 2)。以切胶纯化后的PCR产物为模板,pMD19-T Vector(TaKaRa公司)为载体,16℃连接反应3 h。连接产物转入大肠杆菌JM109感受态细胞(TaKaRa公司),在含X-gal和IPTG的LB培养基上进行蓝白斑筛选,对筛得的古菌16S rRNA基因阳性克隆进行测序。将测得的样品序列与NCBI中的古菌16S rRNA序列进行比较,下载相关古菌序列,导入ARB-Silva的数据库中,并用ARB软件中的NJ算法(Neighbor-Joining)对本试验所得序列进行系统发育分析。所有序列已提交EMBL,序列号为HE648330—HE648377。
1.7 数据分析古菌16S rRNA基因T-RFLP图谱中每一个限制性片段(T-RF)为1个OUT(operational taxonomic unit),各末端限制性片段(T-RF)的相对峰高值(每个T-RF的峰高除以累计峰高值)作为各OUT的相对丰度,相对丰度低于0.5%的OUT不予考虑[26]。计算古菌多样性指数(H′)和均匀度指数(E′):H′=-∑PilnPi,E′= H′/lnS,式中Pi为第i个OUT的相对丰度,S为OUT总数。
取3个平行T-RF相对丰度的平均值,使用R软件的Vegan软件包[27]对T-RFLP结果进行主成分分析(PCA)。结合环境因子,使用R软件对各位点进行冗余分析(RDA)[28]。
2 结果与分析 2.1 基于T-RFLP图谱的古菌群落分析用荧光标记的古菌16S rRNA基因通用引物109F/934R扩增目的片断,Tag I(T^CGA)酶切纯化后得到的样品经末端限制性片段基因扫描得到古菌群落的T-RFLP图谱(图 1)。从图 1可以看出,古菌群落结构简单,均以T-RF188 bp所代表的古菌为主要优势种群,在各区域沉积物古菌群落中均占50%以上的比例,CK区域平均达61.2%,IS区域平均达75.9%,AC区域最高,达到80.3%。相比IS和AC区域,除了T-RF 188 bp,CK区域的其他T-RFs也占有较大的比例。T-RFs 50-100 bp所代表的古菌在CK区域0—20 cm沉积物样品中占有较高的丰度,而在IS和AC区域则很少。说明在这3个区域中,CK区域的古菌群落组成成分最复杂。
|
| 图 1 沉积物中古菌T-RFLP图谱 Fig.1 T-RFLP profiles of archaeal communities of sediments 沉积物样品所处深度已标于图谱右上角,纵向连续3张图谱来自第1张图谱所示深度3个平行位点的沉积物 |
对沉积物古菌16S rRNA基因的T-RFLP结果进行主成分分析,得到图 2。结果显示,3个区域的古菌群落结构存在一定规律,AC区域与CK区域相互独立,IS区域则介于两者之间,且与AC区域具有一定的重叠。此外,CK区域古菌群落从表层到深层差异最大,在PC1主轴上分布最广,IS区域次之,AC区域的差异则更小。T-RFs 78 bp、88 bp、 383 bp和188 bp相关古菌是引起不同区域、不同深度古菌群落差异的主要古菌。与其它3个T-RFs不同的是,T-RFs 188 bp的相对丰度随深度的增加而增加。CK区域深层样品(如:CKe和CKf)因其古菌群落结构单一,与AC区域和IS区域的古菌群落结构更为相近。

|
| 图 2 古菌16S rRNA基因T-RFLP的主成分分析 Fig.2 Principal component analysis for the archaeal 16S rRNA gene T-RFLP 图中小写字母a—f代表深度,分别为:a(0—5 cm)、b(5—10 cm)、c(10—15 cm)、d(15—20 cm)、e(20—25 cm)和f(50—55 cm) |
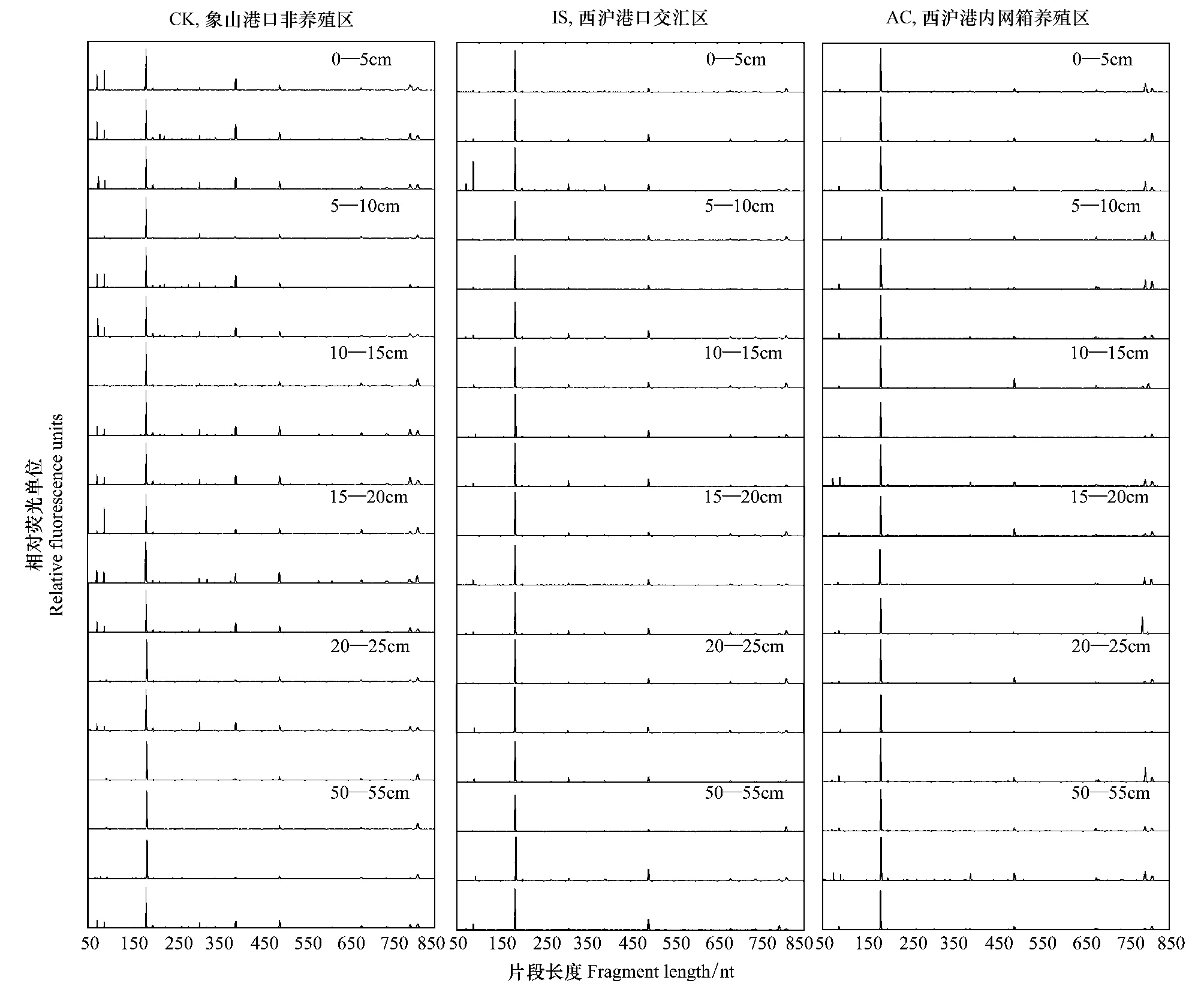
从构建好的CK区域表层0—5 cm的沉积物样品古菌16S rRNA基因文库中随机筛选出48个阳性克隆进行测序。所得样品序列用ARB软件及数据库构建古菌系统发育树(图 3),分析样品中古菌的分类地位。系统发育分析结果表明,在各区域均大量分布的T-RF 188 bp所代表的古菌属于泉古菌门(Crenarchaeota)、热变形菌纲(Thermoprotei)、热变形菌目(Thermoproteales)。泉古菌在整个克隆文库中占了50%,与所得克隆相近的古菌有厌硫球菌属(Sulfophobococcus)和热变形菌属(Thermoproteus)等以硫为能源的厌氧菌。另外50%属于广域古菌门(Euryarchaeota),包括嗜盐杆菌目(Halobacteriales)、热原体目(Thermoplasamatales)和少量的产甲烷古菌。
|
| 图 3 古菌16S rRNA基因序列的系统发育树 Fig.3 Phylogenetic relationship (neighbor-joining) of archaeal 16S rRNA gene sequences |
利用ARB软件中的NJ算法构建,黑体序列为本实验获得,并标注相应的T-RF长度;其它序列均来自NCBI,并标注相应的序列号; Group SdAr表示序列非常接近的组,括号中的值表示该组的序列数量
2.3 基于T-RFLP图谱的古菌多样性分析基于古菌16S rRNA基因T-RFLP的末端限制性片段长度的数目及其相对丰度,计算了各区域不同深度沉积物中古菌的Shannon多样性指数H′和均匀度指数E′(表 1)。总体上,同一区域内,古菌多样性指数和均匀度指数随深度呈下降趋势,但平行之间差异较大,无显著差异。同一深度上,AC区域的多样性指数和均匀度指数均低于CK和IS区域。多样性越低,稳定性相对越高。因而,不同深度沉积物古菌群落结构稳定性:AC>IS>CK。
|
深度/cm
Depth | Shannon多样性指数H′
Shannon Diversity index H′ | 均匀度指数E′
Evenness index E′ | ||||
| CK | IS | AC | CK | IS | AC | |
| CK: 象山港口非养殖区; IS: 西泸港口交汇区; AC: 西泸港网箱养殖区 | ||||||
| 0—5 | 2.37±1.37 | 1.82±1.05 | 1.69±0.97 | 0.76±0.44 | 0.61±0.35 | 0.58±0.33 |
| 5—10 | 1.97±1.13 | 1.79±1.03 | 1.72±0.99 | 0.68±0.39 | 0.62±0.36 | 0.57±0.33 |
| 10—15 | 2.11±1.22 | 1.74±1.01 | 1.57±0.91 | 0.70±0.40 | 0.60±0.34 | 0.56±0.32 |
| 15—20 | 1.98±1.14 | 1.64±0.94 | 1.29±0.74 | 0.65±0.38 | 0.58±0.33 | 0.54±0.31 |
| 20—25 | 2.04±1.18 | 1.58±0.91 | 1.34±0.77 | 0.67±0.39 | 0.56±0.32 | 0.51±0.29 |
| 50—55 | 1.69±0.98 | 1.50±0.87 | 1.75±1.01 | 0.59±0.34 | 0.54±0.31 | 0.58±0.33 |
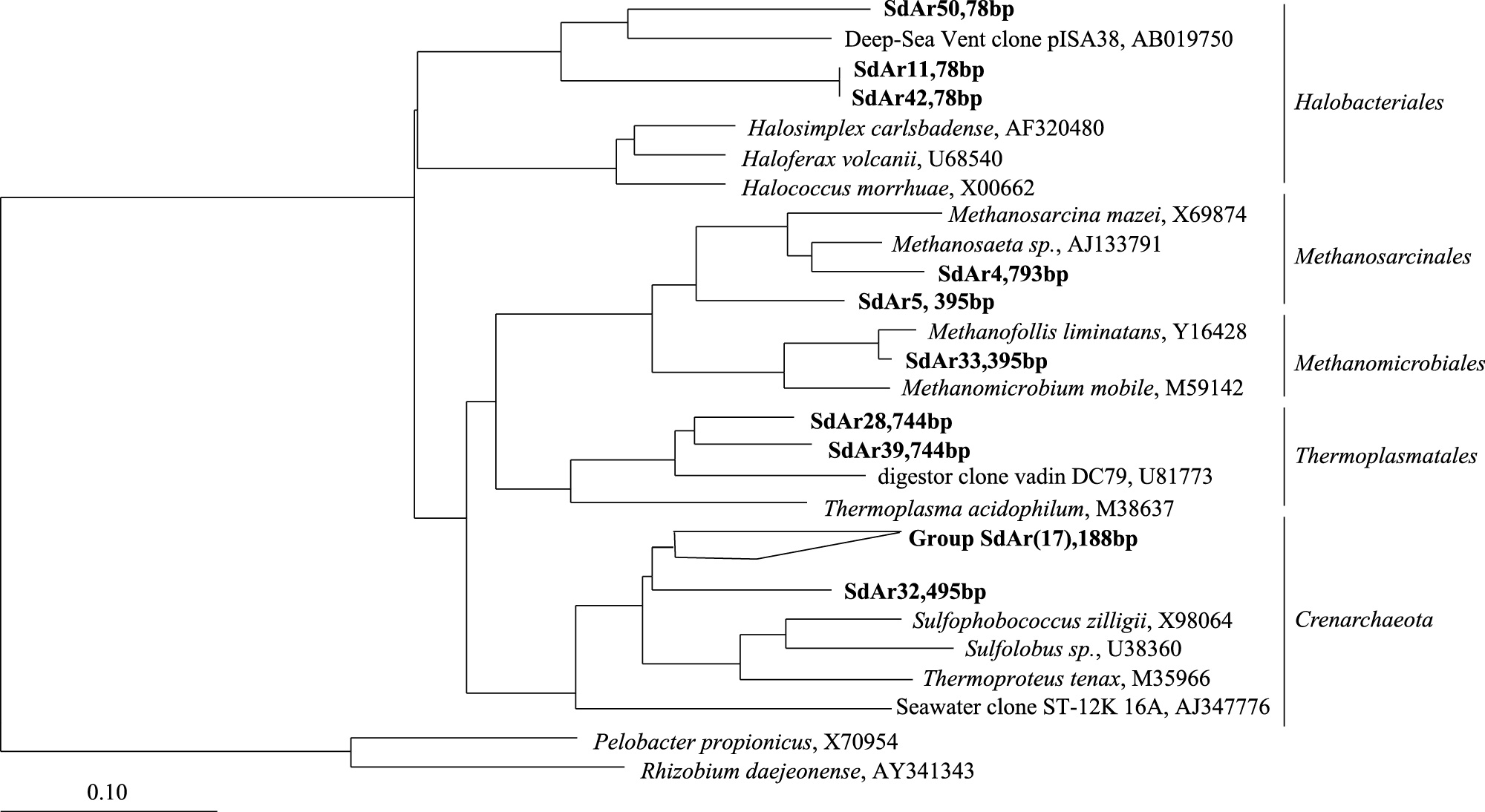
本研究测定了不同深度沉积物中各种盐离子浓度,结合温度、pH和氧化还原电位分析各位点古菌群落分布与环境因子之间的关系。RDA分析(图 4)表明,试验选取的8个环境因子对各位点古菌分布的解释量达87.7%,其中主轴RDA1的解释量为81.3%。CK区域与AC区域也相对独立,IS区域位于两者之间。铵浓度和Eh是影响CK区域古菌群落结构随深度变化的主要因素。影响AC区域的环境因子则主要是一些营养盐类和pH。除氨氮外,其它营养盐类在AC区域的含量均要高于CK区域。
|
| 图 4 不同区域、不同深度沉积物古菌群落结构与环境因子的冗余分析 Fig.4 Redundancy analysis for archaeal community compositions and environmental factors of sediments in different sites and depths 图中小写字母a—f代表深度,分别为:a(0—5 cm)、b(5—10 cm)、c(10—15 cm)、d(15—20 cm)、e(20—25 cm)和f(50—55 cm) |
古菌群落结构的研究表明,养殖活动改变了沉积物中古菌群落成分的组成比例,改变了古菌群落结构(图 2)。象山港网箱养殖活动降低了沉积物中古菌的多样性(表 1),但是未改变其优势菌群,反而因其它古菌类群的减少而增加了优势菌的相对丰度。养殖区沉积物中泉古菌在古菌群落中所占比例平均达80.3%,明显高于非养殖区(61.2%)。泉古菌作为沉积物中的优势菌群,占古菌群落的50%以上(图 1),这与其他海洋环境的古菌研究相类似。张等[29]在福建漳江口红树林沉积物古菌类群的研究中发现,泉古菌在古菌群落中所占比例达66.2%。非养殖区域古菌组成较复杂,除泉古菌外的其它菌群也占有相对较高的丰度(图 1)。T-RF 78 bp的克隆主要为嗜盐杆菌目(Halobacteriales)(图 3),在CK区域,尤其是上层(0—20 cm)沉积物中,具有较高的相对丰度。这可能与CK区域较AC和IS区域更靠近东海有关,受地表径流等影响较小,盐度更高。产甲烷古菌在本研究的克隆文库中共只有2个克隆(2/48),分别属于H2/CO2代谢型的甲烷微菌目(Methanomicrobiales)和兼有多种代谢型的甲烷八叠球菌目(Methanosarcinales)。但是,在缺氧的深海环境中,一般会有大量产甲烷古菌的富集。本研究位点沉积物的氧化还原电位显示,采集的沉积物并未完全厌氧呈还原态[1],可能不是严格厌氧的产甲烷古菌最理想的居所。
IS区域地处养殖区与港口非养殖区之间,是养殖水与东海水相交汇的区域。受到潮汐等动力的影响,养殖区的部分营养元素随着海水流动而影响到交汇区的沉积物。因此,IS区域的各环境参数居中,古菌群落结构也介于两者之间。3个研究区域中,养殖区域沉积物的营养盐浓度较高。与非养殖区相比,网箱养殖区沉积物中有机质含量显著增高[30]。有机质也是影响氮、磷含量和分布的重要因素之一,与氮、磷呈显著的正相关关系[31]。王文强等研究了哑铃湾网箱养殖对表层沉积物的影响,结果表明,养殖区碳、氮、磷和硫化物平均含量分别比非养殖区高1.23—2.12、1.33—2.79、3.71—9.99、3.02—11.84倍,且养殖年限越长含量越高,污染越严重[32]。有研究发现,养殖区底层海水中的磷主要以溶解态磷(DTP)形态存在,网箱养殖使底层海水中各形态磷都有不同程度的增加,其中DIP和DTP最为明显,养殖年限越长,含量越高,停养后也仍对水体产生一定影响;而对养殖区外水体中溶解态磷含量的影响范围则非常有限[33]。这些与养殖区域海水交换较少,而平时投入的饵料等营养物质过多有关,使得大部分未被利用的饵料以及养殖动物的排泄物等在养殖区域及附近沉积下来。也因为营养盐类浓度的增加,使养殖区的古菌群落逐渐改变,不同于非养殖区(图 4)。
综上所述,本文通过T-RFLP技术与克隆测序的方法,通过PCA和RDA分析了古菌群落分布及其与环境因子之间的关系。发现网箱养殖区沉积物的古菌群落结构较非养殖区更加简单,多样性降低。非养殖区古菌群落随深度呈现有规律的变化,受到氧化还原电位和铵浓度的影响。营养盐类和pH是造成养殖区域古菌群落结构区别于非养殖区域的主要环境因素。
| [1] | Qiu Q F, Zhang D M, Ye X S, Zheng Z Z. The bacterial community of coastal sediments influenced by cage culture in Xiangshan Bay, Zhejiang, China. Acta Ecologica Sinca, 2013, 33(2):483-491. |
| [2] | Cai H W, Sun Y L, Zhang X Q. Environmental impact of cage agriculture and the agriculture environmental capability in Xiangshan Harbor. Techniques and Equipment for Environment Pollution Control, 2006, 7(11): 71-76. |
| [3] | Zheng Y L, Zhu H W, Luo Y H. Assessment on the situation of water quality at Xiangshan Port. Marine Environmental Science, 2000, 19(1): 6-59. |
| [4] | Yin W H, Cao Z M, Lan D Z, Zhai B, Wang S S. Distribution characteristics and controlling factors of nutrients in surface sediments in xiangshan bay. Environmental Chemistry, 2007, 26(3): 388-391. |
| [5] | Lv H Q, Chang K M, Shi G D. Circulation and distribution of nitrogen and phosphorus in Xiangshan Bay, China. Oceanologia Et Limnologia Sinica, 2009, 40(2): 138-145. |
| [6] | Zhang L X, Jiang X S, Cai Y H, Li Z E. Comprehensive assessment on the situation of water quality at the red tide-monitoring area of Xiangshan harbor in recent four years. Transactions of Oceanology and Limnology, 2007, (4): 98-103. |
| [7] | Liu L, Ren M, Chen D Q, Xiang Y T. Situation of benthos in seaarea near Wusha Mountain Power Station in Xiangshan Bay. Marine Environmental Science, 2008, 27(A01): 19-22. |
| [8] | Liu J J, Zeng J N, Chen Q Z, Jian Z B, Zhao Y Q, Liao Y B, Shou L, Xu X Q, Huang Y J, Du P. The bacterial ecological distribution in water and sediment of cage culture area in Xiangshanggang Bay. Acta Ecologica Sinca, 2010, 30(2): 377-388. |
| [9] | Wang H L, Yang J F. Spatiotemporal distribution of sulfate-reducing bacteria in Xiangshan Bay and related affecting factors. Chinese Journal of Ecology, 2011, 30(12): 2857-2862. |
| [10] | Bore S I, Hedtkamp S I C, van Beusekom J E E, Fuhrman J A, Boetius A, Ramette A. Time and sediment depth-related variations in bacterial diversity and community structure in subtidal sands. The ISME journal, 2009, 3: 780-791. |
| [11] | Ren L C, Li M Y, Bao S X. The investigation of the marine archaea diversity. Life Science Research, 2006, 10(2): 67-70. |
| [12] | Hurtgen M T. The Marine Sulfur Cycle, Revisited. Science, 2012, 337: 305-306. |
| [13] | Lazar C S, Parkes R J, Cragg B A, Haridon S, and Toffin,L. Methanogenic diversity and activity in hypersaline sediments of the centre of the Napoli mud volcano, Eastern Mediterranean Sea. Environmental Microbiology, 2011, 13: 2078-2091. |
| [14] | Pitcher A, Villanueva L, Hopmans E C, Schouten S, Reichart G J, and Damste J S S. Niche segregation of ammonia-oxidizing archaea and anammox bacteria in the Arabian Sea oxygen minimum zone. The ISME Journal, 2011, 5: 1896-1904. |
| [15] | Valentine D L. Emerging topics in marine methane biogeochemistry. Annual Review of Marine Science, 2011, 3: 147-171. |
| [16] | Schleper C, Jurgens G, Jonuscheit M. Genomic studies of uncultivated archaea. Nature Reviews Microbiology, 2005, 3: 479-488. |
| [17] | Lipp J S, Morono Y K, Inagaki F, Hinrichs K U. Significant contribution of Archaea to extant biomass in marine subsurface sediments. Nature, 2008, 454: 991-995. |
| [18] | Amann R I, Ludwig W, Schleifer K H. Phylogenetic identification and in situ detection of individual microbial cells without cultivation. Microbiology and Molecular Biology Reviews, 1995, 59: 143-169. |
| [19] | Gattinger A, Guenthner A, Schloter M, Munch J C. Characterization of Archaea in soils by polar lipid analysis. Acta Biotechnol, 2002, 23: 133-139 |
| [20] | Tringe S G, Huhenholtz P. A renaissance for the pioneering 16S rRNA gene. Current Opinion in Microbiology, 2008, 11(5): 442-446. |
| [21] | Zhang X L. Cui Y Q, Lv H Y, Zhou H. Application of phylogenetic analysis in ancient DNA research. Journal of Jilin University, 2005, 43(5):696-701. |
| [22] | Marsh M E. Coccolith crystals of Pleurochrysis carterae:crystallographic faces, organization, and development. Protoplasma, 1999, 207: 54-66. |
| [23] | Diao H X. A study of the determination of ammonia in sea water with automatic analyzer oxidation process of sodium hypobromite. Marine Science, 1983, 1: 24-28. |
| [24] | Ma R K, Yu Z H, Yu Y, Zhao L J. Rapid determination of nitrate and nitrite nitrogen in drinking water by dual-wavelength method. Chinese Joural of Spectroscopy laboratory, 2010, 27(3): 822-824. |
| [25] | Banning N, Brock F, Fry J C, Parkes R J, Hornibrook E R, and Weightman A J. Investigation of the methanogen population structure and activity in a brackish lake sediment. Environmental Microbiology, 2005, 7(7): 947-960. |
| [26] | Noll M, Matthies D, Frenzel P, Derakshani M, and Liesack W. Succession of bacterial community structure and diversity in a paddy soil oxygen gradient. Environmental Microbiology, 2005, 7: 382-395. |
| [27] | Dixon P. VEGAN, a package of R functions for community ecology. Journal of Vegetation Science, 2009, 14: 927-930. |
| [28] | Schutte,U M, Abdo Z, Bent S J, Shyu C, Williams C J, Pierson J D, and Forney L J. Advances in the use of terminal restriction fragment length polymorphism (T-RFLP) analysis of 16S rRNA genes to characterize microbial communities. Applied Microbiology and Biotechnology, 2008, 80: 365-380. |
| [29] | Zhang X Y, Xu J, Xiao J, Xu J. Archaeal lineages detected in mangrove sediment in Zhangjiang estuary Fujian province. Journal of Shandong University, 2009, 44(3): 1-6. |
| [30] | Tanner J E, Fernandes M. Environmental effects of yellowtail kingfish aquaculture in South Australia. Aquacult Environ Interact, 2010, 1:155-165. |
| [31] | Huang C J, Dong Q X, Lin J D, Shang X, Li J. Sediment water, organic matter, nitrogen and phosphorus contents in a large-scale mariculture loc ated in Zhelin Bay, eastern Guangdong Province, China. Acta Oceanologica Sinica, 2008, 30(2): 38-49. |
| [32] | Wang W Q, Wei X G, Wen Y M. Influence of cage farming pollution on superficial sediments at Yaling Bay. Journal of Tropical Oceanography, 2006, 25(1): 56-60. |
| [33] | Wei X G, Wen Y M, Wang W Q, Xu X R. Content and seasonal variation of various forms of phosphorus in bottom water in cage culture area, Yaling Bay. Marine Environmental Science, 2005, 24(4): 28-32. |
| [1] | 裘琼芬,张德民,叶仙森,郑珍珍. 象山港网箱养殖对近海沉积物细菌群落的影响. 生态学报, 2013, 33(2):483-491. |
| [2] | 蔡慧文, 孙英兰, 张学庆. 象山港网箱养殖对海域环境的影响及其养殖环境容纳的研究. 环境污染治理与设备, 2006, 7(11): 71-76. |
| [3] | 郑云龙, 朱红文, 罗益华. 象山港海域水质状况评价. 海洋科学, 2000, 19(1): 6-59. |
| [4] | 尹维翰, 曹志敏, 蓝东兆等. 象山港表层沉积物中营养元素的分布特征及影响因素. 环境化学, 2007, 26(3): 388-391. |
| [5] | 吕庆华, 常抗美, 石钢德. 象山港氮、磷营养盐环流和分布规律的研究. 海洋与湖沼, 2009, 40(2): 138-145. |
| [6] | 张丽旭, 蒋晓山,蔡燕红等. 近4年来象山港赤潮监控区水质状况综合评价. 海洋湖沼通报, 2007, (4): 98-103. |
| [7] | 刘莲, 任敏,陈丹琴等. 象山乌沙山电厂附近海域的底栖生物状况. 海洋环境科学, 2008, 27(A01): 19-22. |
| [8] | 刘晶晶, 曾江宁, 陈全震, 江志兵, 赵永强, 廖一波, 寿鹿, 徐晓群, 黄逸君, 杜萍. 象山港网箱养殖区水体和沉积物的细菌生态分布. 生态学报, 2010, 30(2): 377-388. |
| [9] | 王海丽,杨季芳.象山港海域硫酸盐还原菌的时空分布及其影响因素. 生态学杂志, 2011, 30(12): 2857-2862. |
| [11] | 任立成, 李美英, 鲍时翔. 海洋古菌多样性研究进展. 生命科学研究, 2006,10(2): 67-70. |
| [21] | 张小雷, 崔银秋, 吕慧英等. 系统发育分析在古DNA种的应用. 吉林大学学报, 2005, 43(5):696-701. |
| [23] | 刁焕祥. 自动分析仪次溴酸钠氧化法测定测定海水氨氮的研究. 海洋科学, 1983, 1: 24-28. |
| [24] | 马任坤, 徐震虹, 鱼瑛等. 双波长法快速测定饮用水中的硝酸盐氮和亚硝酸盐氮.光谱实验室, 2010,27(3): 822-824. |
| [29] | 张熙颖, 许静, 肖静等. 福建漳江口红树林沉积物中的古菌类群. 山东大学学报, 2009, 44(3): 1-6. |
| [31] | 黄长江, 董巧香, 林俊达等. 粤东大规模海水增养殖区柘林湾表层沉积物中的含水量、有机质、氮和磷.海洋学报, 2008, 30(2): 38-49. |
| [32] | 王文强, 韦献革, 温琰茂. 哑铃湾网箱养殖对表层沉积物的污染. 热带海洋学报, 2006, 25(1): 56-60. |
| [33] | 韦献革, 温琰茂, 王文强等. 哑铃湾网箱养殖区底层水中各种形态P的含量和季节变化. 海洋环境科学, 2005, 24(4): 28-32. |
 2014, Vol. 34
2014, Vol. 34