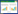文章信息
- 刘石泉, 胡治远, 赵运林
- LIU Shiquan, HU Zhiyuan, ZHAO Yunlin
- 基于DGGE技术的茯砖茶发花过程细菌群变化分析
- Analysis of bacterial flora during the fahua-fermentation process of fuzhuan brick tea production based on DGGE technology
- 生态学报, 2014, 34(11): 3007-3015
- Acta Ecologica Sinica, 2014, 34(11): 3007-3015
- http://dx.doi.org/10.5846/stxb201308052024
-
文章历史
- 收稿日期:2013-08-05
- 修订日期:2014-02-24
2. 湖南城市学院化学与环境工程学院, 益阳 413000
2. College of Chemistry and Environmental Engineering, Hunan City University, Yiyang 413000, China
茯砖茶是我国古老茶类——黑茶中的高档品种,据《明史·茶法》记载,早在明嘉靖三年(公元1524年),茯砖茶就被规定为运往西北供少数民族消费的官茶[1],可见其在历史上的重要地位。茯砖茶属于完全发酵茶,其加工工序在黑茶中最为复杂且耗时较长,包括毛茶筛分、拼配、气蒸渥堆、压制定型、发花干燥、成品包装等步骤[2],各个工艺环节的控制对保证其品质稳定至关重要。过去茯砖茶消费主要集中在边疆少数民族区域,故又被称为“边销茶”。而近年来随着相关研究深入,大量人体和动物实验资料表明茯砖茶对高血压、高血脂、肥胖、糖尿病等疾病具备良好的保健效果,并有提高机体免疫力的作用[3, 4, 5, 6, 7, 8],因而获得了“富贵病杀手”的美誉,进而受到广大国内外消费者的关注与青睐,大有超过昔日普洱茶热潮之势。
作为完全发酵茶,茯砖茶独特品质的形成与微生物作用密切相关,微生物发酵给予了茯砖茶独特的菌花香、醇厚的滋味以及在保健功效方面的提升[9, 10],之前研究者们对茯砖茶中微生物的研究,主要是围绕优势菌群——冠突散囊菌(俗称金花菌)发生、生长工艺(俗称发花工艺)进行,而对于发花过程中同时存在的其他微生物特别是细菌菌群的系统性研究较少见于报道。发花过程中细菌的存在对发花质量、茯砖茶品质有着重要的影响,要提高茯砖茶的质量必须对发花过程中细菌的动态变化加以深入研究,但对发花过程中细菌研究主要集中在平板分离计数、鉴定上[11],不能全面反映茶叶微生物群落的多样性[12],部分“存在但无法培养”的微生物不能被有效分离和鉴定,因而有待于新技术的投入与使用。分子生物学方法如免培养DGGE技术使得研究者能够在分子水平上对微生物群落多样性进行研究,并具有可靠性强、重现率高等优点,适宜对较为复杂的环境中微生物种群结构进行分析[13, 14, 15]。
本研究采用DGGE技术,对益阳茶厂有限公司(湖南产销量最大的茯砖茶生产企业)生产线上不同发花工艺期的茯砖茶样品中细菌群落的结构进行分析,以期能为茯砖茶发花工艺改进研究提供更多信息,为揭示茯砖茶这一发酵食品品质与微生物群落结构关系的深入研究提供实验支撑。
1 材料与方法 1.1 材料 1.1.1 茯砖茶样品采集在益阳茶厂有限公司茯砖茶生产线上采集同一批压制的茯砖茶作为实验样品,分别在压制后进入发花房的第0 天、2 天、4 天、6 天、8 天、10 天、12 天、14 天取样,并编号为H1、H2、H3、H4、H5、H6、H7、H8(采样时间为2013年3月28日—4月11日)。
1.1.2 主要试剂引物338F(CCT ACG GGA GGC AGC AG)、GC-338F(CGC CCG GGG CGC GCC CCG GGG CGG GGC GGG GGC GCG GGG GG CCT ACG GGA GGC AGC AG)和518R(ATT ACC GCG GCT GCT GG)由北京美亿美生物技术有限公司合成、扩增相关试剂均购自北京美亿美生物技术有限公司;聚合酶链式反应(PCR)产物纯化回收采用AXYGEN公司DNA Gel Extraction Kit试剂盒;采用OMEGA 公司Poly-Gel DNA Extraction Kit回收DGGE目的条带;其他试剂均为国产分析纯。
1.1.3 仪器与设备PTC220型PCR仪(美国 BIO-RAD公司);Gel-Doc XR凝胶成像系统(美国Bio-Rad公司);DGGE-2401变性梯度凝胶电泳仪(美国C.B.S.scientific公司)。
1.2 实验方法 1.2.1 茶叶细菌总DNA提取用Shaheen BH[16]方法提取样本细菌基因组DNA,用1%琼脂糖凝胶电泳检测提取DNA样品,置于-20 ℃冰箱内保存。
1.2.2 细菌16S rDNA片段的PCR扩增以不同样品组DNA为模板、用通用引物GC-338F和518R扩增16S rDNA 的V3高变区序列[17, 18]。
PCR扩增体系为:10× PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;GC-338F(20 mmol/L)1 μL;518R(20 mmol/L)1 μL;模板DNA 100 ng;补ddH2O至50 μL。
PCR扩增程序为:94 ℃预变性5 min;94 ℃变性30 s、55 ℃复性30 s、72 ℃延伸30 s、30个循环;最终72 ℃延伸10 min。
PCR产物采用AXYGEN公司DNA Gel Extraction Kit纯化回收。
1.3.3 PCR产物的变性梯度凝胶电泳将PCR所得产物进行DGGE分析,PCR产物取10 μL,选择变性梯度为35%—55%、浓度为8%的聚丙烯酰胺凝胶(化学变性剂为100%尿素7 mol/L和40%的去离子甲酰胺)在1×TAE缓冲液中150 V 60 ℃下电泳4 h,变性梯度凝胶电泳(DGGE)完毕后、采用银染法染色显色。
1.3.4 DGGE凝胶条带回收测序用灭菌手术刀切下待回收DGGE条带,采用OMEGA公司Poly-Gel DNA Extraction Kit回收目的条带。
以2 μL回收产物为模板,338F(CCT ACG GGA GGC AGC AG)/518R(ATT ACC GCG GCT GCT GG)为引物进行PCR扩增。PCR扩增体系(50 μL)为:10× PCR buffer 5 μL;dNTP(2.5 mmol/L)3.2 μL;rTaq(5 U/μL)0.4 μL;338F(20 mmol/L)1 μL;514R(20 mmol/L)1 μL;模板DNA 1 μL;补ddH2O至50 μL。PCR扩增程序为:94 ℃预变性4 min;94 ℃变性30 s,55 ℃复性30 s,72 ℃延伸30 s,30个循环;最后,72 ℃延伸10 min。
切胶回收重新扩增的DNA片段,纯化后连接到pEASY-T载体上,并转化至DH5α感受态细胞中,筛选阳性克隆,由北京美亿美生物技术有限责任公司对插入细菌16S rDNA片段进行序列测定。
1.3.5 序列片段分析将序列提交到GenBank数据库。在GenBank中使用Blast程序进行同源性比较,获得最相似典型菌株的16S rDNA序列[19]。
1.4 多样性分析利用分析软件(Quantity One 4.2.3 ,Bio-Rad) DGGE 图谱进行聚类和相似性分析。多样性分析用多样性指数H、均匀性指数E、丰度指数S,具体计算参见Eichner C A[20]和Sekiguchi H[21]。利用DNAstar 软件包中的SeqMan 程序将DNA 序列进行人工校对,并将所有校对后的序列方向统一为正向,通过在NCBI(美国国立生物技术信息中心)进行Blast 检索,根据比对结果获得序列对应的细菌种属信息。
2 结果与分析 2.1 样本基因组DNA提取经凝胶电泳检测,各组样品的细菌基因组DNA能够较高质量的提取。
2.2 细菌16SrDNA的PCR扩增以GC-338F和518R为引物扩增16S rDNA序列、经2%的琼脂糖凝胶电泳检测,获得约200 bp的DNA片段预期DNA片段(图 1),用于DGGE分析。
|
| 图 1 PCR扩增的16S rDNA部分序列琼脂糖凝胶电泳图 Fig. 1 Temporal and spatial variations of Peridiniopsis sp. density in backwater area of Tongzhuang River in 2009 and 2010 |
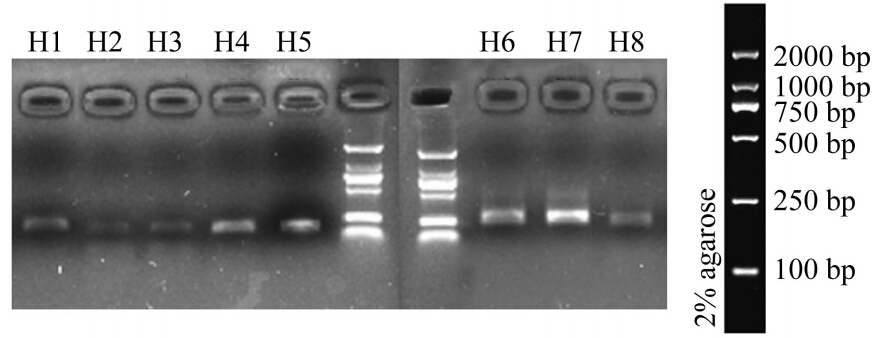
样品细菌种群的16S rDNA PCR产物的变性梯度凝胶电泳分析结果如图 2。
|
| 图 2 不同发花段细菌16S rDNA-PCR产物DGGE图谱 Fig. 2 Bacterial 16SrDNA-PCR product of DGGE map in different pile-fermentation stage |
由图 2可知,在茯砖茶的发花过程中,16S rDNA 的V3高变区序列类型较为丰富,表 1数据表明,多样性指数H在茶叶进入发花房发花的早期阶段(0—2 d)略有降低,随后则稳定升高,在发花末期(14 d)上升到最高水平2.936。均匀性指数E的变化波动较小,同多样性指数H相似呈现初期略下降而后上升的趋势。丰度指数S变化趋势则与多样性指数H基本对应,在样品H6—H8中稳定为20,说明随着茯砖茶发花的进行,茶叶内细菌群落的数量是呈上升趋势的,这和之前的研究者们对茯砖茶发花微生物的检测结果有一定差异,推测是由于在之前茶叶中微生物检测主要采用平板培养观察计数法,部分不能被培养的细菌未能进入统计所致。对于茯砖茶发花过程中微生物的观察结果,文献检索表明主要集中在散囊菌属和其他一些霉菌方面,许多研究者们认为“茯砖茶内部独特的环境及营养组成最适合冠突散囊菌生长,而于其他微生物生长不利”,加上黑毛茶本身具备一定抑菌作用[22],从而简单的推测茯砖茶发花过程中较少有细菌生长代谢的观念,实验证明细菌在发花过程中始终存在,而且发花后期细菌的群落结构趋向稳定,因而忽略细菌在茶叶中的作用,这种观念存在一定的局限性。
| 样品编号 Number of samples | 发花时间/d Fahua-fermentation stage | 多样性指数H Shannon-Wiener Index | 均匀性指数E Evenness | 丰度指数S Richness |
| H1 | 0d | 2.781 | 0.962 | 18 |
| H2 | 2d | 2.771 | 0.959 | 18 |
| H3 | 4d | 2.844 | 0.966 | 19 |
| H4 | 6d | 2.846 | 0.967 | 19 |
| H5 | 8d | 2.858 | 0.971 | 19 |
| H6 | 10d | 2.896 | 0.967 | 20 |
| H7 | 12d | 2.924 | 0.976 | 20 |
| H8 | 14d | 2.936 | 0.980 | 20 |
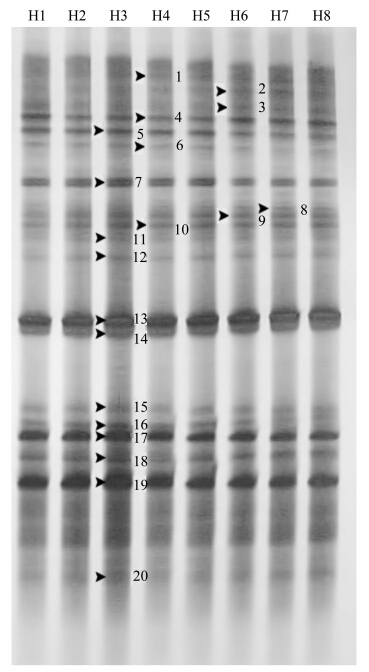
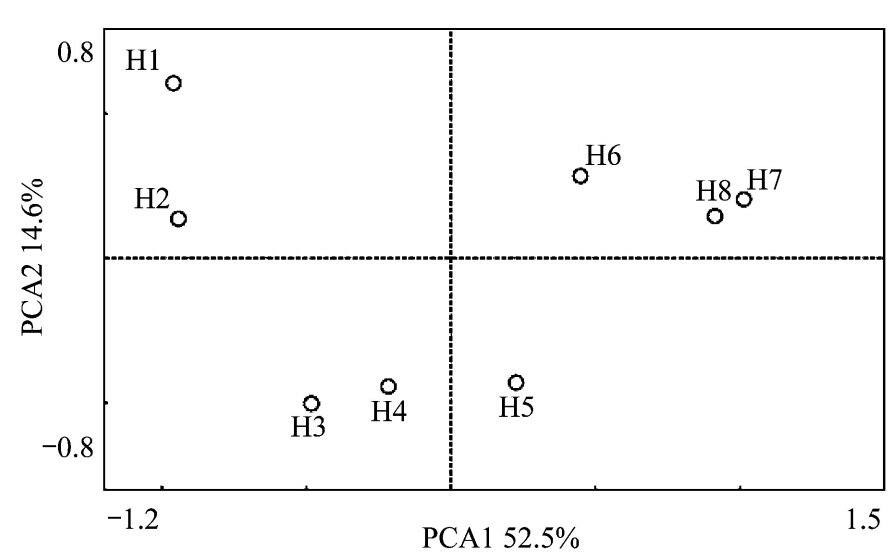
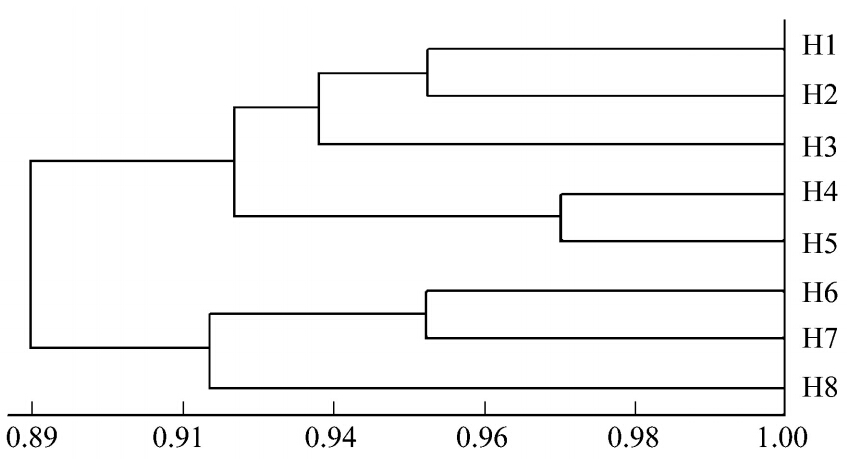
由图 3的主成分分析(PCA)可知,H1、H2分布在第二象限;H3、H4分布在第二象限;H6、H7、H8分布在第一象限,H5单独分布在第三象限,而由图 4的带型进化树也验证了这种变化,H1、H2、H3相似性较近;H4、H5相似性较近;H6、H7、H8相似性较近,这说明在14 d的发花过程中,细菌种群的构成变化较大,分别在发花的0—4 d、6—8 d、10—14 d茶叶内存在着3个差异较大的细菌优势种群结构的演变。推测是在茶叶经过高温气蒸压制后,一部分耐热的细菌以芽孢的方式存活了下来并在茶叶内生长繁殖,而从发花的第3—4天起,随着冠突散囊菌为主导的散囊菌属微生物快速生长,而冠突散囊菌的代谢产物对部分细菌具备一定抑制作用[23],这使得受到拮抗作用的菌群慢慢消失,而取代以能与冠突散囊菌共生的细菌构建优势群落。同时,随着发花过程的进行,烘房温度升高,茯砖茶内水分含量越来越低(由初期的24%—26 %到结束时的12 %以下).这些也影响了茶叶内细菌结构组成,使得细菌种群结构向更耐高温、干燥的趋势变化。
|
| 图 3 不同发花阶段主成分分析(PCA)分析 Fig. 3 Principal Component Analysis (PCA) in different Fahua-fermentation stage |
|
| 图 4 不同发花阶段进化树 Fig. 4 Evolution tree of different Fahua-fermentation stage |
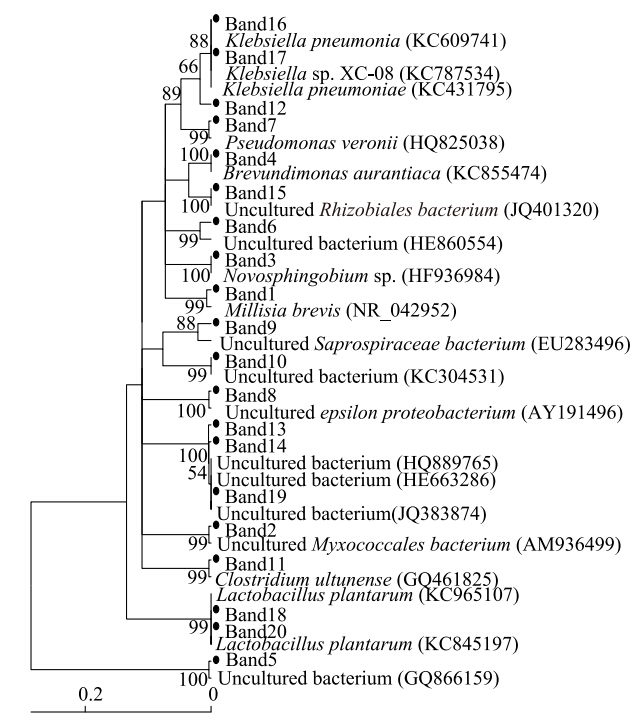
DGGE凝胶条带回收后,以338F/518R为引物进行PCR扩增,获得约200 bp的DNA片段。PCR产物纯化后连接到pEASY-T载体上,转化至DH5α感受态细胞中,筛选阳性克隆测序。测序结果递交GenBank数据库,并于GenBank中的序列进行比对,得到不同条带所代表的不同细菌类型。每个回收条带选取3个克隆进行重复检验比较,编号band1、band2、band3、…、band20表示图 2中位置1—20条带进行了序列测定,将序列提交到GenBank数据库,在GenBank中使用Blast程序进行同源性比较,获得最相似典型菌株的16S rDNA序列如表 2所示,根据这些DGGE带谱所代表的微生物即可判定茯砖茶不同发花阶段细菌群落的组成状况。
| 编号 Number | 相似菌 Similar strain | 相似度 Similarity | 登录号 Accession number | 分类 Classification |
| 16S rDNA 的V3高变区序列进行聚类分析,结果如图 5 | ||||
| Band1 | Millisia brevis(诺卡氏菌属) | 99 | NR042952 | Actinobacteria(放线菌门) |
| Band2 | Uncultured Myxococcales bacterium(不可培养粘球菌属) | 99 | AM936499 | Proteobacteria(变形菌门) |
| Band3 | Novosphingobium sp. (新鞘脂菌属) | 100 | HF936984 | Proteobacteria(变形菌门) |
| Band4 | Brevundimonas aurantiaca(短波单胞菌属) | 100 | KC855474 | Proteobacteria(变形菌门) |
| Band5 | Uncultured bacterium(不可培养细菌) | 99 | GQ866159 | - |
| Band6 | Uncultured bacterium(不可培养细菌) | 98 | HE860554 | - |
| Band7 | Pseudomonas veronii(韦龙氏假单胞菌属) | 99 | HQ825038 | Proteobacteria(变形菌门) |
| Band8 | Uncultured epsilon proteobacterium(不可培养ε-变形菌) | 99 | AY191496 | Proteobacteria(变形菌门) |
| Band9 | Uncultured Saprospiraceae bacterium(不可培养腐败螺旋菌属) | 91 | EU283496 | Bacteroidetes(拟杆菌门) |
| Band10 | Uncultured bacterium(不可培养细菌) | 99 | KC304531 | - |
| Band11 | Clostridium ultunense(突那梭菌属) | 99 | GQ461825 | Firmicutes(厚壁菌门) |
| Band12 | Klebsiella pneumonia(克雷伯氏菌属) | 99 | KC609741 | Proteobacteria(变形菌门) |
| Band13 | Uncultured bacterium(不可培养细菌) | 99 | JQ383874 | - |
| Band14 | Uncultured bacterium(不可培养细菌) | 99 | HQ889765 | - |
| Band15 | Uncultured Rhizobiales bacterium(不可培养根瘤菌属) | 100 | JQ401320 | Proteobacteria(变形菌门) |
| Band16 | Klebsiella pneumoniae(克雷伯氏菌属) | 99 | KC431795 | Proteobacteria(变形菌门) |
| Band17 | Klebsiella sp. XC-08(克雷伯氏菌属) | 100 | KC787534 | Proteobacteria(变形菌门) |
| Band18 | Lactobacillus plantarum(乳杆菌属) | 100 | KC965107 | Firmicutes(厚壁菌门) |
| Band19 | Uncultured bacterium(不可培养细菌) | 99 | HE663286 | - |
| Band20 | Lactobacillus plantarum(乳杆菌属) | 99 | KC845197 | Firmicutes(厚壁菌门) |
|
| 图 5 20个条带建立的进化树 Fig. 5 Evolution tree of 20 band |
一般情况下认为,两种细菌16S rDNA 序列同源性小于98%时属于不同种,而同源性小于93%—95%时,则属于不同的属[24],因此,从比对结果可以获知,在茯砖茶发花过程中有短波单胞菌属(Brevundimonas aurantiaca)、诺卡氏菌属(Millisia brevis)、新鞘脂菌属(Novosphingobium sp)、突那梭菌属(Clostridium ultunense)、韦龙氏假单胞菌属(Pseudomonas veronii)、乳杆菌属(Lactobacillus plantarum KC965107;Lactobacillus plantarum KC845197)、克雷伯氏菌属(Klebsiella pneumonia KC609741;Klebsiella pneumoniae KC431795;Klebsiella sp. XC-08)以及不可培养ε-变形菌(Uncultured epsilon proteobacterium)、不可培养腐败螺旋菌属(Uncultured Saprospiraceae bacterium)、不可培养粘球菌属(Uncultured Myxococcales bacterium)、不可培养根瘤菌属(Uncultured Rhizobiales bacterium)和未知分类地位的不可培养细菌Uncultured bacterium(HE860554)、Uncultured bacterium(GQ866159)、Uncultured bacterium(KC304531)、Uncultured bacterium(HQ889765)、Uncultured bacterium(HE663286)、Uncultured bacterium(JQ383874)六种。
在检测到的20种细菌当中,诺卡氏菌[25]实验证明能分泌生物活性成分,对增强机体免疫功能有着一定的作用,具有抗病原微生物感染及明显的抑肿瘤作用,新鞘脂菌[26]属等能降解呋喃丹,短波单胞菌[27]能产L-脯氨酸,植物乳杆菌[28]可产生有机酸,此外还能产生多种酶如蛋白酶、淀粉酶、脂肪酶等,显然对茯砖茶发花过程中pH值变化及茶叶风味和品质的形成起着重要作用。
发花在茯砖茶的生产工艺中又被称为“第二次发酵”,在之前茶叶原料还有着一次发酵过程即“渥堆”,渥堆之后的茶叶内成分得到微生物初步降解,茶叶纤维被软化,并且一些微生物在茶叶内开始形成优势种群,有利于之后的发花。温琼英等[29]对茯砖茶渥堆中微生物多样性进行检测,认为主要存在的细菌包括无芽孢小杆菌、芽孢杆菌、乳杆菌、金黄色葡萄球菌等,且在渥堆的初期还有大量其他细菌存在,由于当时技术条件限制未能精确鉴定到种。而方祥、赵龙飞等[30, 31]对同属黑茶的普洱茶渥堆过程中微生物的检测结果显示,乳杆菌属、芽孢杆菌属、短杆菌属、球菌属都是普洱茶发酵中的常见微生物,其中芽孢杆菌属的细菌能产生丰富的多酚氧化酶和过氧化物酶等,有利于缩短发酵时间及提高产品品质。通过与本次检测的结果对比可知,茶叶无论是在渥堆发酵还是发花过程中细菌的群落构成均较为丰富,且其中一部分渥堆细菌并未因为气蒸压制而完全失去活力,而是通过芽孢等休眠状态过渡到发花阶段继续生长繁殖。
付秀娟[32]从自然发酵的普洱茶内分离出了两株细菌B-04、B-07,并制成发酵菌液用于接种发酵普洱茶,结果显示,两种细菌单菌种发酵的普洱茶内茶褐素、茶多酚、咖啡碱均有一定程度降低,且B-07分泌的果胶酶在发酵初期时活性较高,证明细菌在茶叶发酵过程中具有一定的积极意义。
另外,本次研究中也发现茶叶中存在克雷伯氏菌、短波单胞菌、假单胞菌和粘球菌等,姚静[33]等也曾在普洱茶中分离了类似细菌,这些微生物通常被认为是条件致病菌,可感染人和动物使其患病,既然能在茯砖茶发花过程样品中却被检出,那么说明它们对于茯砖茶发花肯定有一定影响,但对茶叶品质的利弊、以及具体作用机理还有待进一步研究。
3 讨论本研究通过DGGE技术对黑毛茶发花发酵中不同时间段的细菌群落结构进行分析,发现发花过程中细菌种类较为丰富,在比对出的细菌种类中,Proteobacteria(变形菌门)9株、Firmicutes(厚壁菌门)3株、Actinobacteria(放线菌门)1株、Bacteriodetes(拟杆菌门)1株,还有六株不可培养菌株(表 2),并且群落呈现较为规律的变化,三类差异较大细菌群落分别在发花的0—4d、6—8d、10—14d之间出现,通过对比茯砖茶中优势微生物冠突散囊菌的生长曲线[34],可以发现早期的细菌群落随着冠突散囊菌的生长而受到抑制,而后期细菌与冠突散囊菌基本保持共生关系,细菌总数量随着发花的进行略有上升。其中有一部分细菌如放线菌、乳杆菌是存在于茯砖茶或普洱茶渥堆过程中的常见微生物,而另外一部分如克雷伯氏菌、短波单胞菌等在传统培养方法中都未曾检出过,因此DGGE指纹图谱在此更全面、更真实地反映了茯砖茶发花过程中细菌群落的结构和多样性变化。在利用现代分子技术解析茯砖茶发花细菌结构的基础上,进一步对细菌区系及其相互协同制约的代谢机制、以及与茯砖茶品质之间的具体的关系有望成为今后黑茶研究的重点方向。
茯砖茶因其保健功效作为当代黑茶市场的新宠,发酵技术水平是决定其品质高低的重要因素,发酵相关微生物的研究深入,能更好的人工调控发酵工艺、进一步完善产品品质。以往由于细菌在茯砖茶的发酵过程中数量相对真菌处于劣势地位,再加上检测手段的限制,而被研究者们所忽视,我们的实验证实,细菌在发花过程中大量而且始终存在,我们要在茯砖茶精控发酵中取得进一步进展,就必须重视不同细菌在发酵过程中的作用,根据细菌种类和作用的程度,以不同条件加以控制,充分发挥有益菌种的协同作用,减少或杜绝有害菌的生长,并利用有益菌种的生物转化作用改善茯砖茶品质,提高发花效率,将茯砖茶制造技术提高到一个新的水平。
| [1] | Chen Z F. Chinese Tea. Shanghai: Shanghai Culture Press, 1992: 439-441. |
| [2] | Cai Z A. Tang H P. Hunan Dark Tea. Changsha: Hunan Science and Technology Press, 2006: 12-68. |
| [3] | Wu X L, Liu Z H, Cao D, Jin L S, Lin Y. Effects of Fuzhuan Brick Tea on the adjustment of intestinal immune function in mice. Journal of Tea Science, 2013, 33(2): 125-130. |
| [4] | Fu D H, Liu Z H, Huang J A, Cai W L. Studies on Hyperlipidemia therapy compounds in Fuzhuan Tea. Journal of Tea Science, 2012, 32(3): 217-223. |
| [5] | Wang D, Huang J A, Ye X Y, Cao D, Zhang J W, Liu Z H. The Anti-obesity effects of Fuzhuan brick tea on high-fat-diet induced obesity in rats. Journal of Tea Science, 2012, 32(1): 81-86. |
| [6] | Xu X J, Huang J A, Xiao L Z, Yuan Y, Xiao W J. Effects of Fu Brick Tea and its formula on the content of TG in L-02 Steatotic Hepatocytes. Journal of Tea Science, 2011, 31(3): 247-254. |
| [7] | Xu X J, Hu Y B, Xiao W J. Effects of fermented Camilla sinensis, Fuzhuan tea, on egg cholesterol and production performance in Laying Hens. Agriculture and Food Science, 2012, (1): 6-10. |
| [8] | Manikandan R, Sundaram R, Thiagarajan R, Sivakumar M R, Meiyalagan V, Arumugam M. Effect of black tea on histological and immunohistochemical changes in pancreatic tissues of normal and Streptozotocin-induced diabetic mice (Mus musculus). Microscopy Research and Technique, 2009, 72(10): 723-726. |
| [9] | Fu D H, Liu Z H, Huang J A, Hao F, Wang F, Lei Y G. Variations of components of Fuzhuan tea during processing. Food Science, 2008, 29(2): 64-67. |
| [10] | Huang Q, Li Y P, Chen L J, Che K. Study on changes of active components in dark tea during liquid fermentation by Eurotium cristatum. Food Science, 2007, 28(12): 231-234. |
| [11] | Hu Z Y. Flora Diversity and Fermentation Process Codition of Fuzhuan Brick-tea in Hunan Area[D]. Changsha: Hunan Agricultural University, 2012. |
| [12] | Zhong W H, Cai Z C. Methods for studying soil microbial diversity. Chinese Journal of Applied Ecology, 2004, 15(5): 899-904. |
| [13] | Liu W J, Bao Q H, Jirimutu, Qing M J, Chen X, Sun T, Li M, Zhang J, Yu J, Bilige M, Sun T, Zhang H. Isolation and identification of lactic acid bacteria from Tarag in Eastern Inner Mongolia of China by 16S rRNA sequences and DGGE analysis. Microbiological Research, 2012, 167(2): 110-115. |
| [14] | Hesham Ael-L, Qi R, Yang M. Comparison of bacterial community structures in two systems of a sewage treatment plant using PCR-DGGE analysis. Journal of Environmental Sciences, 2011, 23(12): 2049-2054. |
| [15] | Tu T, Li L, Mao G N, Wang Y Y. Analysis of bacterial diversity in the Songhua river based on Nested PCR and DGGE. Acta Ecologica Sinica, 2012, 32(11): 3505-3515. |
| [16] | Humayoun S B, Bano N, Hollibaugh J T. Depth distribution of microbial diversity in Mono Lake, a meromictic soda lake in California. Applied and Environmental Microbiology, 2003, 69(2): 1030-1042. |
| [17] | Ampe F, Ben Omar N, Moizan C, Wacher C, Guyot J P. Polyphasic study of the spatial distribution of microorganisms in Mexican Pozol, a fermented maize dough, demonstrates the need for Cultivation-independent methods to investigate traditional fermentations. Applied and Environmental Microbiology, 1999, 65(12): 5464-5473. |
| [18] | Thompson J R, Marcelino L A, Polz M F. Heteroduplexes in Mixed-template amplifications: Formation, consequence and elimination by 'Reconditioning PCR'. Nucleic Acids Research, 2002, 30(9): 2083-2088. |
| [19] | Attwood T K, Parry-Smith D J, Luo J C. Fundamental Concepts of Bioinformatics. Beijing: Peking University Press, 2002: 97-113. |
| [20] | Eichner C A, Erb R W, Timmis K N, Wagner-Döbler I. Thermal gradient gel electrophoresis analysis of bioprotection from pollutant shocks in the activated sludge microbial community. Applied and Environmental Microbiology, 1999, 65(1): 102-109. |
| [21] | Sekiguchi H, Watanabe M, Nakahara T, Xu B H, Uchiyama H. Succession of bacterial community structure along the Changjiang River determined by denaturing gradient gel electrophoresis and clone library analysis. Applied and Environmental Microbiology, 2002, 68(10): 5142-5150. |
| [22] | Fu D H, Yu Z Y, Huang J A, Luo Z M. Research on the bacteriostatic effect of water extract of brick tea produced in different years. China Tea, 2011, 33(1): 10-12. |
| [23] | Li J L, Hu B H, Zhao Y B, Liu S C, Jiang Y J, Liu Z H. Anti-microbial activity of fermentation supernatants of Eurotium cristatum. Food Science, 2011, 32(11): 157-160. |
| [24] | Wang Z X, Xu Y, Zhou P J. Taxonomy o f a new species of haloalkalophilic archaron. Acta Microbiologica Sinica, 2000, 40(2):115-120. |
| [25] | Zhang Z L, Tang W L, Huang Y Z, Hong J J. Bioactivity of Nocardia Rubra cell. Microbiology, 2009, 36(3): 389-392. |
| [26] | Jing X T, Li Q F, Guo W, Kong X Y, Zhang L H. Research progress of Carbofuran-degrading bacteria. Journal of Anhui Agricultural Sciences, 2010, 38(27): 15032-15034. |
| [27] | Zhao S J, Dou W F, Li H, Zhang D D, Shi J S, Xu Z H. Breeding of brevundimonas diminuta for L-proline and optimization of its fermentation conditions. Chinese Journal of Bioprocess Engineering, 2012, 10(4): 21-25. |
| [28] | Wang S Q, Bao Y, Dong X M, Su F, Yao G Q, Zhang H P. Physiological function and application of Lactobacillus Plantarum. Journal of Agricultural Science and Technology, 2010, 12(4): 49-55. |
| [29] | Wen Q Y, Liu S C. Variation of the microorganism groups during the Pile-fermentation of Dark Green tea. Journal of Tea Science, 1991, 11(S1): 10-16. |
| [30] | Fang X, Chen D, Li J J, Zhao C Y, Li B, Huan G Z, Chen Z Z. Identification of microbial species in Pu-erh tea with different storage time. Modern Food Science and Technology, 24(2): 105-108, 160-160. |
| [31] | Zhao L F, Zhou H J. Study on the main microbes of Yunnan Pu-er tea during Pile-fermentation process. Journal of Shangqiu Teachers College, 2005, 21(2): 129-133. |
| [32] | Fu X J, Song W J, Xu Y Q, Li C W. Effects of different microorganisms on the fermentative process of Pu-er tea. Journal of Tea Science, 2012, 32(4): 325-330. |
| [33] | Yao J, Chen D, Zheng X Y, Wang Y. Isolation and molecular identification of the bacterial colonization during the pile fermentation process of Pu-erh tea. Journal of Anhui Agricultural Sciences, 2013, 41(6): 2667-2668, 2671-2671. |
| [34] | Hu Z Y, Liu S C, Zhao Y L, Li L M, Liu S Q, Li Y Z. Dynamic microbial changes of Fuzhuan Brick Tea during processing and identification of the dominant fungi. Food Science, 2012, 33(19): 244-249. |
| [1] | 陈宗樊. 中国茶经. 上海: 上海文化出版社, 1992: 439-441. |
| [2] | 蔡正安, 唐和平. 湖南黑茶. 长沙: 湖南科学技术出版社, 2006: 12-68. |
| [3] | 吴香兰, 刘仲华, 曹丹, 金莉莎, 林勇. 茯砖茶对小鼠肠道免疫功能调节作用的研究. 茶叶科学, 2013, 33(2): 125-130. |
| [4] | 傅冬和, 刘仲华, 黄建安, 蔡汶莉. 茯砖茶降脂功能成分研究. 茶叶科学, 2012, 32(3): 217-223. |
| [5] | 王蝶, 黄建安, 叶小燕, 曹丹, 张健炜, 刘仲华. 茯砖茶减肥作用研究. 茶叶科学, 2012, 32(1): 81-86. |
| [6] | 徐小江, 黄建安, 肖力争, 袁勇, 肖文军. 茯砖茶及其配方对脂肪变性L-02肝细胞中TG含量的影响. 茶叶科学, 2011, 31(3): 247-254. |
| [9] | 傅冬和, 刘仲华, 黄建安, 郝翻, 王芳, 雷玉国. 茯砖茶加工过程中主要化学成分的变化. 食品科学, 2008, 29(2): 64-67. |
| [10] | 黄群, 李彦坡, 陈林杰, 车科. 冠突散囊菌液态发酵过程中黑茶活性成分变化研究. 食品科学, 2007, 28(12): 231-234. |
| [11] | 胡治远. 湖南地区茯砖茶菌群多样性及发花工艺优化研究 [D]. 长沙: 湖南农业大学, 2012. |
| [15] | 屠腾, 李蕾, 毛冠男, 王莹莹. 利用细胞计数手段和DGGE技术分析松花江干流部分地区的细菌种群多样性. 生态学报, 2012, 32(11): 3505-3515. |
| [19] | (英)谈莲莎(Attwood T K), (英)史密斯(Parry-Smith D J)著//罗静初译. 生物信息学概论. 北京: 北京大学出版社, 2002: 97-113. |
| [22] | 傅冬和, 余智勇, 黄建安, 罗珍美. 不同年份茯砖茶水提取物的抑菌效果研究. 中国茶叶, 2011, 33(1): 10-12. |
| [23] | 李佳莲, 胡博涵, 赵勇彪, 刘素纯, 姜越君, 刘仲华. 冠突散囊菌发酵液的抑菌作用. 食品科学, 2011, 32(11): 157-160. |
| [24] | 王振雄, 徐毅, 周培瑾. 嗜盐碱古生菌新种的系统分类学研究. 微生物学报, 2000, 40(2): 115-120. |
| [25] | 张祝兰, 唐文力, 黄颖桢, 洪金基. 红色诺卡氏菌的生物活性. 微生物学通报, 2009, 36(3): 389-392. |
| [26] | 荆新堂, 李勤凡, 郭伟, 孔祥雅, 张丽慧. 呋喃丹降解菌的研究进展. 安徽农业科学, 2010, 38(27): 15032-15034. |
| [27] | 赵世杰, 窦文芳, 李会, 张旦旦, 史劲松, 许正宏. 缺陷短波单胞菌高产L-脯氨酸菌株的选育及其发酵条件优化. 生物加工过程, 2012, 10(4): 21-25. |
| [28] | 王水泉, 包艳, 董喜梅, 苏芳, 姚国强, 张和平. 植物乳杆菌的生理功能及应用. 中国农业科技导报, 2010, 12(4): 49-55. |
| [29] | 温琼英, 刘素纯. 黑茶渥堆(堆积发酵)过程中微生物种群的变化. 茶叶科学, 1991, 11(S1): 10-16. |
| [30] | 方祥, 陈栋, 李晶晶, 赵超艺, 李斌, 黄国资, 陈忠正. 普洱茶不同贮藏时期微生物种群的鉴定. 现代食品科技, 2008, 24(2): 105-108, 160-160. |
| [31] | 赵龙飞, 周红杰. 云南普洱茶渥堆过程中的主要微生物初探. 商丘师范学院学报, 2005, 21(2): 129-133. |
| [32] | 付秀娟, 宋文军, 徐咏全, 李长文. 不同种类微生物对普洱茶发酵过程的影响. 茶叶科学, 2012, 32(4): 325-330. |
| [33] | 姚静, 陈迪, 郑晓燕, 王云. 普洱茶渥堆发酵过程中细菌种群的分离与分子鉴定. 安徽农业科学, 2013, 41(6): 2667-2668, 2671-2671. |
| [34] | 胡治远, 刘素纯, 赵运林, 李罗明, 刘石泉, 李燕子. 茯砖茶生产过程中微生物动态变化及优势菌鉴定.食品科学, 2012, 33(19): 244-249. |
 2014, Vol. 34
2014, Vol. 34